A- и В- формы ДНК. Структура РНК.
Часть 1
1.Получила Файлы gatc_a.pdb, gatc_b.pdb и gatc_z.pdbЧасть 2
2.1
2.2.
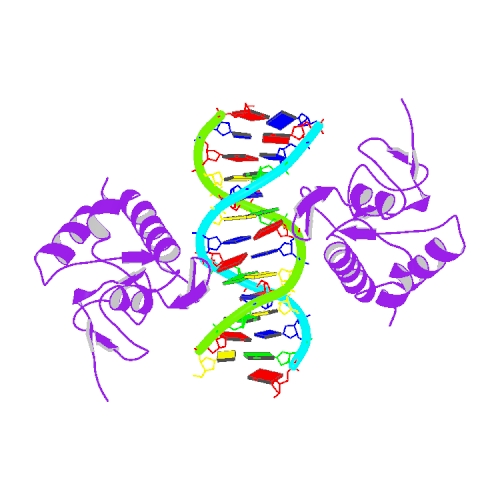 1mhd.pdb
1mhd.pdb2.3.
 Разрывов в ДНК нет
Разрывов в ДНК нетЧасть 3
3.1Выбираем Cytosine 24 (в А-, В-, Z-формах)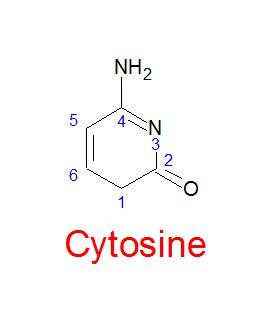
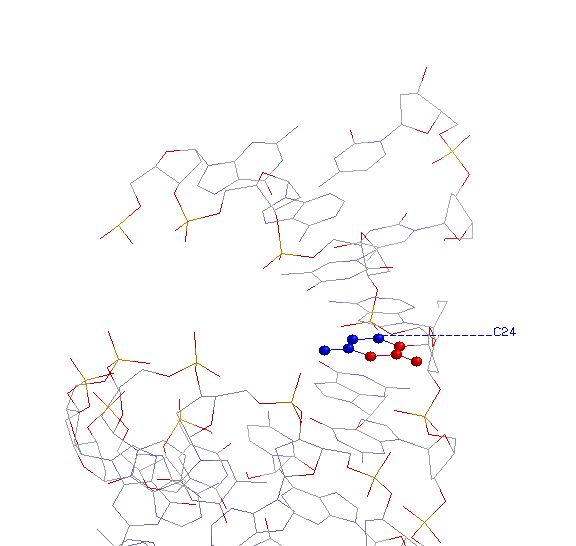
A, B- формы: С6, С5, С4, Т4 обращены в сторону большой бороздки (Красные)
N3, C2, O2, N1 обращены в сторону малой (синие)
В Z-форме наоборот
3.2
2.
A-форма |
B-форма |
Z-форма |
|
| Тип спирали | правая |
правая |
левая |
| Шаг спирали | 28.03 |
33.75 |
43.50 |
| Число оснований на виток | 11 |
10.4 |
12 |
| Ширина большой бороздки | 16.81 (A34B.P-G9A.P) |
17.21 (G29B.P-G9A.P) |
15.17 (G15B.P-C50A.P) |
Ширина малой бороздки |
7.98 (G25B.P-A10A.P) |
11.69 (T7A.P-A38B.P) |
7.20 (G53A.P-G151B.P) |
3.3 Измерение торсионных углов
| α | β | γ | δ | ε | ζ | χ | ||
| А-форма | -51.69 | 174.79 | 41.68 | 79.09 | -147.84 | -75.06 | -157.18 | |
| В-форма | -29,87 | 136,38 | 31,1 | 143.42 | -140.77 | -160.52 | -97.96 |
Несоответствие со значениями в презентации вероятно из-за того, что в данном случае углы посчитаны для Цитозина, а в презентации для Гуанина.
Краткое описание структуры в файле 1р4s.pdb
В файле приведены координаты атомов молекул из Thermus Thermophilus: белок пролин-тРНК синтетаза.Для исследования была выбрана Т цепь тРНК, имеющая следующую последовательность:
[1] 5'- CGGGGAGUAGCGCAGCCCGGUAGCGCACCUCGUUCGGGACGAGGGGGGCGCUGG 5MU PSU CAGAUCCAGUCUCCCCGACCA-3` [77]
В последовательости на 3'-конце имеется триплет ССА, к которому присоединяется аминокислота. Координаты ССА не определены, определены координаты атомов 4-69 н.к.
Исследование вторичной структуры
C помощью программы find_pair (find_pair -t rna.pdb stdout | analyze) покета 3DNA были определены возможные водородные связи между азотистыми основаниями. ФайлВ соответствие с полученными данными, акцепторные стебель состоит из участка 4-7 и комплементарного ему участка 66-69
Т-стебель: 49-53 и 61-65
D-стебель: 10-13 и 22-25
Антикодоновый стебель: 38-44 и 26-32
| Рис.1. Вторичная структура аргининовой тРНК из Thermus Thermophilus. | Скрипт для получения изображения |
 |
restrict rna
backbone 100 background white select 4-7,66-69 color green select 10-13,22-25 pause color blue select 49-53,61-65 pause color gray select 38-44,26-32 color black |
Неканоническая пара:

Присутствует вариабельная пется: 45-48.
В Т-петле отсутствует тимидин. Присутствует псевдоуридин-5'-монофосфат и 5-метилуридин-5'-монофосфат.
В D-петле отсутствует дигидроуридин.
Исследование третичной структуры.
Между основаниями конца акцепторного стебля и начала Т-стебля возможно стекинг-взаимодействие: 7,66-49,65 составляет 2.69; 10,25-44,29 составляет -0.8.Из этого следует, что стекинг-взаимодействие между данными основаниями маловероятно.
Наложенеи оснований


Рассмоотрим дополнительные стабилизирующие водородные связи (не вошедшие в стебли):
(0.012) T:..54_:[5MU]u-**-xG[..G]:..58_:T (0.006) - внутри Т-петли
(0.026) T:..55_:[PSU]Px**+xG[..G]:..18_:T (0.012) - между Т и D петлями
(0.003) T:..14_:[..A]A-**-xU[..U]:...8_:T (0.004) - между D-петлей и нуклеотидом между акцепторным и D-стеблем
(0.004) T:..15_:[..G]Gx**+xC[..C]:..48_:T (0.007) - между D-петлей и вариабельной петлей
(0.011) T:..19_:[..G]G-----C[..C]:..56_:T (0.005) - между D и Т петлями
Следовательно, стабилизация идет в основном за счет неканонических взаимодействий, при этом D-пется участвует чаще (вероятно из-за её большей длины).
Предсказание вторичной структуры тРНК
Используется программа einverted пакета EMBOSS| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'4-7 3' 5'66-69 3' Всего 4 пары |
предсказано 4 из 7 | Предасказано 7 из 7 |
| D-стебель | 5'10-13 3' 5'22-25 3' Всего 4 пары |
Ни одна пара не предсказана | Предсказано 4 пары |
| T-стебель | 5'49-53 3' 5'61-65 3' Всего 5 пар |
Ни одна пара не предсказана | предсказано 5 |
| Антикодоновый стебель | 5'38-44 3' 5'26-32 3' Всего 7 пар |
предсказано 6 из 7 пар | 7 пар |
| Общее число канонических пар нуклеотидов | 15 | 10 | 15 |
Лучшее предсказание вторичной структуры тРНК по алгоритму Зукера
Использовался параметр P=15. (Чем больше значение этого параметра, тем больше вариантов предсказания структуры)