1. Определение таксонов
При помощи таксономического сервися NCBI я определила, к каким таксонам относятся отобранные мной в прошлос задании бактерии. Это отображено в таблице ниже:| Название | Мнемоника | Таксономия |
| Bacillus anthracis | BACAN | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Enterococcus faecalis | ENTFA | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Geobacillus kaustophilus | GEOKA | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus delbrueckii | LACDA | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | LACLM | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | LISMO | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus epidermidis | STAES | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
2. Из данного списка функций белков я выбрала одну: EFG (Elongation factor G).
Далее из банка Swiss-Prot получаем последовательности белком с данным фактором элонгации:
seqret sw:efg_lacdaЗатем при помощи кманты cat объединяем все полученные последовательности в один файл:
cat efg_bacan.fasta efg_entfa.fasta efg_geoka.fasta efg_lacda.fasta efg_laclm.fasta efg_lismo.fasta efg_staes.fasta > all_bacteria.fastaПри помои программы muscle выравниваем полученные последовательности:
muscle -in all_bacteria.fasta -out alignment_all_bacteria.fastaПолученное выравнивание открываем в программе JalView и представляем в "блочной" форме с блоками шириной в 100 остатков и раскраской по проценту идентичности:
проект JalView и fasta последовательность.
3. Реконструкция филогенетического дерева
Далее я реконструировала филогенетическое дерево 4мя методами, доступными из JalView.1. Average Distance Using % Identity
| Полученное дерево | Правильное дерево |
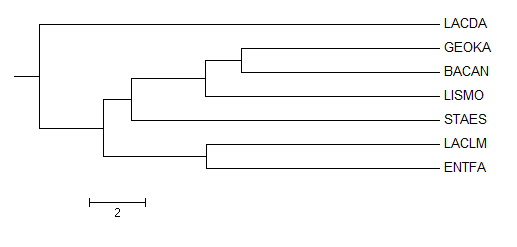 | 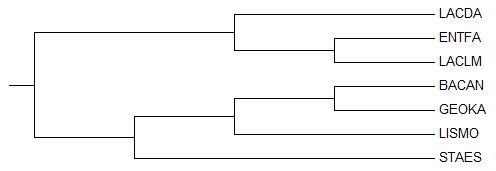 |
(Bacan, GEOKA, LISMO, STAES)vs(LACDA, ENTFA, LACLM) у правильного дерева против
(BACAN, GEOKA, LISMO) VS (LACDA LACLM, ENTFA) у полученного дерева
2. Neighbour Joining Using % Identity
| Полученное дерево | Правильное дерево |
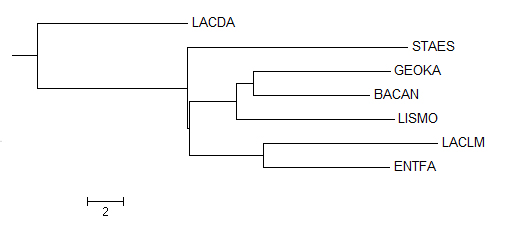 |  |
(GEOKA, BACAN, LISMO, STAES) vs (LACDA,ENTFA, LACLM,) у правильного дерева против
(GEOKA, BACAN, LISMO) vs (LACDA, STAES, LACLM, ENTFA) у полученноо дерева
3. Average Distance Using BLOSUM62
| Полученное дерево | Правильное дерево |
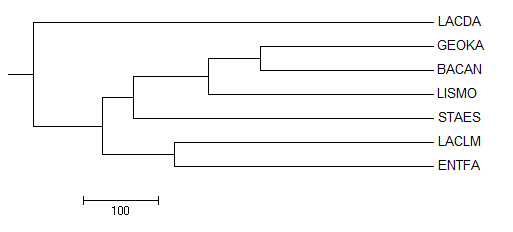 |  |
4. Neighbour Joining Using BLOSUM62
| Полученное дерево | Правильное дерево |
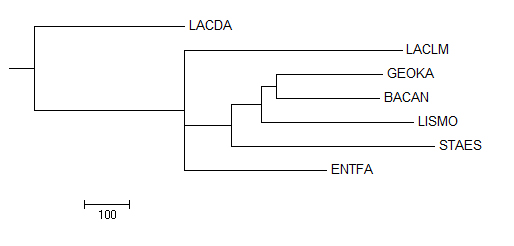 |  |
4. Реконструкция филогенетического дерева методом Maximum Parsiоny
| Полученное дерево | Правильное дерево |
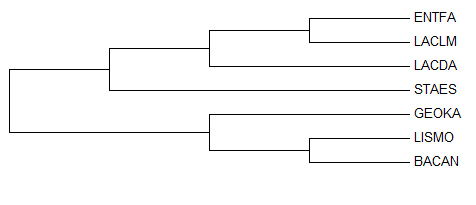 |  |
{ENTFA, LACLM, LACDA, STAES}vs{GEOKA, LISMO, BACAN} против у правильного дерева:
{LACDA, ENTFA, LACLM}vs{BACAN, GEOKA, LISMO, STAES}
(BACAN, GEOKA) VS (LISMO, STAES, LACDA, ENTFA, LACLM) у правильного дерева против (BACAN, LISMO) vs (GEOKA, STAES, LACDA, LACLM, ENTFA)