1. Описание трансмембранных белков с известной 3D структурой
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков, которые я самостоятельно отобрала, я определила следующие параметры| PDB код | Тип | Тип мембранны | Толщина гидрофобной части мембраны | Медиана числа остатков в одном трансмембранном участке |
| 2ksd | Спираль | Внутренняя мембрана Escherichia coli | 27.9 ± 2.8 A | 20,5 |
| 3rce | Спираль | Внутренняя мембрана Campylobacter lari | 28.8 ± 0.9 A | 18 |
| 3rhw | Спираль | Плазматическая мембрана Caenorhabditis elegans | 30.2 ± 0.7 A | 20 |
| 3qra | Баррель | Внешняя мембрана Yersinia pestis | 25.2 ± 1.4 A | 7,5 |
| 3dzm | Баррель | Внешняя мембрана Thermus thermophilus | 28.5 ± 2.6 A | 10 |
| 2fgq | Баррель | Внешняя мембрана Comamonas acidovorans | 25.0 ± 0.9 A | 8 |
2. Отбор гомологов
Используя PSI-BLAST был проведен поиск гомологов моего белка 1YCE (цепь А). Так как этот белок из прокариотического организма (Ilyobacter tartaricus), то изначально я исключила филум Fusobacteria, посмотрев таксономию. Проведя 4 итерации PSI-BLAST с e-value 1e-5 и максимальным количеством хитов, увеличенным до 500 (и 1000 в двух случаях), я получила 10 гомологов моего белка.3. Анализ структуры выданного белка
В базе данных OPM я нашла выданный мне белок 1YCE. В баз данных TCDB он найден не был. Однако, если пройти по ссылке на OPM, то она приводит к статье на сайте TCDB про семейство АТФ-аз.Используя полученную информацию, я заполнила следующую таблицу:
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей (бета-тяжей) к нормали | Количество трансмембранных спиралей (бета-тяжей в бочонке) | Название белка |
| 1YCE | Ilyobacter tartaricus | Внутренняя мембрана | 3.A.2.* | 0 ± 0° | 22 (11 субъединиц) | F-type Sodium ATP-ase |
4. Анализ множественного выравнивания трансмембранных белков
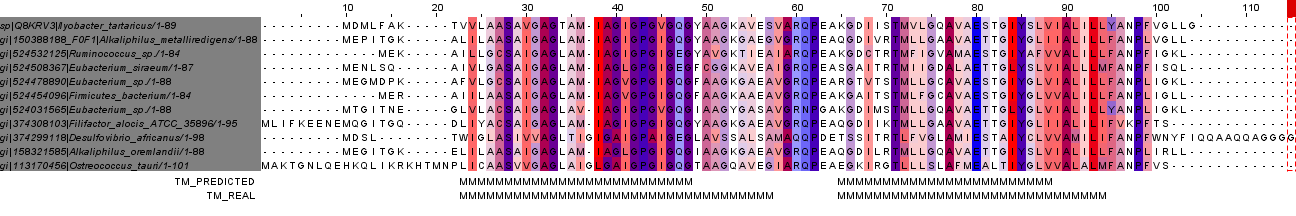
При помощи программы muscle я построила множественное выравнивание, которое затем загрузила в программу JalView. Исходный белок (у которого есть структура) находится в верхней строчке. Была добавлена строка аннотации TM_REAL, и на ней буквой М были помечены участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой.Затем была добавлена вторая строка аннотации - TM_PREDICTED, основанная на предсказании трансмембранных спиралей гомолога моего белка, выданном программой TMHMM.
Была выбрана цветовая схема Hydrophobicity, позволяющая различать гидрофобные и гидрофильные остатки. Также окрашивание происходило по консервативности (46%). Гидрофобные остатки выделены красным цветом. Как можно видеть, трансмембранные участки довольно консервативны, а нетрансмембранные участки неконсервативны. Трансмембранные участки, предсказанные программой TMHMM, более менее хорошо совпадают с реальными
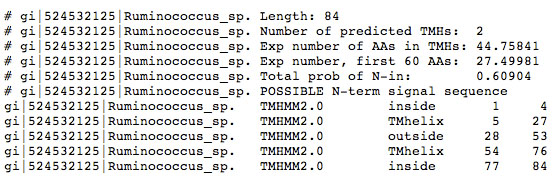 |
 |