1. Укоренение в среднюю точку
Я выбрала дерево, полученное в предыдущем задании (Neighbour Joining Using % Identity), укоренила его в среднюю точку при помощи программы retree пакета PHYLIP:1. Скопировала файл с деревом в Newick-формате в файл с именем intree;
2. Запустила retree.exe с помощью Far manager;
3. Вызвала help, чтобы разобраться в программе, нажав знак "?";
4. Для укоренения в среднюю точку нажала "M" ("Midpoint root the tree");
5. Для записи укорененного дерева в файл нажала "W"
Далее полученный файл можно открыть в программе MEGA, предварительно дав ему формат .tre
| Укорененное в среднюю точку дерево | Неукорененное в среднюю точку дерево |
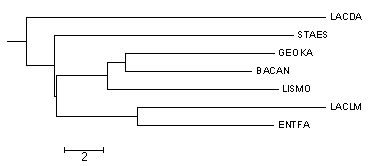
|
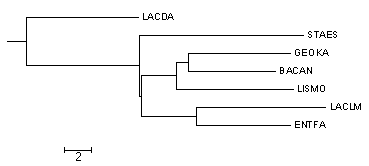
|
| Начальное дерево |
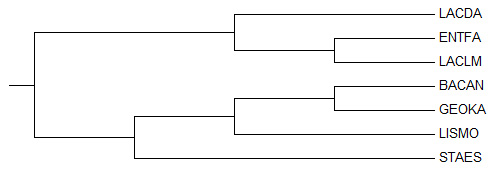
|
Укоренение произошло в правильное место. Также, дерево было выровнено, так как из-за данного метода ветки встают в произвольном порядке.
Укоренять деревья, построенные методом UPGMA, в среднюю точку не имеет смысла, так как все они укорененные и неметрические (с топологией, но без длин ветвей).
2.Использование внешней группы
Деревя, построенные методом "Maximum parsimony" (максимальной экономи) невозможно укоренить в среднюю точку, потому что этот метод определяет дерево таким образом, чтобы минимизировать количество мутаций. Он не реконструирует длины ветвей, поэтому немозможно посчитать самый длинный путь от листа к листу и найти среднюю точку.Однако существует способ обойти эту проблему - воспользоваться укоренением с помощью внешней группы. Это можно сделать, используя в качестве внешней группы белок того же семейста из другой бактерии (E. coli в нашем случае).
Добавляем к файлу с невыровненными последовательностями белков фирмикут последовательность из кишечной палоча, после чего выравниваем вместе.
muscle -in all_bact_ecoli.fasta -out aligment_all_bact_ecoli.fastaНе забыв отредактировать имена (то есть оставив только мнемонику видов), я импортировала полученный файл в программу MEGA и построила дерево "Maximum parsimony".
Далее выполнила следующие действия:
Subtree => Root => Show Subtree Separetely
| Maximum parsimony | Изначальное дерево |
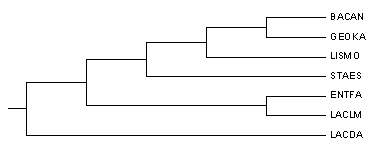
|

|
Метод максимальной экономии, в отличие от других, не оценивает длины ветвей. Получанное дерево практически не отличается от изначального.
Однако, если в изначальном дереве первое ветвление разделяет дерево на:
{LACDA; ENTFA; LACLM} vs {BACAN; GEOKA; LISMO; STAES};
то в полученном дереве ветвь LACDA выделилась.
3.Бутстрэп
В данном задании необходимно провести бутстрэп-анализ филогении выбранных мною ранее белков, используя один из методов, доступных из программы MEGA. Для этого в окне, которое открывается после вызова программы (UPGMA дерево), в меню "Test Phylogeny" выбираем "Bootstrap method" и указываем число реплик (100).| Original tree | Bootstrap consensus tree |
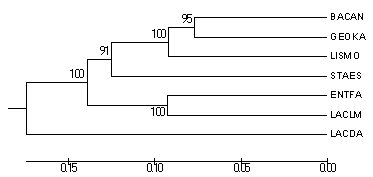
|
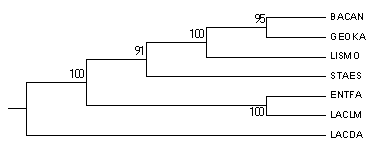
|
| Начальное дерево |

|
Различий между Original tree и Bootstrap consensus tree нет. Однако, они не совпадают с начальным деревом.
По идее, построение по части данных (число реплик, равное 100) такое же по достоверности, как и по полноценным данным.