Умения - D2
D2: совмещение структур
Для совмещения при помощи сервиса PDBeFold было выбрано 5 структурных гомологов моего белка 1RLJ: 4N82, 3F90, 3KAP, 4HEQ, 3ESY. Визуализавия в Jmol (рисунок 1) показал, что структуры более-менее похожи.
Рис. 1. Совмещение выбранных структурных гомологов 1RLJ, JMol, сервис PDBeFold.
Выравнивания по структуре и последовательности не идентичны, что можно увидеть на рисунке 2. Например на данном усастке (39-54 а.о.), во множественном выравнивани последовательностей есть невыровненные остатаки белков, плюс гэпы, из-за которых остатки не выравниваются так, как в структуре.
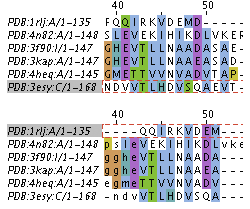
Рис. 2. Выравнивания, размеченные по Crustalx, по структуре (сверху) и последовательности, полученное методом Muscle (снизу).
Вероятно, выравние по структуре более правильное.
Совмещение по заданному выравниванию
Для работы была выбрала структура loga. Цепь альфа - 118-202; цепь бета - 119 - 245. Далее при помощи сервиса Sheep были получены карты бета-листов (Рисунки 3,4)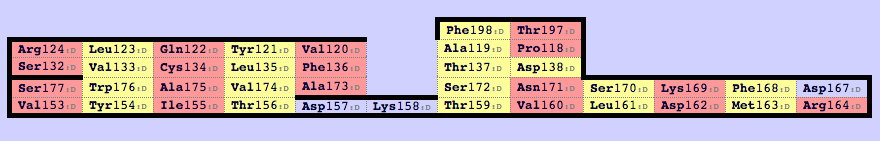
Рис. 3. Карта бета-листа для домена из цепи альфа.
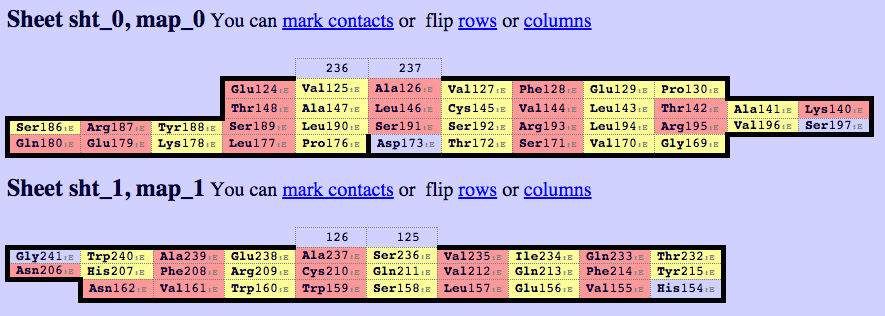
Рис. 4. Карта бета-листа для домена из цепи бета.
На рисунке 5 карта бета листа (цепь бета) соответсвует ориентации карты цепи альфа. Выровненны консервативные цистеины.
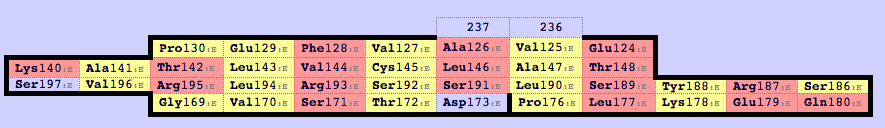
Рис. 5. Карта бета-листа для домена из цепи бета (map0), соответствуящая ориентация.
Затем необходимо было составить команду для pymol, которая совмещает структуры по полученному выравниванию.
pair_fit alpha_sel///121-123+133-135+174-176/CA, beta_sel///126-128+144-146+191-193/CARMSD = 0.485

Рис. 6. Совмещение доменов T-клеточного рецептора из цепи альфа (зеленый цвет) и цепи бета (голубой цвет).
Файл с совмещением. Ход полипептидной цепи в пространстве совпадает, то есть топологии сходны.