Работа с Pymol
1. Оцените возможности Sculpting, в Wizard->Demo->Sculpting
Интересная функция pymol на "поиграть". Дается какой-то белок, который можно видоизменять двумя способами. Мило.
2. Мутация в белке, которая должна привести к потере связывания с лигандом
Для начала, я оценила зону контакта. На рисунке 1 атомы на расстоянии менее 5 Å показаны в модели sticks, лиганд окрашен красным. Остальной белок в виде cartoon. При помощи команды distance (порог 3.5) получила изображение возможных водородных связей между белком и лигандом.
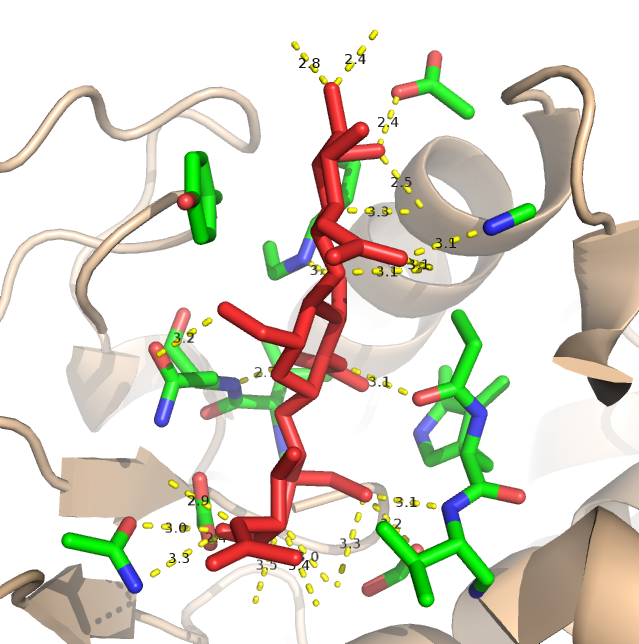
Рис. 1. Белок 1LMP, контакт с лигандом.
Далее нужно провести мутацию такую, чтобы связь с лигандом пропала. Я решила заменить ASP'101 на LEU, выбрав ротамер, который больше мешает лиганду. Действительно, мутация привела к потери связи.
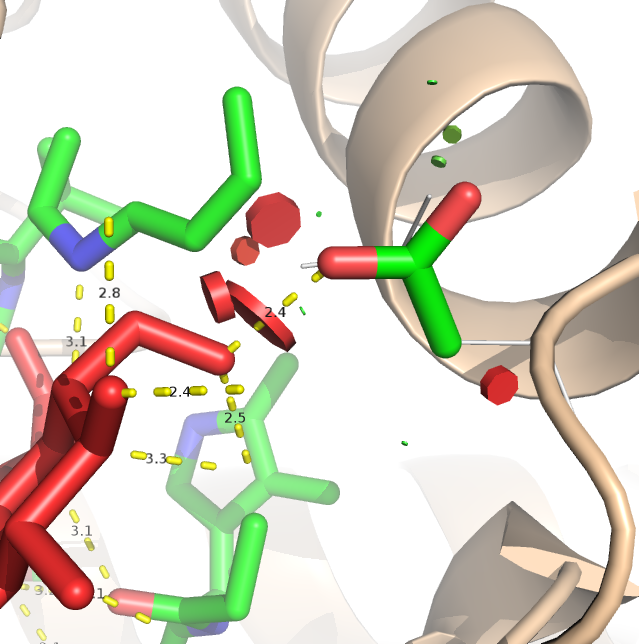
Рис. 2. Выбор ротамера при мутации ASP'101 на LEU
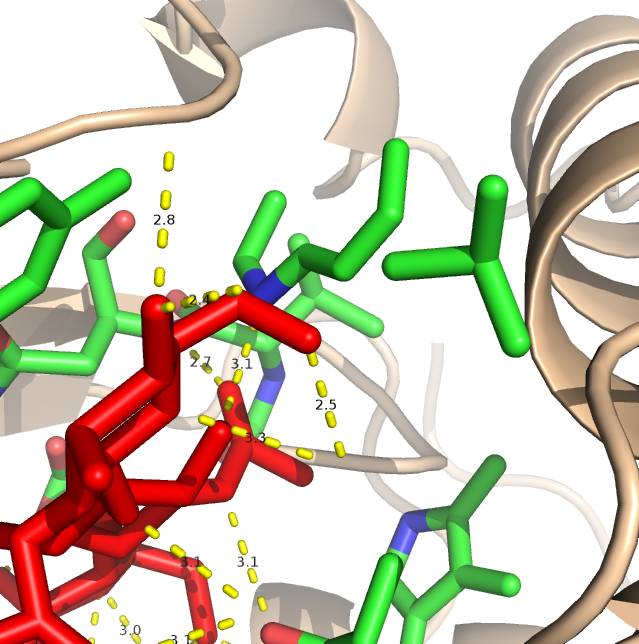
Рис. 3. Отсутствие связи с лигандом после мутации ASP'101 на LEU'101.
3. Создание анимационного ролика
Фиолетовым цветом изображен 1LMP, охрой - мутантный белок. Лиганд окрашен в красный.
4. Присоединение флуоресцентной метки
Для присоединения флуоресцентной метки TAMRA я, сначала, изменила формат скачанного файла в pdb, а затем при помощи команд fuse и torsion получила следующее изображение (рис. 4):
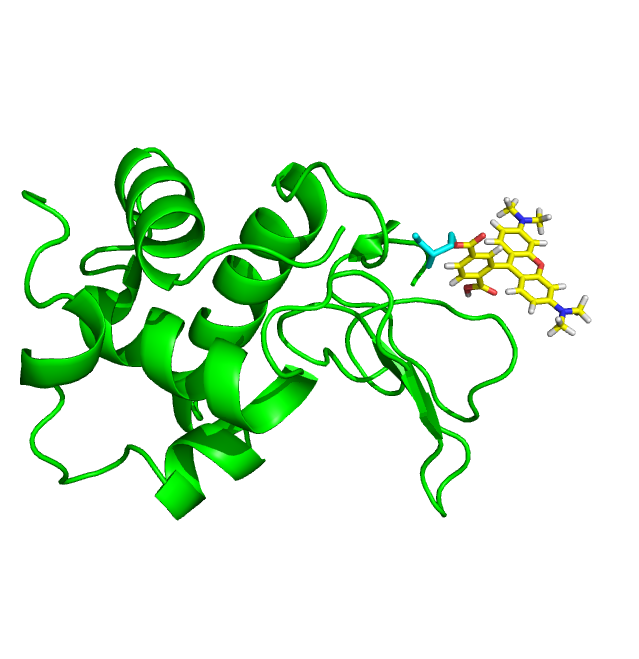
Рис. 4. Белок 1LMP (зеленый) с присоединенной флуоресцентной меткой (желтый) к SER'81.
5. Поли-аланиновая альфа спираль
Для создания поли-аланиновой альфа спирали длиной в 100 аминокислот я написала скрипт. На рисунке 5 предоставлено изображение цепочки.

Рис. 5. Поли-аланиновая альфа спираль.
6. Построение структуры кубана
SMILES нотация кубана - C12C3C4C1C5C2C3C45.
echo C12C3C4C1C5C2C3C45 > cubane.smi
Далее я при помощи babel построила третичную структуру (рис. 6):
obgen cubane.smi > cubane.mol
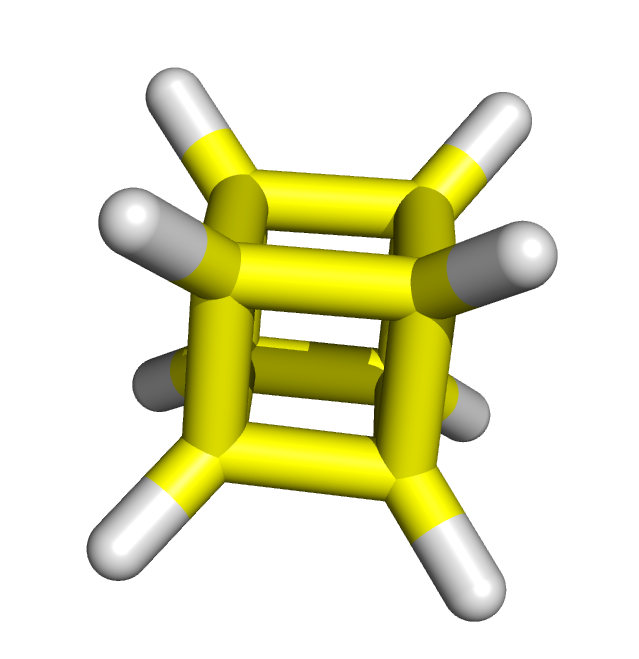
Рис. 6. Третичная структура кубана.