Вычисление параметров для молекулярной механики
1-2.
Была дана оптимизированная структура этана в виде z-matrix, где вместо значений длин и углов связей стоят переменные. Необходимо создать порядка 20 разных файлов для расчёта энергии в Gamess с разными значениями по длине одной из связей. Для начала я добавила шапку для dft к файлу и сохранила его под названием et.inp:
$CONTRL COORD=ZMT UNITS=ANGS dfttyp=b3lyp RUNTYP=ENERGY $END $BASIS GBASIS=N31 NGAUSS=6 POLAR=POPN31 NDFUNC=1 $END $GUESS GUESS=HUCKEL $END $system mwords=2 $end $DATA
Чтобы проверить, работает ли файлик, я запустила Gamess. Всё работает без ошибок!
Ссылки на файлы
et.inpet.out
3.
Создаем файл скрипта make_b.bash, как описано в задании, поменяв значение стартовой длины изменяемой связи так, чтобы она соответствовала файлу et.inp:
#!/bin/bash
for i in {-10..10}; do
nb=$(echo "scale=5; 1.52986 + $i/50" | bc -l)
sed "s/cc=1.001/cc=$nb/" et.inp > b_${i}.inp
done
Запускаем скрипт:
bash ./make_b.bash
На выходе получаем 21 inp файл, при этом в каждом разное значение для переменной cc.
Ссылки на файлы
make_b.bash4.
Затем необходимо запустить рассчет для полученных файлов. Для этого приписываем к файлу скрипта перед строкой done следующее:
gms b_${i}.inp 1 > b_${i}.log
Чтобы извлечь значения инергии из log файлов, используем команду awk. Перед строкой gms ставим знак # (комментируем), а после вводим:
awk '/TOTAL ENERGY =/{print $4}' b_${i}.log
Чтобы вывести значения длин связей, ставим следующее перед командой awk:
echo -n "$nb "
Запускаем скрипт и пишем результат в файл:
bash ./make_b_awk.bash > bond
Результаты:
1.32986 -79.7339315289 1.34986 -79.7406173737 1.36986 -79.7462868578 1.38986 -79.7510331813 1.40986 -79.7549412921 1.42986 -79.7580886263 1.44986 -79.7605457819 1.46986 -79.7623771323 1.48986 -79.7636413832 1.50986 -79.7643920816 1.52986 -79.7646780790 1.54986 -79.7645439543 1.56986 -79.7640303992 1.58986 -79.7631745699 1.60986 -79.7620104053 1.62986 -79.7605689179 1.64986 -79.7588784564 1.66986 -79.7569649422 1.68986 -79.7548520851 1.70986 -79.7525615748 1.72986 -79.7501132582
Ссылки на файлы
bondmake_b_gms.bash
make_b_awk.bash
5.
Далее необходимо построить зависимость энергии молекулы от длины одной связи. Для начала запустила gnuplot и ввела команду:
plot "bond"
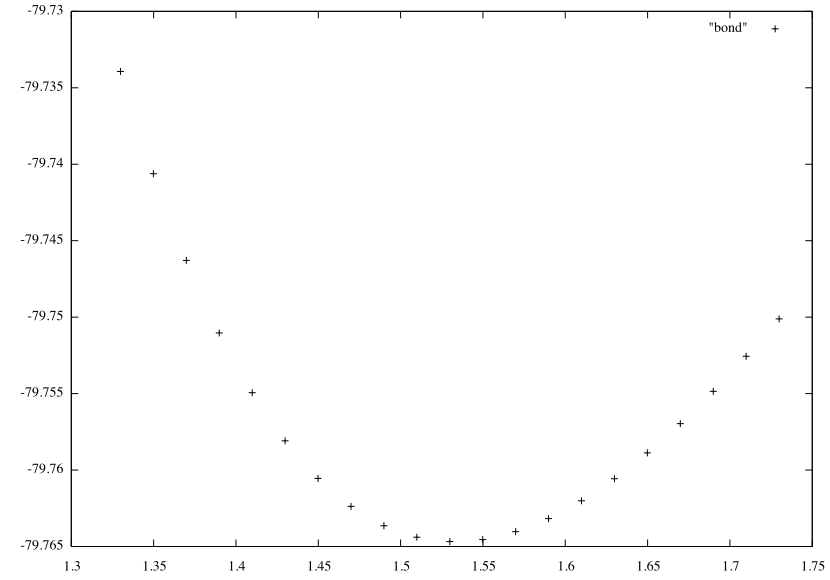
Рис. 1. Длина связи C-C.
На рисунке 1 видим график, похожий на параболу. Ищем коэффициенты в функции f(x)=a+k(x-b)^2, которые бы позволили наиболее близко описать наблюдаемую зависимость. Задаем функцию в развернутом виде:
f(x)=a + k*x*x - 2*k*x*b + k*b*b
и задаем стартовые значения коэффициентов:
a=-80 k=1 b=1.5
Подгоняем коэффициенты под имеющиеся точки в файле bond:
fit f(x) "bond" via a,k,b
Ниже представлены полученный коэффициенты:
Final set of parameters Asymptotic Standard Error ======================= ========================== a = -79.7652 +/- 0.0004522 (0.000567%) k = 0.563608 +/- 0.02335 (4.142%) b = 1.55432 +/- 0.002455 (0.1579%)
Рисуем графичек:
plot "bond", f(x)
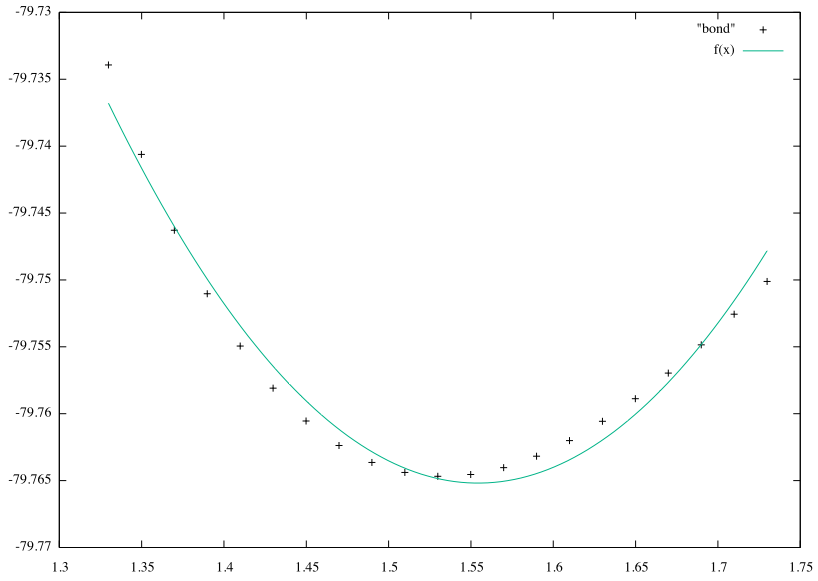
Рис. 2. Длина связи C-C.
Как можно видеть на рисунке 2, функция не совсем совпадает. Можно предположить, что в данном случае нужно использовать другую, более сложную функцию зависимости энергии связи от ее длины.
Ссылки на файлы
fit1.log6.
Необходимо проделать те же операции для валентного угла HCH, при этом его значения должны изменяться от 109.2 до 113.2.
109.200 -79.7645100964 109.400 -79.7645396600 109.600 -79.7645663953 109.800 -79.7645902988 110.000 -79.7646113672 110.200 -79.7646295973 110.400 -79.7646449862 110.600 -79.7646575313 110.800 -79.7646672302 111.000 -79.7646740809 111.200 -79.7646780815 111.400 -79.7646792309 111.600 -79.7646775278 111.800 -79.7646729717 112.000 -79.7646655623 112.200 -79.7646552997 112.400 -79.7646421847 112.600 -79.7646262180 112.800 -79.7646074011 113.000 -79.7645857358 113.200 -79.7645612241
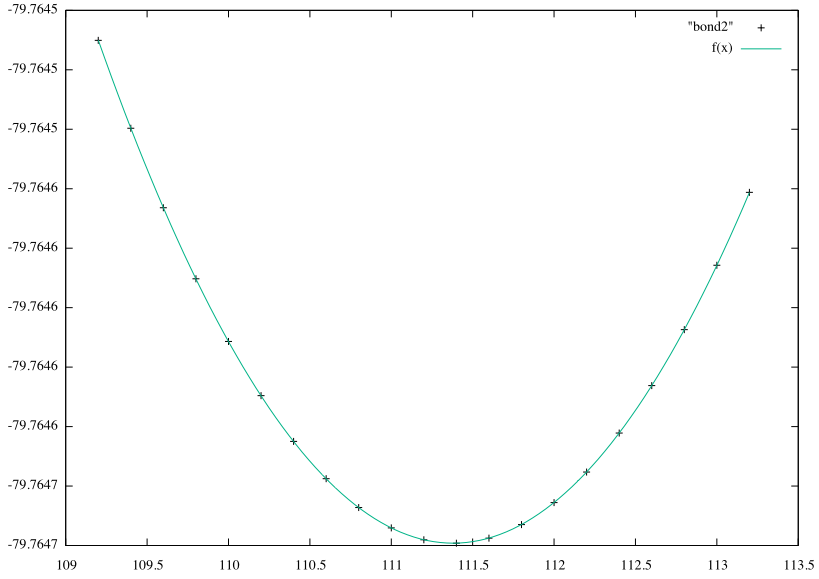
Рис. 3. Валентный угол HCH.
Ссылки на файлы
make_2.bashmake_2_gms.bash
make_2_awk.bash
fit2.log
7.
Повторяем процедуру для торсионного угла d3, его значения должны изменяться от -180 до 180 c шагом 12. Не проводим подгонку.
-180 -79.7646780790 -168 -79.7642336928 -156 -79.7630628377 -144 -79.7616148156 -132 -79.7604407798 -120 -79.7599827599 -108 -79.7604407798 -96 -79.7616148156 -84 -79.7630628377 -72 -79.7642336928 -60 -79.7646780790 -48 -79.7642336928 -36 -79.7630628377 -24 -79.7616148156 -12 -79.7604407798 0 -79.7599827599 12 -79.7604407798 24 -79.7616148156 36 -79.7630628377 48 -79.7642336928 60 -79.7646780790 72 -79.7642336928 84 -79.7630628377 96 -79.7616148156 108 -79.7604407798 120 -79.7599827599 132 -79.7604407798 144 -79.7616148156 156 -79.7630628377 168 -79.7642336928 180 -79.7646780790
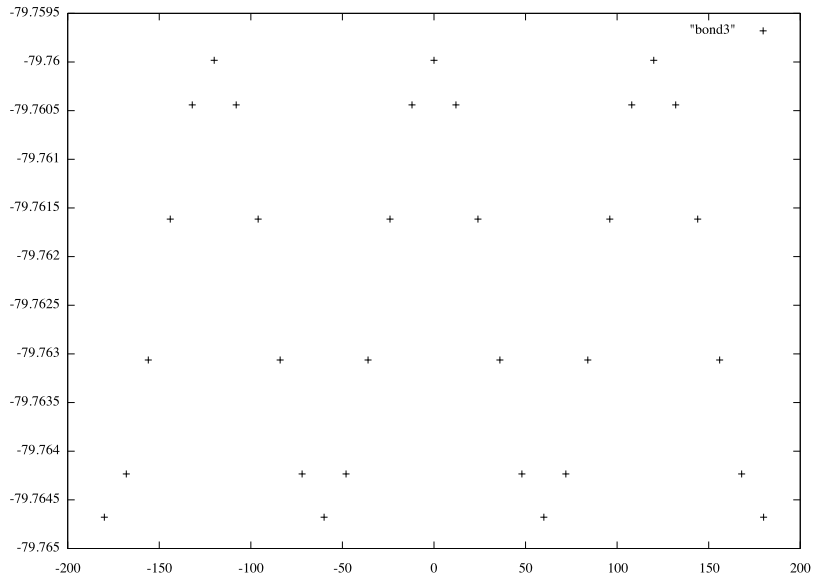
Рис. 4. Торсионный угол d3.
Важно понимать, что -180 и +180 - это один и тот же угол, а значит, мы видим три минимума и три максимума.
Ссылки на файлы
make_3.bashmake_3_gms.bash
make_3_awk.bash