Задание 1.
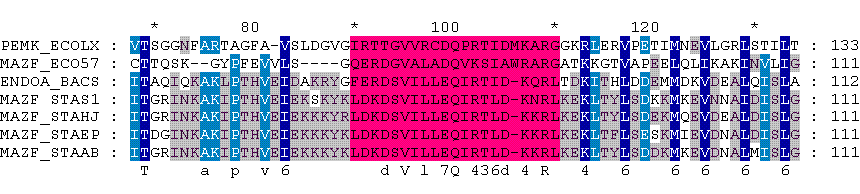
Выбранный для паттерна фрагмент выделен малиновым цветом.
" Создание паттернов аминокислотных последовательностей"
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности | F-E-R-D-S-V-I-L-L-E-Q-I-R-T-I-D-K-Q-R-L | 1 | найдена лишь последовательность моего белка ENDOA_BACSU |
| Сильный | [FILQ]-[DER]-[KRT]-[DT]-[GS]-V-[AIV]-[LR]-[ACL]-[DE]-Q-[IPV]-[KR]-[ST]-[IL]-[AD]-X(0,1)-[KR]-[AKNQ]-R-[GL] | 20 | найдены все последовательности |
| Слабый | [DER]-[KRST]-[DT]-[GS]-V-[AILV]-[LR]-[ACL]-[DENQ]-Q-[ILPV]-[KR]-[ST]-[ILMV]-[AD]-X(0,3)-[KR]-[AKRNQ] | 20 | ВСЕ |
При поиске последовательностей по сильному паттерну было найдено 20 последовательностей, однако все остальные последовательности, не входящие в множественное выравнивание, по которому делался паттерн, принадлежат одинаковым белкам организмов одного рода.
Это было предсказуемо изначально, ведь паттерн строился по выравниванию, в котором большинство белков из организмов того же рода.
Такое небольшое количество найденных последовательностей еще раз говорит о редкости белка.
При поиске последовательностей по слабому паттерну нашлись те же самые 20 белков. В таблице указан первоначальный слабый паттерн, который однако не намного отличается от сильного. Но в ходе выполнения работы я провела поиск по еще более слабым паттернам:
[DER] -[KRST]-[DT] -[GS] -V-[AILV] -[LR] -[ACL]-[DENQ]-Q-[ILPV] -[KR]-[ST]-[ILMV]-[AD] -X(0,3)-[KR]-[AKRNQ] (табличный)
[DEKNRQ]-[KRST]-[DST]-[GST]-V-[AILMV]-[ILRK]-[ACL]-[DENQ]-Q-[ILMPV]-[KR]-[ST]-[ILMV]-[ADENQ]-X(0,3)-[KR]-[AKRNQ]
[KRST]-[DST]-[GST]-V- X -[ILRK]-[ACL]-[DENQ]-Q- X -[KR]-[ST]-[ILMV]-[ADENQ]-X(0,3)-[KR]
[KRST]-[DST]-[GST]-V- X -[ILRK]-[ACL]-[DENQ]-Q- X -[KR]-[ST]-[ILMV]-[ADENQ]
Однако результат все время оказывался одинаковым: 20 одних и тех же белковых последовательностей.
Создание слабого паттерна:
[FILQ]-[DER]-[KRT] -[DT]-[GS]-V-[AIV] -[LR]-[ACL]-[DE] -Q-[IPV] -[KR]-[ST]-[IL] -[AD]-X(0,1)-[KR]-[AKNQ]-R-[GL]
[DER]-[KRST]-[DT]-[GS]-V-[AILV]-[LR]-[ACL]-[DENQ]-Q-[ILPV]-[KR]-[ST]-[ILMV]-[AD]-X(0,3)-[KR]-[AKRNQ]
Обрезаны 1 позиция сначала и 2 позиции сконца; в 3 позицию добавлен S (на основании родства S-T); в 7 позицию добавлен L (на основании родства L-I-V ); в 10 позицию добавлены N, Q (родство D-N, E-Q), в 12 добавлен L (родство L-I-V); в 15 добавлены M, V (родство L-I-V-M); в 17 позиции ослаблены условия пропуска с(0,1)до (0,3).
Задание 2.
Все описанные в PROSITE мотивы в заданном белке ENDOA_BACSU
| Идентификатор документа PROSITE (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (регулярное выражение) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00016 | RGD | Cell attachment sequence | Паттерн | R-G-D | неспецифична | 1 |
| PS00008 | MYRISTYL | N-myristoylation site Сайт N-миристоилирования |
Паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | неспецифична | 1 |
| PS00006 | CK2_PHOSPHO_SITE | Casein kinase II phosphorylation site Сайт фосфорилирования казеин киназы II |
Паттерн | [ST]-x(2)-[DE] | неспецифична | 2 |
| PS00005 | PKC_PHOSPHO_SITE | Protein kinase C phosphorylation site Сайт фосфорилирования протеин киназы С |
Паттерн | [ST]-x-[RK] | неспецифична | 1 |