- Краткое описание структуры в файле 1F7V.pdb
В файле приведены координаты атомов следующих молекул из SACCHAROMYCES CEREVISIAE (YEAST), экспрессированные в ESCHERICHIA COLI : TRNA(ARG) (Аргининовая тРНК) (Цепь В); ARGINYL-TRNA SYNTHETASE (Аргинил-тРНК синтетаза EC: 6.1.1.19) (Цепь А) .
Для исследования была выбрана цепь В, представляющая Аргининовую тРНК со следующей последовательностью:
[901] 5' - PSU U C C U C G U 1MG 2MG C C C A A H2U G G H2U C A C G G C M2G PSU C U G G C U I C G A A C C A G A A G A H2U U 5MC C A G G 5MU PSU C A 1MA G U C C U G G C G G G G A A G C C A - 3' [976],
где 901 и 970 - номера первого и последнего нуклеотида.
На 3'-конце есть триплет CCA, к которому присоединяется аргинин. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями ( ссылка на выходной файл ). В соответствии с полученными данными
АКЦЕПТОРНЫЙ СТЕБЕЛЬ состоит из участка 901-906 и комплементарного ему участка 972-967 :
1 (0.043) B:.901_:[PSU]P-*---A[..A]:.972_:B (0.002) | 2 (0.002) B:.902_:[..U]U-----A[..A]:.971_:B (0.004) | 3 (0.004) B:.903_:[..C]C-----G[..G]:.970_:B (0.004) | 4 (0.005) B:.904_:[..C]C-----G[..G]:.969_:B (0.004) | 5 (0.003) B:.905_:[..U]U-*---G[..G]:.968_:B (0.008) | 6 (0.003) B:.906_:[..C]C-----G[..G]:.967_:B (0.011) |
Т-СТЕБЕЛЬ состоит из участка 949-952 и комплементарного ему участка 965-962:
8 (0.013) B:.949_:[5MC]c-----G[..G]:.965_:B (0.005) | 9 (0.004) B:.950_:[..C]C-----G[..G]:.964_:B (0.007) | 10 (0.006) B:.951_:[..A]A-----U[..U]:.963_:B (0.005) | 11 (0.010) B:.952_:[..G]G-----C[..C]:.962_:B (0.006) |
D-СТЕБЕЛЬ состоит из участка 910-913 и комплементарного ему участка 925-922:
21 (0.007) B:.910_:[2MG]g-----C[..C]:.925_:B (0.008) | 22 (0.003) B:.911_:[..C]C-----G[..G]:.924_:B (0.010) | 23 (0.004) B:.912_:[..C]C-----G[..G]:.923_:B (0.012) | 24 (0.010) B:.913_:[..C]C-*--xC[..C]:.922_:B (0.005) |
АНТИКОДОНОВЫЙ СТЕБЕЛЬ состоит из участка 939-942 и комплементарного ему участка 931-928:
15 (0.008) B:.939_:[..C]C-----G[..G]:.931_:B (0.010) | 16 (0.003) B:.940_:[..C]C-----G[..G]:.930_:B (0.007) | 17 (0.006) B:.941_:[..A]A-----U[..U]:.929_:B (0.005) | 18 (0.010) B:.942_:[..G]G-----C[..C]:.928_:B (0.004) |
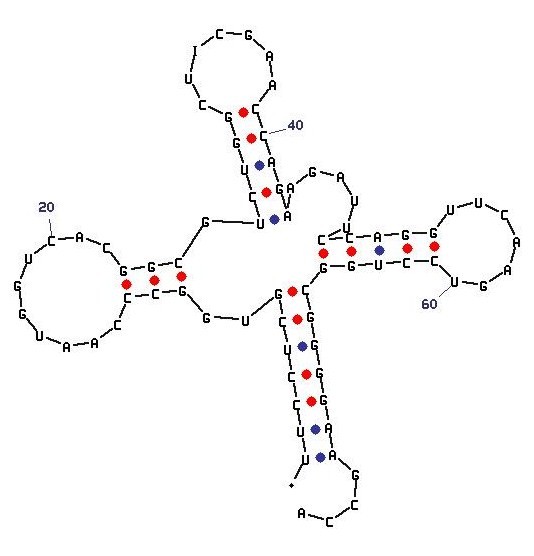
Рис.1. Вторичная структура Аргининовой тРНК из SACCHAROMYCES CEREVISIAE Скрипт для получения изображения 
акцепторный стебель выделен красным
Т-стебель - зеленым
D-стебель - синим
антикодоновый - оранжевымselect all color white wireframe off backbone 70 select 901-906 or 967-972 color red select 949-952 or 962-965 color green select 910-913 or 922-925 color blue select 939-942 or 928-931 color orange
Структуру стеблевых дуплексов поддерживают 12 канонических и 4 неканонических пар оснований. Неканоническая пара: (C913-C922)
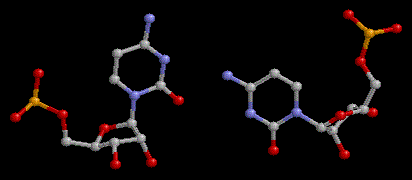
Можно отметить также
а)наличие вариабельной петли (944-948);
а)отсутствие остатка тимидина в Т-петле;
а)наличие дигидроуридинов в D-петле (916, 919):
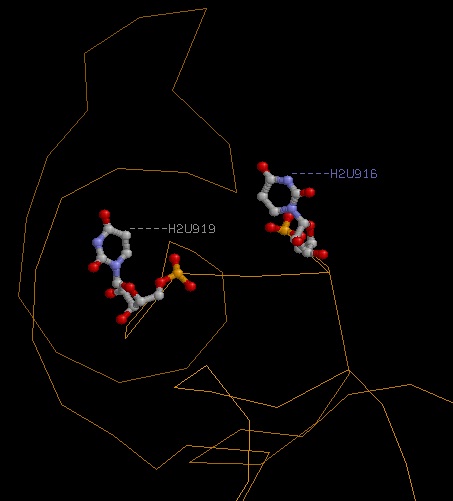
- Исследование третичной структуры
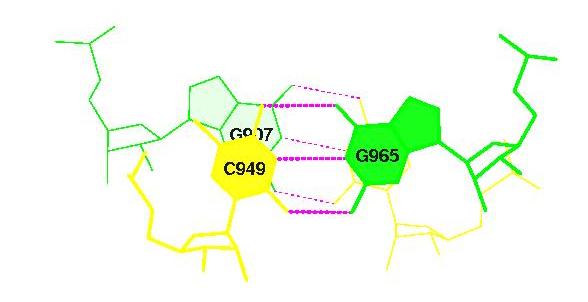
Стекинг-взаимодествие между основаниями акцепторного (G907-C966) и T-стебля (C65-G49).С площадью перекрываия ( 5.71)
2. Дополнительные водородные связи между основаниями D- и Т-петель.
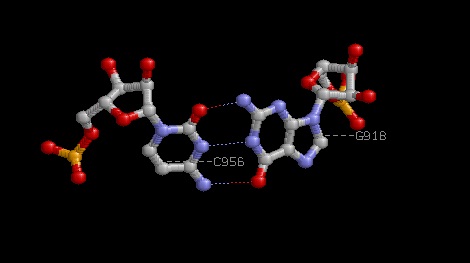
Есть две водородные связи между основаниями D- и Т-петель:14 (0.044) B:.955_:[PSU]Px**+xG[..G]:.917_:B (0.009) 27 (0.010) B:.918_:[..G]G-----C[..C]:.956_:B (0.003)
Например пара G918-C956:
Также есть дополнительные водородные связи между основанием D-петели и основанием акцепторного стебля:
25 (0.003) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.004)
и основанием D-петели с основанием антикодонового стебля:
26 (0.005) B:.915_:[..A]Ax**+xU[..U]:.948_:B (0.006)
- Предсказание вторичной структуры тРНК
1. Сохранить тРНК последовательность в fasta формате.
2. Заменить модифицированные нуклеотиды на стандартные. (инозин не менялся)
3. P=10
4. Получилась более-менее приятная картинка. В ней присутствуют все петли, в том числе и вариабельная.
Для быстрого поиска инвертированных последовательностей была использована программа einverted. Рекомендуемые параметры для einverted:
Gap penalty = 3
Minimum score threshold = 5
Match score = 3
Mismatch score = -3
tRNA: Score 15: 5/5 (100%) matches, 0 gaps 27 tctgg 31 ||||| 43 agacc 39 tRNA: Score 15: 5/5 (100%) matches, 0 gaps 49 ccagg 53 ||||| 65 ggtcc 61
Участок структуры
Позиции в структуре
(по результатам find_pair)Результаты предсказания
по алгоритму ЗукераАкцепторный стебель 5' 901-907 3'
5' 966-972 3'
Всего 7 парПредсказано 7 пар из 7 реальных D-стебель 5' 910-913 3'
5' 922-925 3'
Всего 4 парыПредсказано 4 пары из 4 реальных T-стебель 5' 949-953 3'
5' 961-965 3'
Всего 5 парПредсказано 5 пар из 5 реальных Антикодоновый стебель 5' 939-943 3'
5' 927-931 3'
Всего 5 парПредсказано 5 пар из 5 реальных Общее число канонических пар нуклеотидов 21 21