EnsEMBL
Первое, что сразу отметилось как приятное и полезное - это выделение трех организмов: человек, мышь и zebrafish(данио-рерио или брахиданио-рерио)-
так как они одни из самых употребляемых. Перейдем на страницу для человека. На ней содержится описание данных в этой версии сборки. Поищем ген HLA-G сначала по названию.
Получаем на страничке результатов табличку с ссылками:

При переходе по ссылке Gene Human нам выдается список находок, удовлетворяющих запросу 'HLA-G' у человека.Насколько я поняла, в моем случае все 7 находок удовлетворяли одному и тому же гену, но для него записи отличались. В каждой записи было свое расположение гена. Правильно ли я поняла, что HSCHR6_MHC_COX:29788431-29792600:1 означает: HSCHR- в файлах с таким названием содержится информация о вариантах гаплотипа, а в целом речь идет о том, что искомый ген содержится в области (MHC_COX:29788431-29792600:1)хромосомы 6?
Перейдя по эттой ссылке мы получаем изображение хромосомы с выделенным регионом и изображение региона, на котором желтым цветом выделена искомая последовательность HLaA-G. Если нажать на наш ген или любой другой, то появляется табличка с краткими данными о нем:
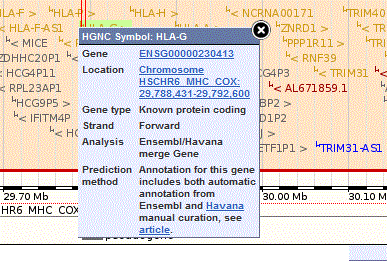
Кроме того, на этом же первом изображении выделено разными цветами:
Гены, кодирующие белки- красным или желтым; псевдогены- серые; некодирующие гены- голубые; РНК-кодирующие гены- светло-фиолетовые.
Кроме того оттенками синего выше размечены континги.
Еще выше есть масштаб изображения - полоска с указанием - 1 Mb.
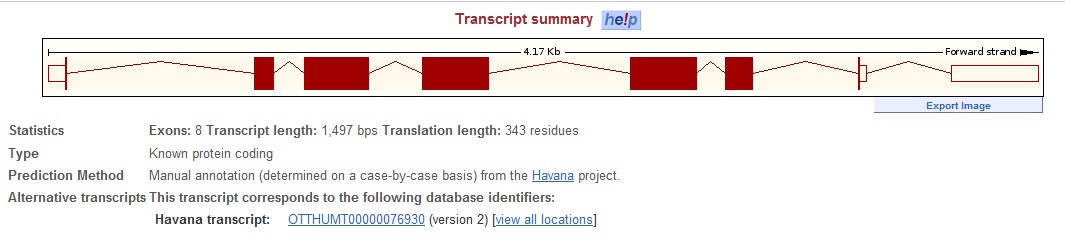
Картинка ниже представляет данные о транскриптах с гена HLA-G.
Также открывается вкладочка с данными по этим транскриптам. В таблице приводятся имя транскрипта, его ID, длина, ID белка, длина белка, тип этого транскрипта и CCDS. CCDS - это
кодирующая последовательность в наборе конценсусов кодирующей последовательности проаннотированная Ensembl, Vega, UCSC и NCBI. Это я вынесла из глоссария EnsEMBL, расположенного в разделе "Help & Documentation".
Причем в этой таблице можно добавлять и убирать отображаемые колонки. Если нажать на ссылки в ID белка, то появляется информация о белке:
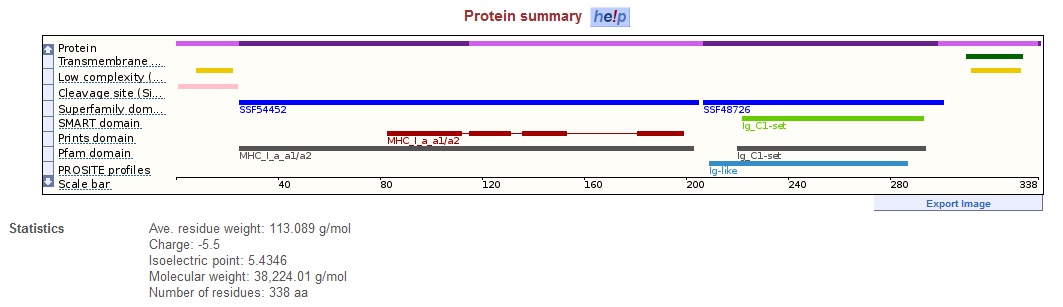
Та же возможность есть и для транскрипта. Причем опять открывается отдельная вкладочка в EnsEMBL, что очень удобно - позволяет всегда вернуться назад, не потеряв результата.
Левое меню предоставляет много возможностей. Например для транскрипта HLA-G-002 при нажатии на кнопку Supporting evidence (48) открывается изображение:

Насколько я поняла из объяснения,открывающегося при нажатии на кнопку "help" перед изображением:
Ensembl/Havana транскрипты являются результатом выравнивания белка и сDNA (снова из глоссария: это ДНК, полученная обратной транскрипцией с мРНК, т.е это ДНК версия мРНК) последовательностей генома.
И это изображение нам представляет как раз те самые сDNA и EST (Expressed Sequence Tags) и белки.
Экзоны представлены красными прямоугольниками вверху. Данные Ensembl находятся в верхней половине, внизу на более темном фоне расположены данные Havana.
Спускаемся ниже по меню: Exons (8). Появляется таблица с последовательностями экзонов и интронов и фланкирующих последовательностей для одного транскрипта - HLA-G-002. Причем последовательность написана в направлении 5' к 3' вне зависимости от того, на какой цепи находится ген. Очень удобно - что эту таблицу можно скачать. Экзоны на выравнивании выделены заглавными буквами: нетранслируемая область - фиолетовым цветом, кодирующая последовательность - черным. Интроны строчными голубыми буквами, фланкирующие последовательности строчными зелеными.
При нажатии в меню на кнопку "General identifiers" (290) появляются внешние ссылки на другие банки данных (напр. UniProtKB/Swiss-Prot)для каждого белка приводится ID и выравнивание белка с транскриптом. Также просто на наши транскрипты в других банках.
Далее полазив по этому меню, можно найти информацию о белках, их доменах, описаниях в Pfam.
Вернемся к самой первой табличке с результатами. При переходе по ссылке Somatic mutation открываются записи с мутациями, ссылающимися на этот ген. Следующая графа Transcript вновь возвращает нас к всем транскриптам этого гена. Variation отправляет к вариациям это гена (?).
При поиске по последовательности через BLAST/BLAT открывается изображение положения гена на кариотипе:
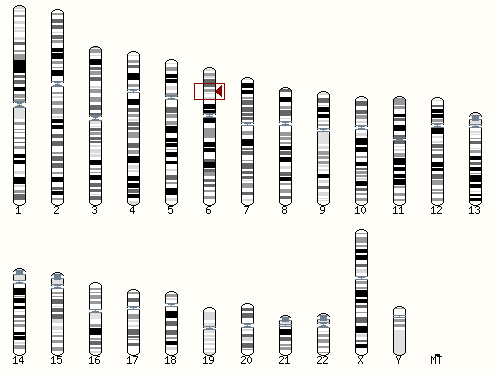
А ткже расположение выравниваний относительно поданной на вход последовательности и таблица с соответствующими выравниванию ссылками: на выравнивания, участки генома, на описания структуры участка.
Также можно изменять колонки таблицы.
Пройдем по гиперссылке "Contig view", обозначенной маленькой буквой "C", и снова выходим на страницу, на которую попадали по первой ссылке из таблицы.
Vega
В этом браузере геномы только человека, гориллы, мыши, кабана, собаки, кенгуру Валлаби, рыбы Danio rerio. В остальном же он несильно отличается от EnsEMBL, разве только содержит результаты ручного аннотирования HAVANA. При этом в отличие от EnsEMBL здесь меньше возможностей в левом меню.
NCBI
В нем также показывается где искомый ген располагается в хромосоме, в каком регионе. При этом со страницы результата поиска есть ссылки на само описание гена, на соответствующие ему континги, на запись белка, на выравнивание матричной РНК и искомой последовательности, на найденные экзоны, ссылку на выравнивания с гомологичными генами, на STS, CCDS. В целом можно найти все то, что было и в "Ансамбле", но с большими затратами сил, к тому же нет такой порой необходимой кнопочки HELP.
UCSC
Более скромное оформление, зато очень легко можно увеличивать до нужного размера нужную область. Сразу представленыизображение выравниваний гена с геном других организмов.