Задание 1. Построение дерева по нуклеотидным последовательностям.
Поискав ссылки на запись EMBL, описывающую полный геном каждой из бактерий, в соответствующих записях Swiss-Prot, описывающих белок EFG, получили нуклеотидные последовательности 16S рРНК.
При этом проблемы возникли с STRPN. В полном геноме штамма, на который ссылается запись Swiss-Prot EFG_STRPN, рРНК неразмечены. Пришлось использовать BLASTN, хотя потом нашелся штамм Streptococcus pneumoniae с размеченными рРНК. Последовательность из него и использовалась для выравнивания.
Использованные AC записей EMBL, описывающих полные геномы, координаты рРНК в этих записях: список .
Полученные нуклеотидные последовательности в fasta-формате (команда seqret -sask): файл
Полученное выравнивание, с помощью программы muscle: выравнивание
На основании выравнивания постороено с помощью программы fdnaml неукорененное дерево:
+-----CLOTE
|
| +BACAN
| +--4
| | +-BACSU
| |
1---3 +--LACAC
| | +---6
| | | +LACDA
| +--5
| | +--STRPN
| +--2
| +--LACLM
|
+-------FINM2
Верное дерево:
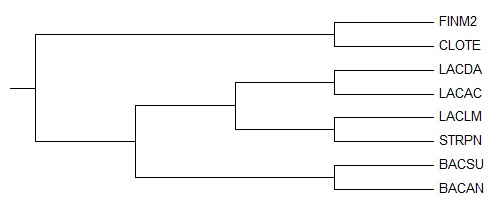
Как видно из изображений деревьев, при правильном укоренении верхнего они идентичны: совпали все ветви.
Используя матрицу расстояний (fdnadist) с помощью программы fkitsch (метод Фитча – Марголиаша ) было построено другое дерево, которое, однако, хуже предыдущего:
Вместо {LACDA, LACAC, LACLM, STRPN} vs {FINM2, CLOTE, BACSU, BACAN} присутствует ветвь {LACDA, LACAC, BACSU, BACAN} vs {FINM2, CLOTE, LACLM, STRPN}
+-LACAC
+--7
! +-LACDA
+-6
! ! +-BACAN
! +--5
+-4 +-BACSU
! !
! ! +---STRPN
+-2 +-3
! ! +---LACLM
--1 !
! +-----CLOTE
!
+------FINM2
Задание 2. Построение и анализ дерева, содержащего паралоги.
Найдем в выбранных бактериях достоверные гомологи белка CLPX_BACSU.
Чтобы найти гомологов в заданных организмах, воспользуемся файлом proteo.fasta, где лежат записи банка UniProt. Создаем индексные файлы для поиска по файлу proteo.fasta:
formatdb -i proteo.fasta -p T -n base
Необходимо провести поиск программой BLASTP гомологов (с порогом на E-value=0,0001) и отобрать по мнемонике видов только те находки, которые относятся к отобранным бактериям.
blastall -p blastp -d base -i CLPX_BACSU.fasta -e 0.0001 -o blastp.out
Результат поиска blastp.out
Список отобранных для дальнейшей работы белков: список
Затем выравниваем полученные последовательности программой muscle. Строим матрицу расстояний программой fprotdist. Подаем этот файл на вход программе fneighbor.
Дерево гомологов:
+-------FTSH_STRPN
!
! +-HSLU_LACAC
! +---3
! ! +-HSLU_LACDA
! +-----5
! ! ! +-HSLU_BACAN
! ! +-4
! ! +-CLPY_BACSU
1---------------------------6
! ! +--------CLPC_BACSU
! ! +-------------------2
! ! ! +----CLPE_BACSU
! ! !
! +-7 +---B0S2N5_FINM2
! ! +-10
! ! ! +--CLPX_CLOTE
! ! !
! +-----11 +-CLPX_LACLM
! ! +-9
! ! ! +--CLPX_STRPN
! +-12
! ! +CLPX_BACSU
! +-8
! +CLPX_BACAN
!
+--------B0S0E3_FINM2
Два гомологичных белка будем называть ортологами, если они:
а) из разных организмов;
б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами.
Можно определить, считая, что дерево реконструировано верно:
Ортологи:
1) HSLU_LACAC и HSLU_LACDA
2) HSLU_BACAN и CLPY_BACSU
3) B0S2N5_FINM2 и CLPX_CLOTE
4) CLPX_LACLM и CLPX_STRPN
5) CLPX_BACSU и CLPX_BACAN
Паралоги:
1) CLPC_BACSU и CLPE_BACSU