Задание 1.
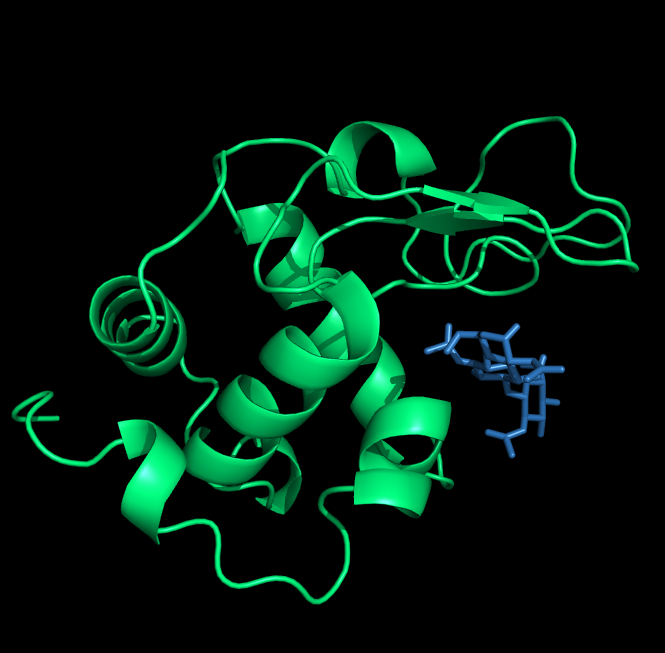
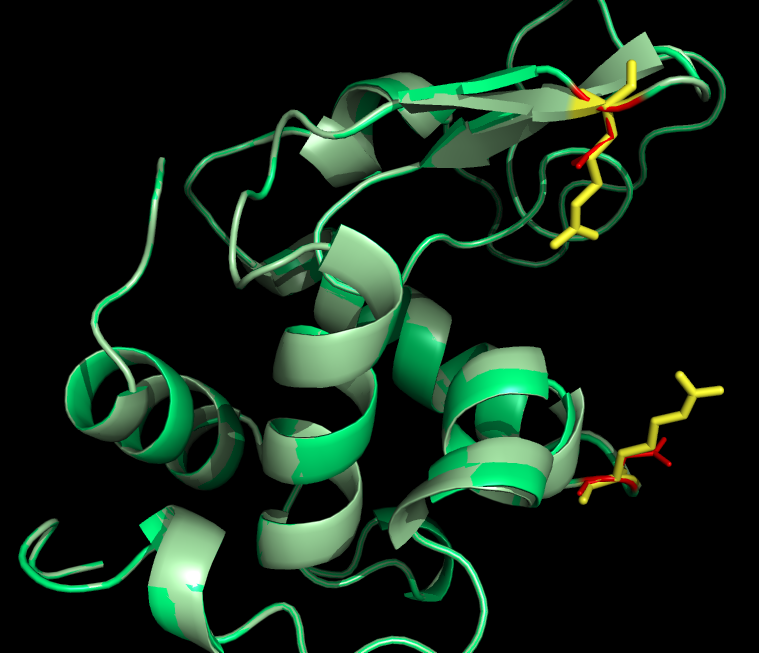
Для работы дан белок 1LMP. На рисунке показана его структура с лигандом (синий) - остатки 130, 131, 132.
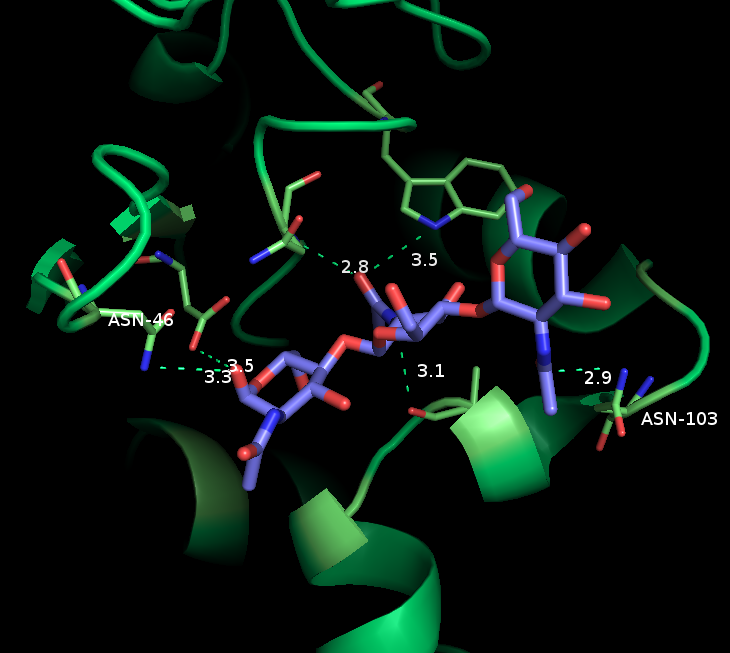
Оценила район возможного взаимодействия остатков белка с лигандом. Выделены остатки, которые могут образовывать водородные связи, на расстоянии 3.5 А от атомов N и О лиганда.
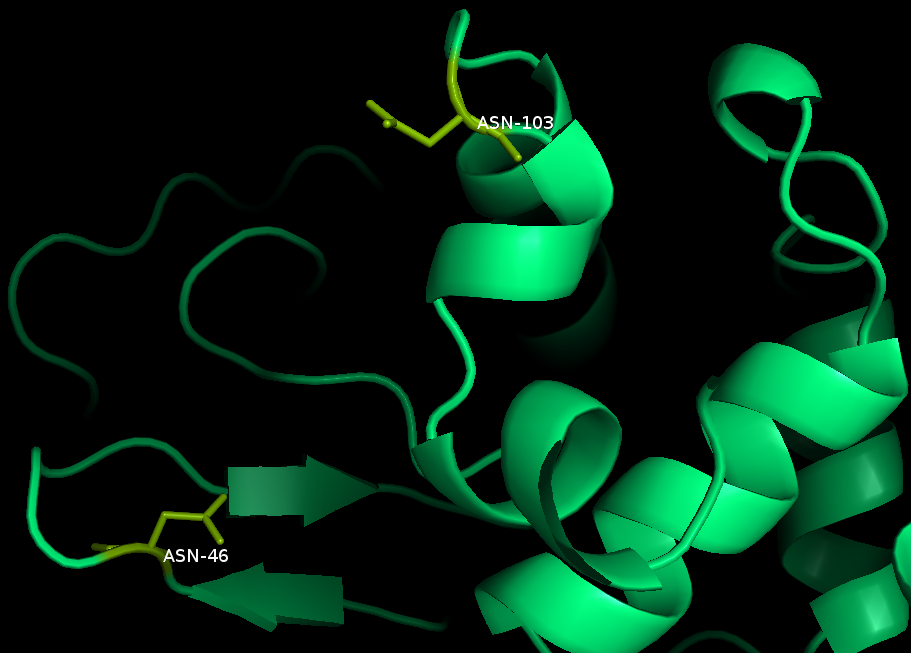
Показаны 2 остатка по которым провела мутагенез. Один из них располагался ближе остальных к лиганду и, если заменить его на более объемный остаток, возможно это помешает связыванию лиганда.
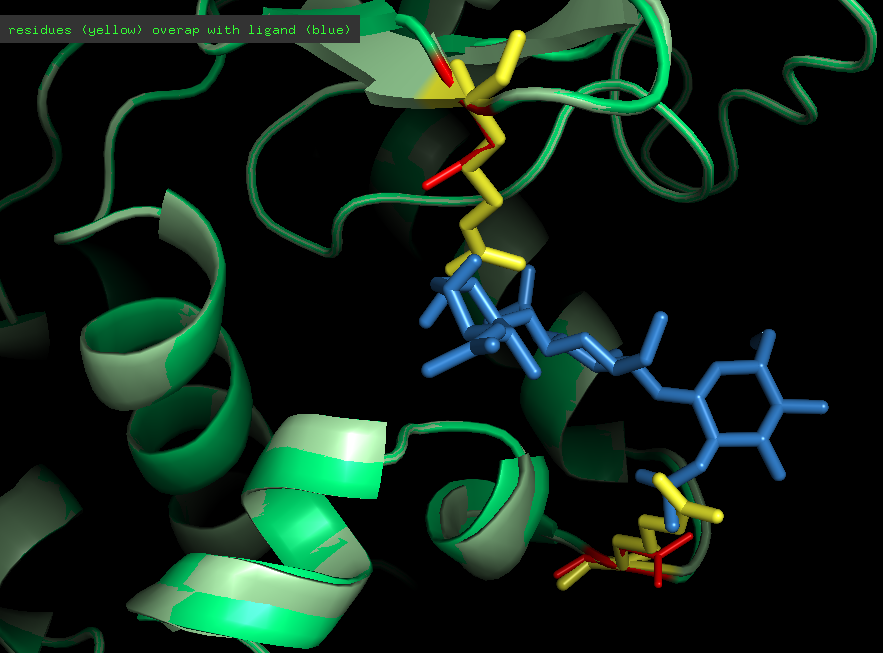
Выравнивание 2 белков: 1LMP и мутантный. Несовпадающие остатки показаны: красный - Asn(исходный), желтый - Arg(после мутации)
Мутантные аминокислоты перекрываются с лигандом, мешая ему связаться.
Показаны 2 структуры. Слева исходная, способная связать лиганд, справа - структура после мутагенеза.
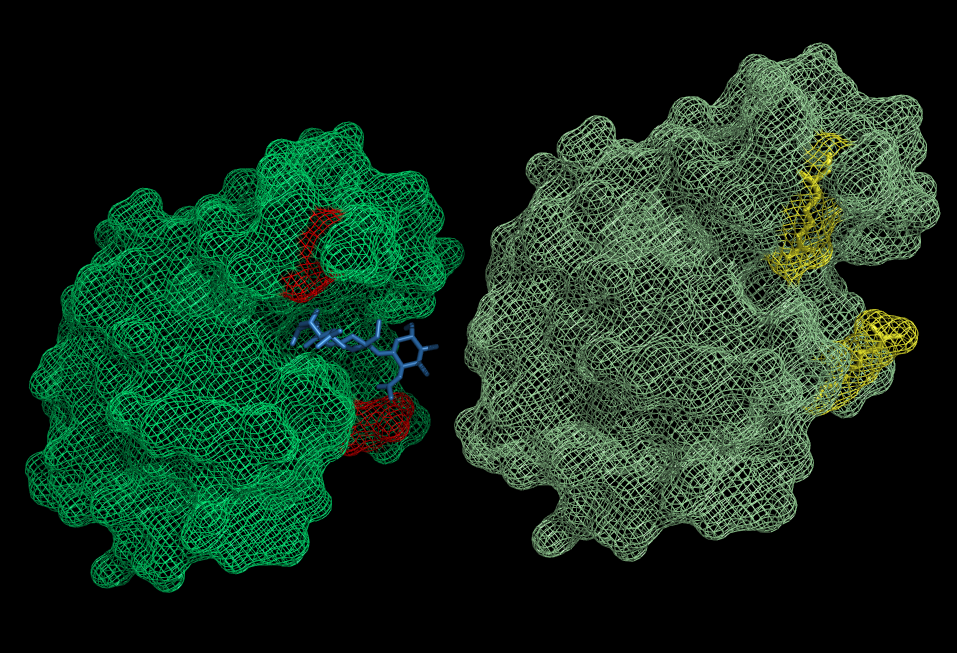
Но вообще, все гораздо нагляднее в мини-ролике о проведенном мутагенезе. РОЛИК
Задание 2.
Пришитая флюресцентная метка TAMRA к Ser36.
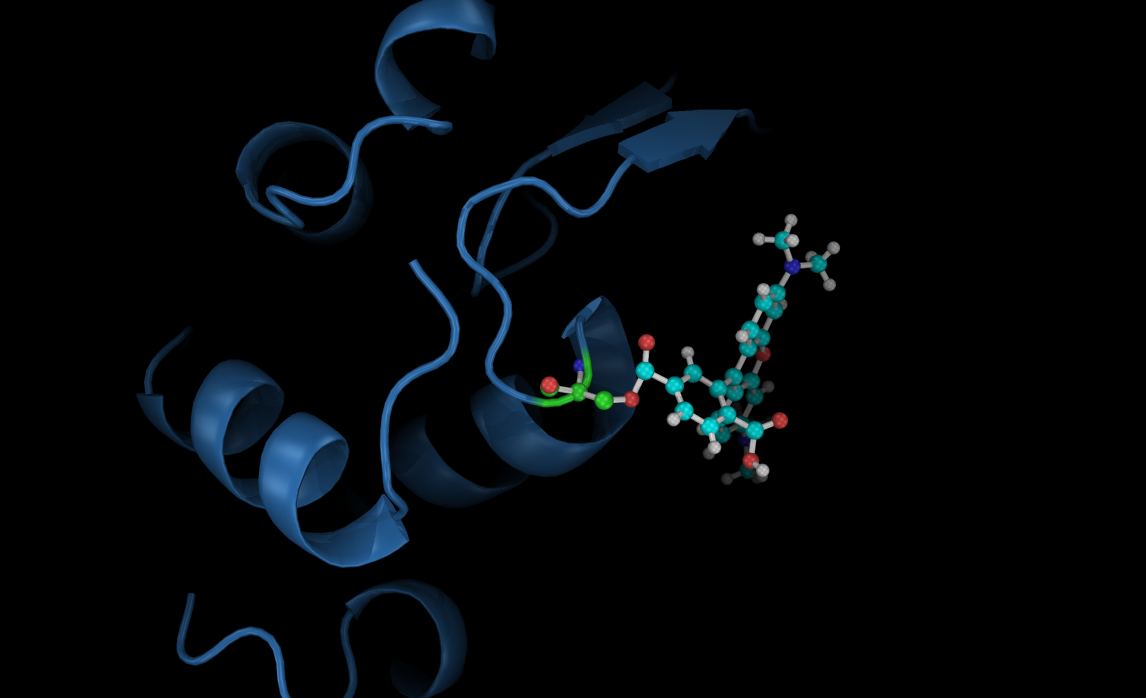
Задание 3.
Полилейциновая последовательность (100 а.о.). Измерила углы &phi и &psi в альфа-спирали белка 1LMP. Они оказались: &phi = -73.5, &psi = -43.
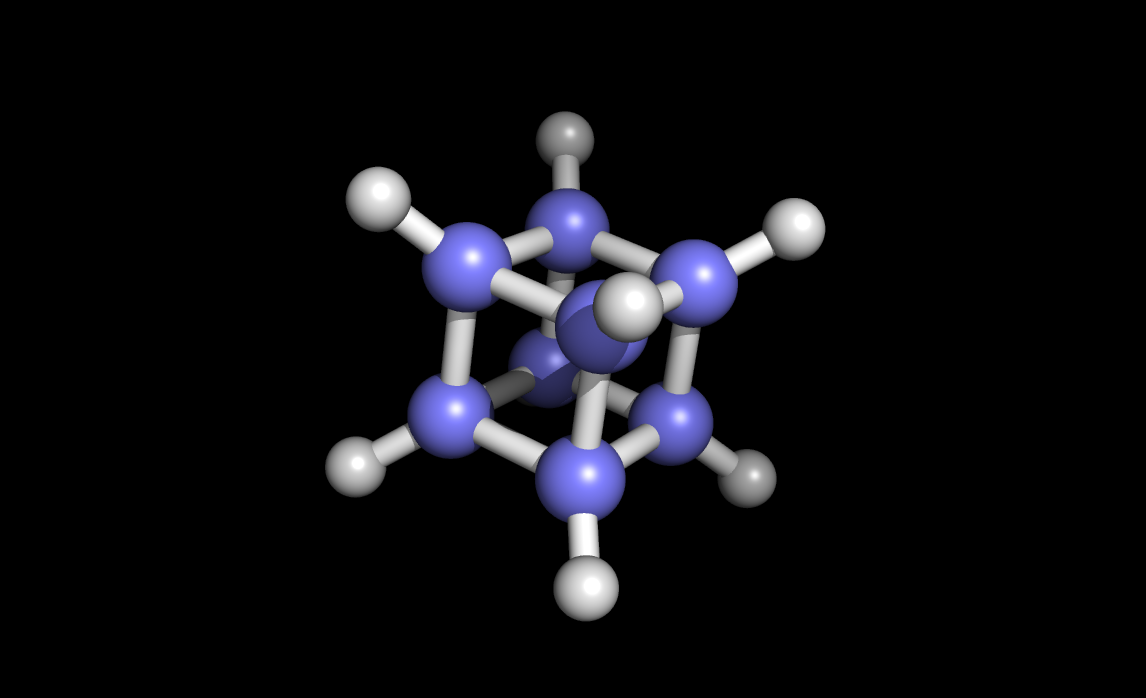
Измерила эти углы в построенной полилейциновой последовательности: -139 и 135. В соответствии с этими данными внесла корректировку в углы в скрипте. Задание 4. Используя возможности babel строить третичную структуру вещества на основе SMILE нотации, построена молекула кубана
(кубан в SMILE сохранен в файле cub.smi (obgen cub.smi > cub.mol))

Скрипт:
cmd.fragment ("leu")
for x in range(2, 101):
cmd.edit("resi "+ str(x)+" and name C")
editor.attach_amino_acid("pk1","leu")
cmd.edit("resi "+str(x)+" and name N", "resi "+str(x)+" and name CA")
cmd.torsion(65)
cmd.edit("resi "+str(x)+" and name C", "resi "+str(x)+" and name CA")
cmd.torsion (-178)
cmd.zoom ("all")
cmd.center ("all")