1. Было построено выравнивание последовательности(с помощью Сlustal в формате PIR) из структуры 1LMP и последовательности Q37896 (LYS_BPB03) из Bacillus phage B103. Эта исследуемая последовательность имеет 263 а.к., таким образом, лиганд будет иметь 264 номер при моделировании.
Выравнивание было изменено - изменены названия последовательностельностей (seq - для Q37896 и 1lmp- для последовательности из PDB-банка). Добавлены строки:
sequence:XXXXX::::::: 0.00: 0.00 # для моей последовательности structureX:1lmp_now.ent:1 :A: 132 :A:undefined:undefined:-1.00:-1.00 # для последовательности с известной структурой
Полученное выравнивание: alig_2.pir
2. Скачана из PDB-банка структура 1LMP, в ней удалена вода и изменены названия атомов лиганда: для каждого остатка лиганда к атому добавлена буква А, B, C. Затем номер лиганда заменен на 130 для всех остатков. Полученная структура: 1lmp_now.ent
3. Для написания скрипта для моделирования проведен анализ структуры. Выявлены остатки, взаимодействующие с лигандом (находящиеся в радиусе 3.5 А и способные к формированию водородной связи). Все они изображены на рисунке:
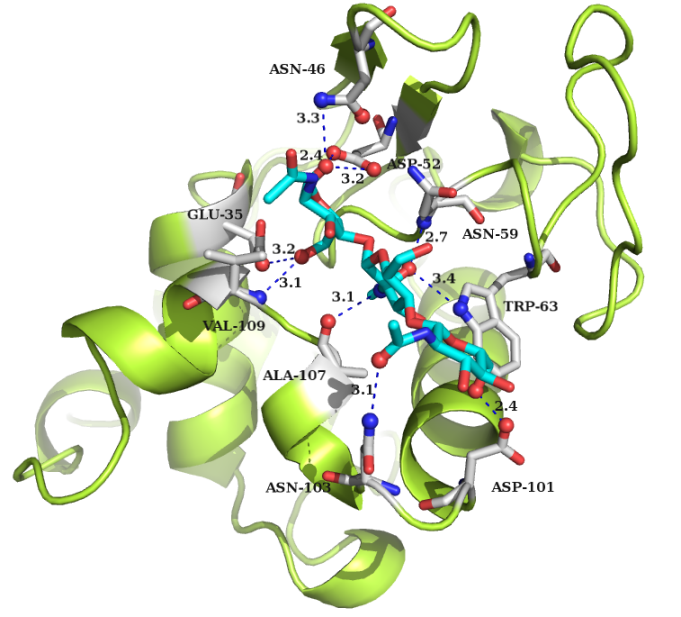
В выравнивании они отмечены цветом. Показаны атомы, взаимодействующие с лигандом. (Черные надписи). Для белка с неизвестной структурой предположены атомы контактов. (Красные надписи).
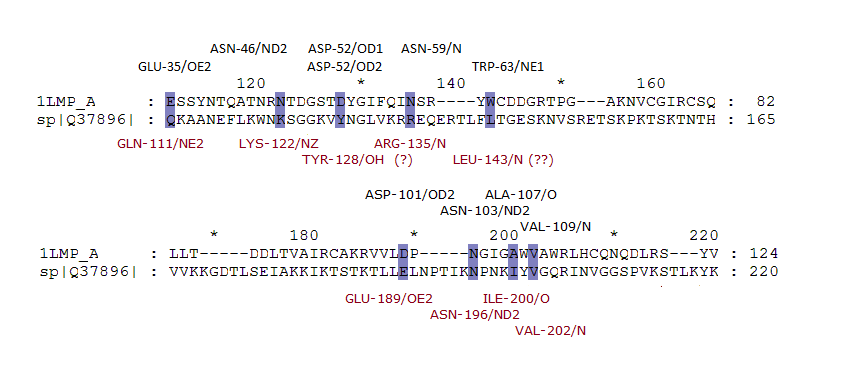
Сильно вызывают сомнения в исследуемой последовательности контакты лиганда с остатками Tyr122 и Leu143. Поэтому они не участвуют в скрипте.
Вообще для использования в скрипте я выбрала 4 а.к, которые либо несильно изменились от последовательности к последовательности, либо в которых можно обнаружить примерно похожие атомы для контакта с лигандом. Также мне показалось необходимым при выборе этих а.к., чтобы они лежали с обеих сторон от лиганда (т.е. были на разных сторонах кармана для лиганда). Итого у меня получилось:
А.К в 1LMP атом контакта(а.к.) атом в лиганде -> А.К. в seq атом контакта(а.к.) атом в лиганде
Asp-101 OD2 NAG'130/O6 Glu-189 OE2 NAG'264/O6A
Asn-103 ND2 NAG'130/O7 Asn-196 ND2 NAG'264/O7A
Val-109 N NDG'132/O6 Val-202 N NDG'264/O6C
Asn-59 N NAG'131/O7 Arg-135 N NAG'264/O7B
Ala-107 O NAG'131/N2 Ile-200 O NAG'264/N2B
4. Получено 5 моделей:
seq1.pdb
seq2.pdb
seq3.pdb
seq4.pdb
seq5.pdb
Вот они все вместе:
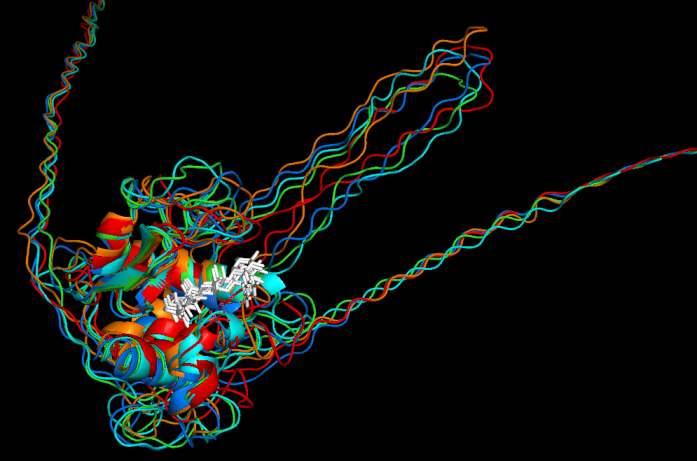
Видно, что для N- и С- концов структуры не получено (это было понятно и до этого, так-как для этих учатсков нет выравнивания с известной последовательностью), еще неструктурирована петля внутри по той же причине. Я решила далее убрать концы и анализировать уложенную часть
Ниже приведено их выровненное с известной структурой 1LMP:
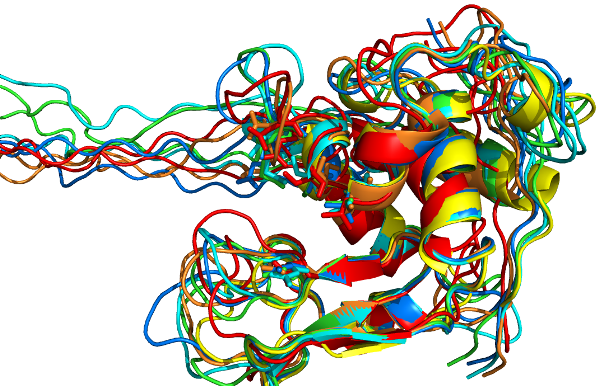
Цвета соответствуют:
желтый - 1LMP
оранжевый - seq1
красный - seq2
зеленый - seq3
синий - seq4
голубой - seq5
На следующем изображении серым отмечены остатки, контактирующие с лигандом, которые фиксировались при моделировании. (Цвета структур те же)
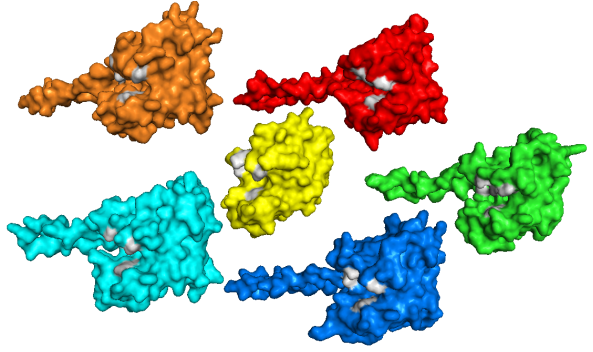
По выравниванию структур видно, что все они имееют одинаковый набор спиралей и листов, сходный со структурой 1LMP.
Из всех 5 полученных больше всех отличается структура 2 (красная), можно предположить, что она будет не самая правильная.
Также видно, что во всех моделях имеется карман для посадки лиганда, причем везде примерно одинаковый.
Проведем анализ структур, используя WHATIF -> Structure validation.
№ Ramachandran Anomalous bond lengths Anomalous bond angels модели Z-score Z-score Z-score 1 -1,253 0,985 1,639 2 -1,756 1,140 1,840 3 -1,039 0,968 1,373 4 -1,194 0,984 1,444 5 -1,117 0,963 1,371
Как и предполагалось, самая никудышная структура - вторая (красная). Самые приятные 5 и 3. (голубая и зеленая, соответственно)