Все выполненные команды в этом занятии:
obgen nag.smi > nag.mol
# создание 3D модели лиганда NAG по SMILES
babel -imol nag.mol -opdb nag.pdb
# конвертирование структуры лиганда в pdb
export PATH=${PATH}:/home/preps/golovin/progs/bin
# задание пути к скриптам
prepare_ligand4.py -h
# изучение функционирования скрипта prepare_ligand4
prepare_ligand4.py -l nag.pdb
# конвертирование pdb-файла лиганда в pdbqt-формат
prepare_receptor4.py -r prot.pdb
# конвертирование pdb-файла белка в pdbqt-формат (неподвижный белок)
vina --config vina.cfg --receptor prot.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
# докинг с неподвижной структурой белка
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r prot.pdbqt -s GLU189_ASN196_ARG135_ILE200
# задание подвижных остатков GLU189_ASN196_ARG135_ILE200 в белке
vina --config vina.cfg --receptor prot_rigid.pdbqt --flex prot_flex.pdbqt --ligand nag.pdbqt --out nag_prot_flex.pdbqt --log nag_prot_flex.log
# докинг с подвижными остатками белка
bash scr
# запуск скрипта для докинга с модифицированным лигандом
Использованные и полученные файлы:
Для проведения докинга была выбрана 5 структура, полученная в прошлом моделировании: seq5.pdb . Из нее была предварительно удалена молекула лиганда. Стоит также отметить, что в структуре были удалены N- и С- концевые участки, моделирование которых не было осуществлено из-за отсутствия данных в выравнивании.
Получена SMILES для сахарида NAG: nag.smi
Получена pdb-структура лиганда NAG: nag.pdb
Проведено конвертирование лиганда и белка в pdbqt-формат: nag.pdbqt , prot.pdbqt
Проведено фиксирование структуры белка с аминокислотами GLU189, ASN196, ARG135, ILE200 - им позволено быть подвижными: prot_rigid.pdbqt , prot_flex.pdbqt. Эти а.о. были выбраны, так как они использовались в качестве направляющих при моделировании структуры белка и имеют водородные связи с NAG в моделированном белке.
В структуре белка создан псевдоатом (pseudoatom в PyMol) для определения центра масс белка. Его координаты записаны в файл vina.cfg , для указания места поиска сайта для лиганда.
Проведен докинг для неподвижной структуры. Файл с информацией : nag_prot.log . Файлы с полученными моделями: nag_prot.pdbqt , prot.pdbqt
Проведен докинг для подвижной структуры. Файл с информацией : nag_prot_flex.log . Файлы с полученными моделями: nag_prot_flex.pdbqt , prot_rigid.pdbqt , prot_flex.pdbqt
Проведено изменение структур лиганда - добавление заместителей вместо СН3: OH, NH3, H, Ph. Файлы соответственно: oh.smi , n.smi , h.smi , ph.smi
Написан скрипт для осуществления докинга всех этих соединений в структуру белка (неподижную и с подвижными а.к.(те же, что и выше)): scr .
Получены результаты докинга этих лигандов.
Файлы с информацией:
для заместителя H: h_prot.log - неподвижный белок,
h_prot_flex.log - с подвижными остатками
для заместителя OH: oh_prot.log - неподвижный белок,
oh_prot_flex.log - с подвижными остатками
для заместителя NH3: n_prot.log - неподвижный белок,
n_prot_flex.log - с подвижными остатками
для заместителя Ph: ph_prot.log - неподвижный белок,
ph_prot_flex.log - с подвижными остатками
Файлы с моделями:
для заместителя H: h_prot.pdbqt , prot.pdbqt - неподвижный белок,
h_prot_flex.pdbqt , prot_rigid.pdbqt , prot_flex.pdbqt - с подвижными остатками
для заместителя OH: oh_prot.pdbqt , prot.pdbqt - неподвижный белок,
oh_prot_flex.pdbqt , prot_rigid.pdbqt , prot_flex.pdbqt - с подвижными остатками
для заместителя NH3: n_prot.pdbqt , prot.pdbqt - неподвижный белок,
n_prot_flex.pdbqt , prot_rigid.pdbqt , prot_flex.pdbqt - с подвижными остатками
для заместителя Ph: ph_prot.pdbqt , prot.pdbqt - неподвижный белок,
ph_prot_flex.pdbqt , prot_rigid.pdbqt , prot_flex.pdbqt - с подвижными остатками
Результаты и обсуждение
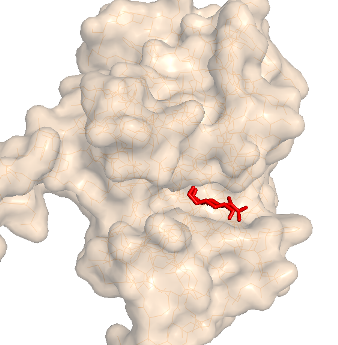
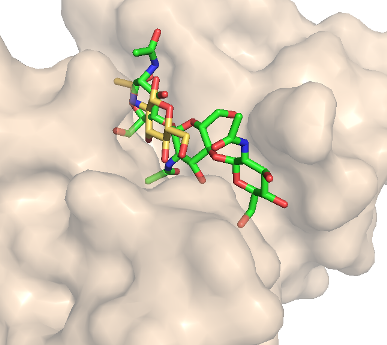
1. При обычном докинге получились структуры с лучшей энергией следующие:
| 1 |
|
|
affinity = -5.4 (kcal/mol) |
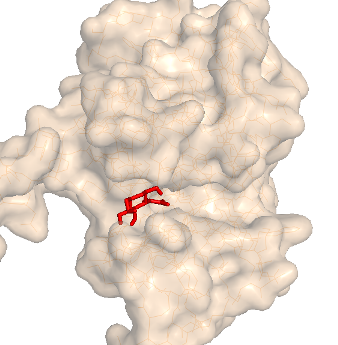
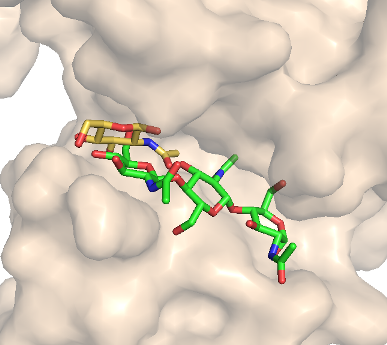
| 2 |
|
|
affinity = -5.2 (kcal/mol) |
| 3 |
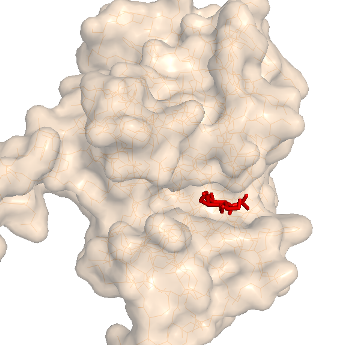
|
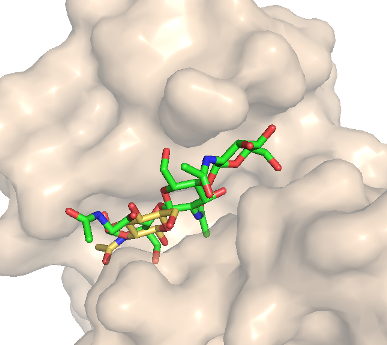
|
affinity = -5.2 (kcal/mol) |
Это подтверждается наложением структуры с лигандом, смоделированной ранее (зеленый во 2 столбике).
Однако, 2 модель менее точно отражает истинное положение лиганда - он почти на поверхности белка, а не в кармане.
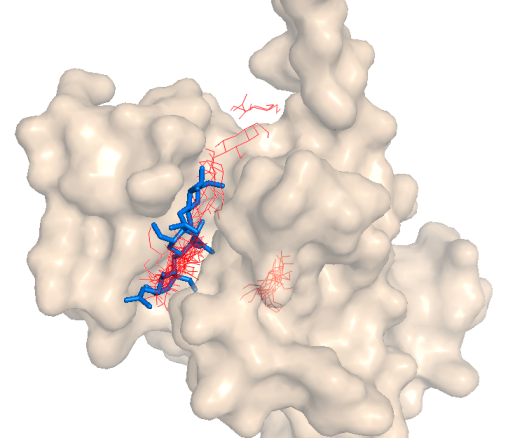
Также интересна группа моделей, в которой лиганд залез внутрь белка. Ну и есть модель, где лиганд просто лежит на поверхности.
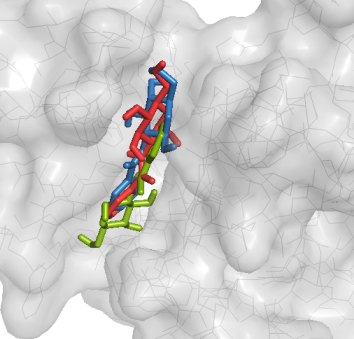
2. При докинге с подвижными остатками структуры с лучшей энергией следующие:
(зеленые а.к. - исходное положение; синие а.к. - смоделированное положение;
серый лиганд - лиганд в структуре, полученной на прошлом занятии; красный лиганд - смоделированное положение)
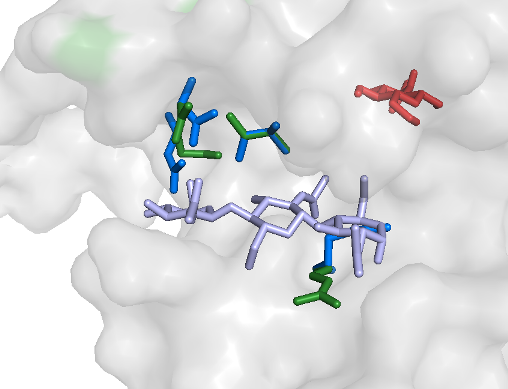
| 1 |
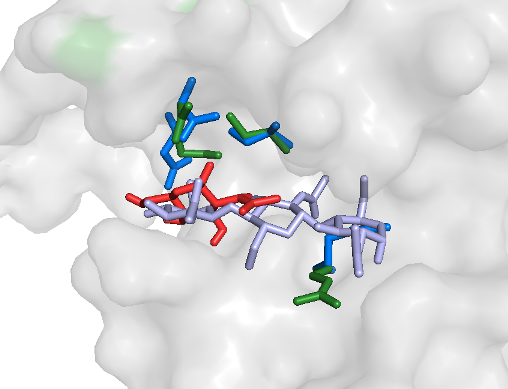
|
affinity = -6.0 (kcal/mol) |
| 2 |
|
affinity = -5.5 (kcal/mol) |
| 3 |
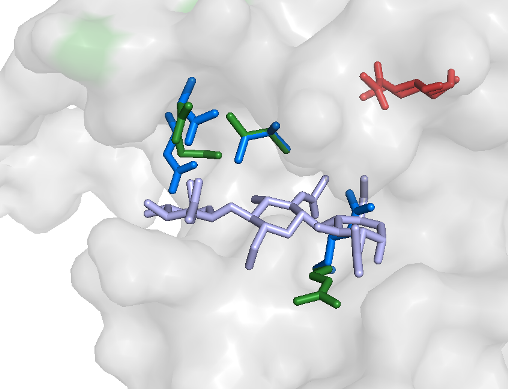
|
affinity = -5.3 (kcal/mol) |
Моделирование с подвижным белком более точно отраажает модель взаимодействия рука-перчатка.
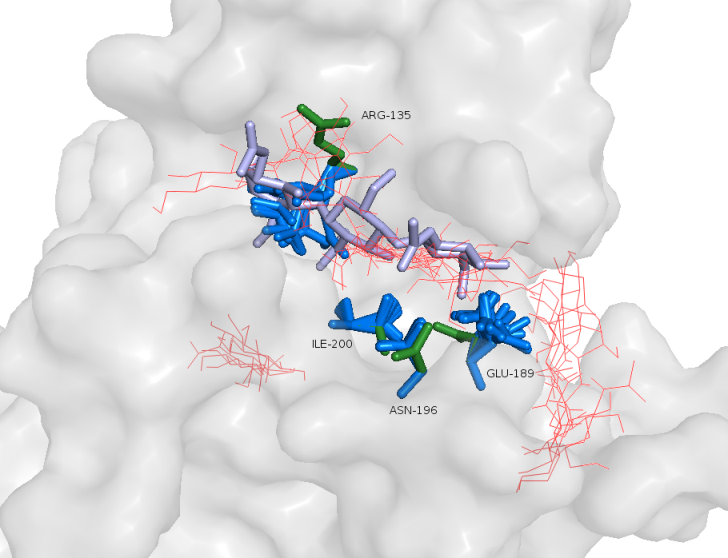
Так случилось, потому что в исходной структуре аргинин взаимодействовал с NDG-частью трисахарида.
Здесь же, он делает карман для моносахарида меньше, перекрывая его поперек, что, по-видимому, позволяет лиганду лучше удерживаться в кармане.
Возможно, поэтому энергия для 1 случая меньше, чем при обычном моделировании с неподвижным белком.
3. При докинге с измененными заместителями лиганда структуры с лучшей энергией следующие:
(красный - 1 модель, синий - 2 модель, зеленый - 3 модель, соответствующие значениям энергии)
| -OH | -NH3 | -H | -Ph | |
| Энергии 3 лучших структур (неподвижный белок) | 1. -5.3 2. -5.3 3. -5.2 |
1. -5.5 2. -5.3 3. -5.2 |
1. -5.7 2. -5.1 3. -5.0 |
1. -6.7 2. -6.6 3. -6.5 |
| Расположение 3 лучших структур (неподвижный белок) | 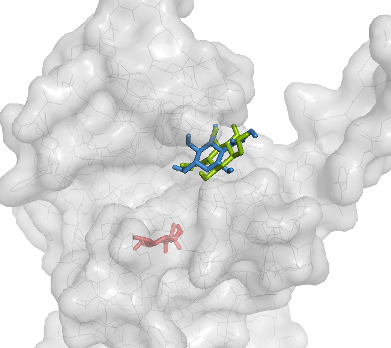 |
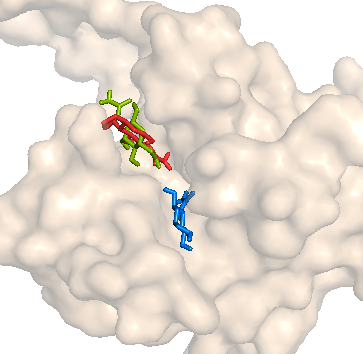 |
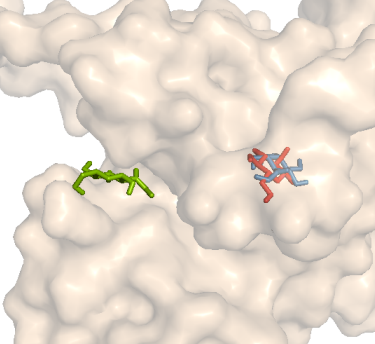 |
 |
| Все положения лиганда (неподвижный белок) | 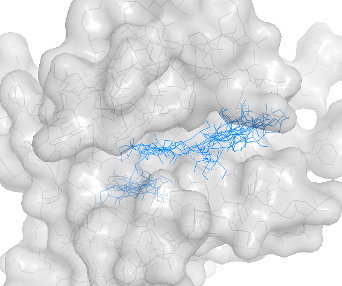 |
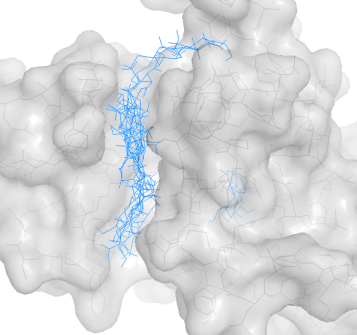 |
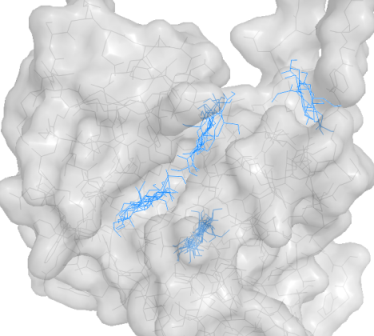 |
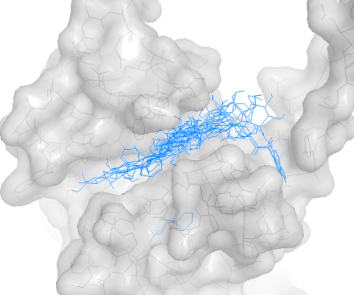 |
| Энергии 3 лучших структур (подвижный белок) | 1. -6.0 2. -5.5 3. -5.5 |
1. -6.2 2. -5.8 3. -5.7 |
1. -5.7 2. -5.4 3. -4.9 |
1. -6.5 2. -6.5 3. -6.1 |
| Расположение 3 лучших структур (подвижный белок) | 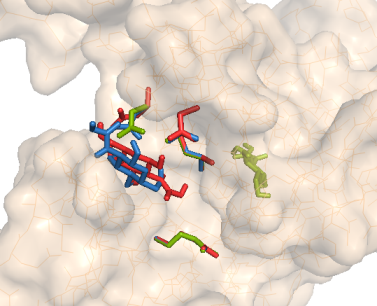 |
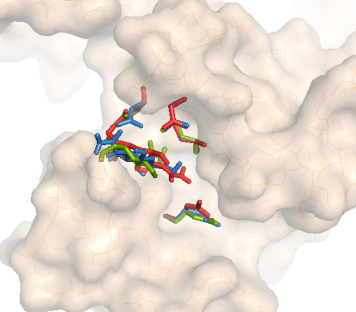 |
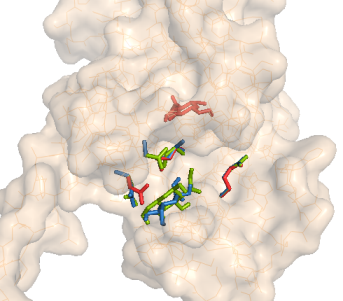 |
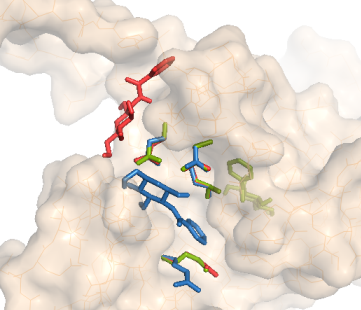 |
| Все положения лиганда (подвижный белок) | 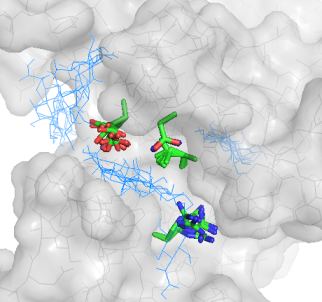 |
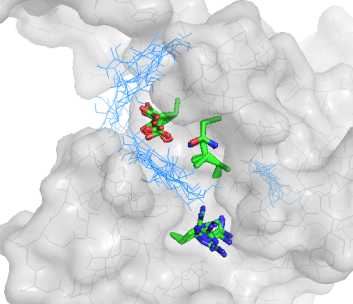 |
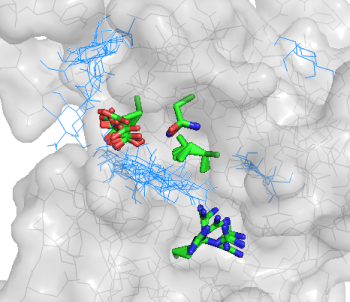 |
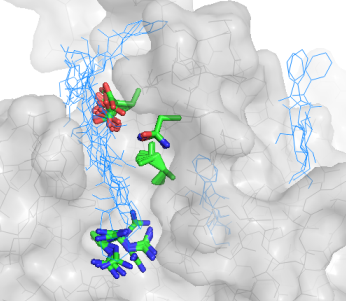 |
При неподвижном докинге:
Самые хорощие результаты получаются для NH3 и Ph-заместителей: все 3 лучшие структуры попадают в карман.
При этом лиганд с фениловым заместителем имеет наилучшую среди всех заместителей энергию.
Возможно, так получается из-за его кольца, значительно увеличивающего размер лиганда, в результате чего он более похож по размеру на исходный трисахарид.
При подвижном докинге:
Для -ОН: докинг с подвижным белком привел к лучшим резульататам: теперь 2 лучшие структуры лежат в кармане.
Положение аргинина не меняется в 3 лучших структурах и практически ен меняется во всех структурах.
Это еще раз подтверждает, что аргинин непосредственно не участвует в связывании моносахарида NAG.
Для -NH3: Аргинин снова практически не меняется во всех моделях.
При этом, как и при неподвижном докинге, все 3 лучшие модели содержат лиганд в нужном кармане.
Стоит заметить, однако, что теперь лиганд в них располагается практически одинаково.
Для H: использование подвижной структуры белка улучшило результат докинга. В трех лучших структурах наблюдени япро аргинин те же, энергия улучшается.
Для Ph: а вот лиганд с фенилом повел себя при подвижном докинге хуже. Получился большой разброс в положении лиганда.
Стоит отметить, что только одна модель из 3 лучших имеет правильное расположение.
Как Вы, наверное, уже догадались, этот практикум мне понравился больше всего.