Последовательности нуклеиновых кислот: банк EMBL
Задание 1. Знакомство со структурой банка EMBL посредством SRS
EMBL - Европейская Лаборатория Молекулярной Биологии (European Molecular Biology Laboratory),
EBI- Европейский Биоинформатический институт (European Bioinformatics Institute),
SRS - Система Поиска Последовательностей (Sequence Retrieval System).
Адрес: srs.ebi.ac.uk
a. Дата и количество записей последнего проиндексированного релиза EMBL .
Library Page -> EMBL (release) (клик)
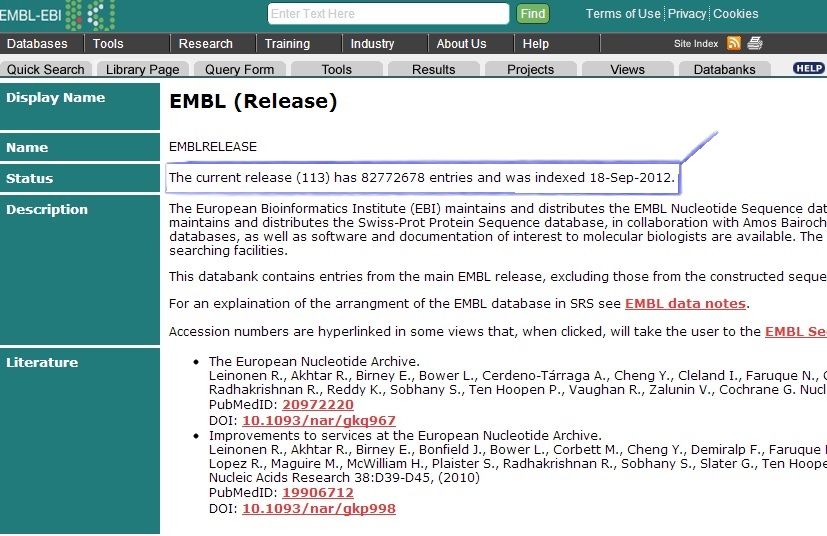
Количество записей: 82772678. Последняя индексация: 18 сенября 2012 года.
b. Список классов, количество записей в них.
Library Page -> EMBL (release) (клик)
-> Data Class
-> List values
Результат:
| CON: | Constructed sequence | - |
| EST: | Expressed Sequence Tag | - |
| GRV: | Genome Reviews | - |
| GSS: | Genome Survey Sequence | 34528104 |
| HTC: | High Throughput cDNA sequencing | 491770 |
| HTG: | High Throughput Genome sequencing | 152599 |
| MGA: | Mass Genome Annotation | - |
| PAT: | Patent | 24364832 |
| SET: | Project set (EMBL WGS Masters only) | - |
| STD: | Standard | 13920617 |
| STS: | Sequence Tagged Site | 1322570 |
| TSA: | Transcriptome Shotgun Assembly | 7992186 |
| WGS: | Whole Genome Shotgun | - |
c. Список разделов, количество записей в них.
Library Page -> EMBL (release) (клик)
-> Divisions
-> List values
Результат:
| ENV: | Environmental Samples | Образцы, выделенные из окружающей среды | 7762556 |
| FUN: | Fungi | Грибы | 2402829 |
| HUM: | Human | Человек | 11304977 |
| INV: | Invertebrates | Беспозвоночные | 7398340 |
| MAM: | Other Mammals | Другие млекопитающие | 6741732 |
| MUS: | Mus musculus | Мышь обыкновенная | 5163724 |
| PHG: | Bacteriophage | Бактериофаг | 8503 |
| PLN: | Plants | Растения | 20284404 |
| PRO: | Prokaryotes | Прокариоты | 1639517 |
| ROD: | Rodents | Грызуны | 1313761 |
| SYN: | Synthetic | Искусственные | 4045013 |
| TGN: | Transgenic | Трансгенные | 285306 |
| UNC: | Unclassified | Неклассифицированные | 8617170 |
VRL: | Viruses | Вирусы | 1358516 |
VRT: | Other Vertebrates | Другие черепные | 4446330 |
d*. Тенденции в заполнении банка данных.
Выбранные разделы: HUM, ENV и PRO. Класс: STD. Выбранные годы и месяцы для статистики: январь, ферваль, март 2011 и 2012 годов.
Я проводила поиск с помощью Extended Query Form. Использовала банк EMBL, а не EMBL (release), так как он не охватывает исследуемые промежутки времени.
Пример запроса (Класс: STD. Раздел: ENV, HUM или PRO. Дата создания: январь 2011 года, включая 1 февраля, что не принципиально):
(([embl-Class:std] & (([embl-Division:env] | [embl-Division:hum]) | [embl-Division:pro])) & [embl-DateCreated#20110101:20110201])
Результаты получила и оформила в виде документа exel с двумя графиками:
result.xlsx
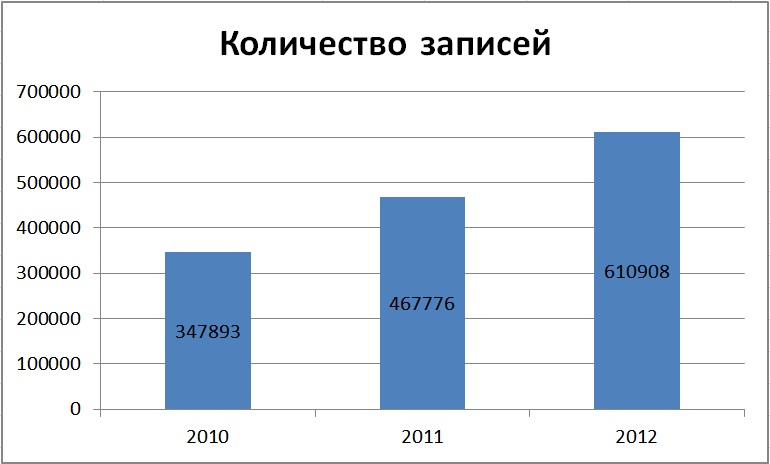
Первый отображает зависимость суммарного числа статей за период январь-март от года:
Второй позволяет оценить зависимость и от месяца внутри года:
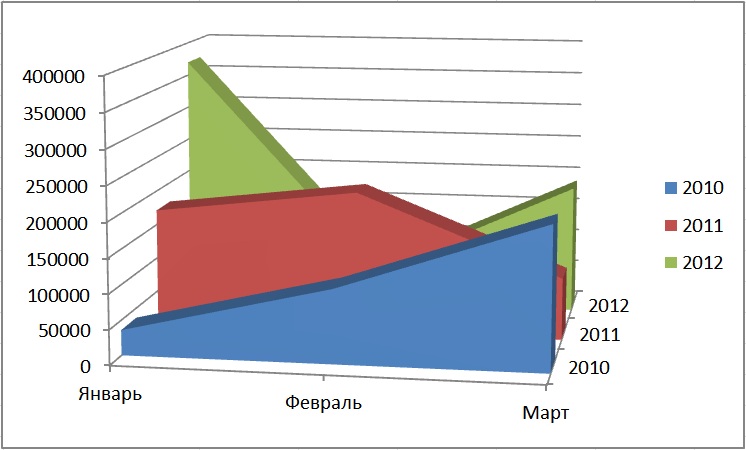
Замечаю, что от года к году статей добавляется всё больше. Тем не менее, зависимость
количества новых статей от времени не прямая для оценки по месяцам. Это видно из неожиданно
низкого числа статей в феврале 2012.
Задание 2. Описание гена в записи банка EMBL.
Следую по адресу P:\y11\Term_3\Block_2.
Нахожу файл
BA000025.embl
, открываю. Из описания (Homo sapiens genomic
DNA, chromosome 6p21.3, HLA Class I region.) делаю вывод,
что передо мною - последовательность региона HLA Class I - локуса p21.3 шестой хромосомы.
В FAR с помощью F7 нахожу название данного гена: HLA-C. Первое совпадение в тексте: 2333 строка из 41850.
Участок, из которого можно взять требуемую информацию: join(671525..671597,671728..671997,672248..672523,
673111..673386,673511..673630,674071..674103,
674211..674258,674423..674427)
Направление гена - прямое.
(Отсутствует упоминание "complement". Обратное направление гена было бы при записи "complement(join(.. .. ..))")
Число кодирующих участков: 8. Интронов: 7 (некодирующие участки).
Длина первого кодирующего учатка: 671525-671597+1=73
Длина последнего кодирующего участка: 674427-674423+1=5
Длина первого интрона: 671727-671598-1=128
Длина интрона перед последним кодирующим участком: 674422-674259-1=162
Задание 3. Нахождение белка по фрагменту гена.
Вырезаю самый длинный кодирующий участок в отдельный файл.
Для этого использую командную строку.
Самый длинный экзон - третий, в 276 нуктеотидов.
Вырезать его последовательность можно с помощью seqret.
Интересно, что его можно взять и
из отдельной записи о гене HLA-C
(я беру запись с названием HE995438, которая представляет
собою последовательность аллели HLA-C*05:01var),
и из заданной записи BA000025 (результат секвенирования локуса хромосомы).
############
# Кстати, достать запись embl о гене HLA-C можно, используя команду:
# entret embl:HE995438 HLA-C.embl
# Получаю файл HLA-C.embl.
#
#
############
Последовательность действий в командной строке для первого случая,
получение файла HLA-C.fasta:
C:\2012_2013\Block2>seqret embl:HE995438 HLA-C.fasta -sask
Read and write (return) sequences
Begin at position [start]: 740
End at position [end]: 1015
Reverse strand [N]:
Для второго случая, получение файла
HLA-C(1).fasta:
C:\2012_2013\Block2>seqret embl:BA000025 HLA-C(1).fasta -sask
Read and write (return) sequences
Begin at position [start]: 672248
End at position [end]: 672523
Reverse strand [N]:
Используя blastn (с пометкой "Align two or more sequence"), сравниваю последовательности:
######################
>lcl|60821
Length=276
Score = 457 bits (247), Expect = 2e-133
Identities = 265/274 (97%), Gaps = 0/274 (0%)
Strand=Plus/Plus
Query 1 GGTCTCACACCCTCCAGAGGATGTATGGCTGCGACCTGGGGCCCGACGGGCGCCTCCTCC 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1 GGTCTCACACCCTCCAGAGGATGTATGGCTGCGACCTGGGGCCCGACGGGCGCCTCCTCC 60
Query 61 GCGGGTATAACCAGTTCGCCTACGACGGCAAGGATTACATCGCCCTGAATGAGGACCTGC 120
|||||||| |||||| ||||||||||||||||||||||||||||||||| ||||||||||
Sbjct 61 GCGGGTATGACCAGTCCGCCTACGACGGCAAGGATTACATCGCCCTGAACGAGGACCTGC 120
Query 121 GCTCCTGGACCGCCGCGGACAAGGCGGCTCAGATCACCCAGCGCAAGTGGGAGGCGGCCC 180
||||||||||||||||||||| ||||||||||||||||||||||||| |||||||||||
Sbjct 121 GCTCCTGGACCGCCGCGGACACCGCGGCTCAGATCACCCAGCGCAAGTTGGAGGCGGCCC 180
Query 181 GTGAGGCGGAGCAGCGGAGAGCCTACCTGGAGGGCACGTGCGTGGAGTGGCTCCGCAGAT 240
||| ||||||||||| ||||||||||||||||||||||||||||||||||||||||||||
Sbjct 181 GTGCGGCGGAGCAGCTGAGAGCCTACCTGGAGGGCACGTGCGTGGAGTGGCTCCGCAGAT 240
Query 241 ACCTGGAGAACGGGAAGAAGACGCTGCAGCGCGC 274
||||||||||||||||| ||||||||||||||||
Sbjct 241 ACCTGGAGAACGGGAAGGAGACGCTGCAGCGCGC 274
######################
|
Последовательности незначитально отличаются, что может происходить по двум причинам:
незначительно отличающиеся генотипы, взятые для секвенирования; разные аллели одного гена.
Ради интереса и тренировки навыков, проведу дальнешие действия с обоими последовательностями.
Теперь нужно найти белки, содержащие участки, близкие к
считываемому с полученных последовательностей. Для этого использую
Translated
BLAST: blastx (BLASTX производит поиск по базам данных белков,
используя транслированную последовательность. Фактически, поиск можно производить с любой кодирующей последовательностью НК).
В поле Database указываю UniProtKB/Swiss-Prot(swissprot), хотя такого пункта нет в задании.
В обоих последовательностях обнаружен консервативный домен MHC_I:
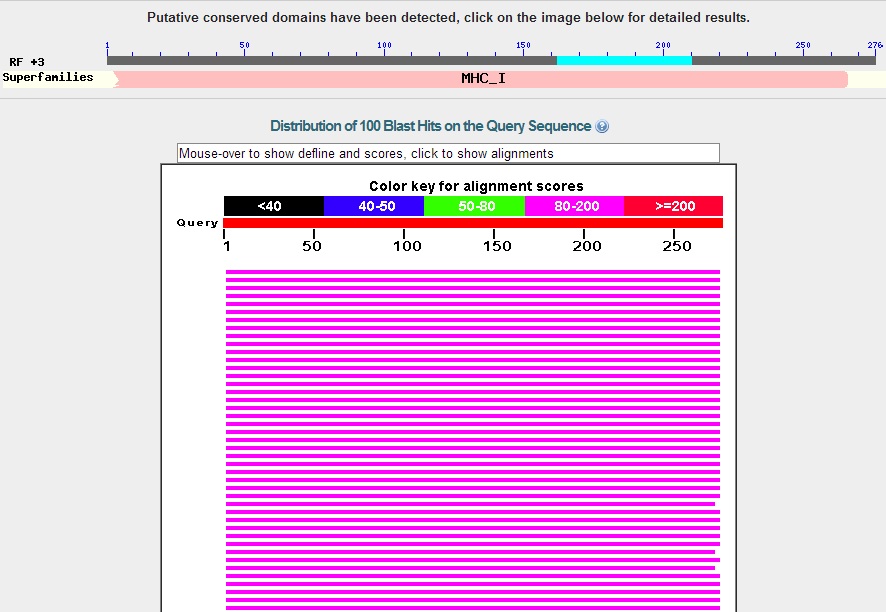
Получаем такие выравнивания с его последовательностью соответствено:
######################
Description PssmId Multi-dom E-value
MHC_I[pfam00129], Class I Histocompatibility antigen, domains alpha 1 and 2; 201024 no 1.09e-46
Class I Histocompatibility antigen, domains alpha 1 and 2;
Cd Length: 178 Bit Score: 149.44 E-value: 1.09e-46
10 20 30 40 50 60 70 80
....*....|....*....|....*....|....*....|....*....|....*....|....*....|....*....|
10844 RF +3 1 SHTLQRMYGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQRKLEAARAAEQLRAYLEGTCVEWLRRY 80
Cdd:pfam00129 91 SHTLQWMYGCDVGPDGRLLRGYEQFAYDGKDYIALNEDLRSWTAADPAAQITKRKWEAAGEAERERAYLEGECVEWLRRY 170
|
....*...
10844 RF +3 81 LENGKETL 88
Cdd:pfam00129 171 LENGKETL 178
|
######################
Description PssmId Multi-dom E-value
MHC_I[pfam00129], Class I Histocompatibility antigen, domains alpha 1 and 2; 201024 no 1.09e-46
Class I Histocompatibility antigen, domains alpha 1 and 2;
Cd Length: 178 Bit Score: 149.44 E-value: 1.09e-46
10 20 30 40 50 60 70 80
....*....|....*....|....*....|....*....|....*....|....*....|....*....|....*....|
13081 RF +3 1 SHTLQRMYGCDLGPDGRLLRGYNQFAYDGKDYIALNEDLRSWTAADKAAQITQRKWEAAREAEQRRAYLEGTCVEWLRRY 80
Cdd:pfam00129 91 SHTLQWMYGCDVGPDGRLLRGYEQFAYDGKDYIALNEDLRSWTAADPAAQITKRKWEAAGEAERERAYLEGECVEWLRRY 170
|
....*...
13081 RF +3 81 LENGKKTL 88
Cdd:pfam00129 171 LENGKETL 178
######################
|
Примечательно, что лучшие совпадения для "аллельноного"
и "локусного" вариантов восьмого экзона HLA-C не совпадают.
Для аллели HLA-C Cw*5 лучший вариант - он же,полное совпадение; для
HLA-C из файла - аллель Cw*12 (сортировка по E-value) и аллель Cw*7 (сортировка по Max Ident).
######################
sp|Q9TNN7.1|1C05_HUMAN RecName: Full=HLA class I histocompatibility antigen, Cw-5 alpha
chain; AltName: Full=MHC class I antigen Cw*5; Flags: Precursor
Length=366
GENE ID: 3106 HLA-B | major histocompatibility complex, class I, B
[Homo sapiens] (Over 100 PubMed links)
Score = 167 bits (423), Expect = 4e-50, Method: Compositional matrix adjust.
Identities = 91/91 (100%), Positives = 91/91 (100%), Gaps = 0/91 (0%)
Frame = +3
Query 3 SHTLQRMYGCDLGPDGRLLRGYNQFAYDGKDYIALNEDLRSWTAADKAAQITQRKWeaar 182
SHTLQRMYGCDLGPDGRLLRGYNQFAYDGKDYIALNEDLRSWTAADKAAQITQRKWEAAR
Sbjct 116 SHTLQRMYGCDLGPDGRLLRGYNQFAYDGKDYIALNEDLRSWTAADKAAQITQRKWEAAR 175
Query 183 eaeqrraYLEGTCVEWLRRYLENGKKTLQRA 275
EAEQRRAYLEGTCVEWLRRYLENGKKTLQRA
Sbjct 176 EAEQRRAYLEGTCVEWLRRYLENGKKTLQRA 206
######################
sp|P30508.2|1C12_HUMAN RecName: Full=HLA class I histocompatibility antigen, Cw-12 alpha
chain; AltName: Full=MHC class I antigen Cw*12; Flags:
Precursor
Length=366
GENE ID: 3107 HLA-C | major histocompatibility complex, class I, C
[Homo sapiens] (Over 100 PubMed links)
Score = 149 bits (376), Expect = 3e-43, Method: Compositional matrix adjust.
Identities = 88/91 (97%), Positives = 88/91 (97%), Gaps = 0/91 (0%)
Frame = +3
Query 3 SHTLQRMYGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQrkleaar 182
SHTLQRMYGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQRK EAAR
Sbjct 116 SHTLQRMYGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQRKWEAAR 175
Query 183 aaeqlrayleGTCVEWLRRYLENGKETLQRA 275
AEQ RAYLEGTCVEWLRRYLENGKETLQRA
Sbjct 176 EAEQWRAYLEGTCVEWLRRYLENGKETLQRA 206
######################
sp|P10321.3|1C07_HUMAN RecName: Full=HLA class I histocompatibility antigen, Cw-7 alpha
chain; AltName: Full=MHC class I antigen Cw*7; Flags: Precursor
Length=366
GENE ID: 3107 HLA-C | major histocompatibility complex, class I, C
[Homo sapiens] (Over 100 PubMed links)
Score = 147 bits (370), Expect = 3e-42, Method: Compositional matrix adjust.
Identities = 90/91 (99%), Positives = 90/91 (99%), Gaps = 0/91 (0%)
Frame = +3
Query 3 SHTLQRMYGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQrkleaar 182
SHTLQRM GCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQRKLEAAR
Sbjct 116 SHTLQRMSGCDLGPDGRLLRGYDQSAYDGKDYIALNEDLRSWTAADTAAQITQRKLEAAR 175
Query 183 aaeqlrayleGTCVEWLRRYLENGKETLQRA 275
AAEQLRAYLEGTCVEWLRRYLENGKETLQRA
Sbjct 176 AAEQLRAYLEGTCVEWLRRYLENGKETLQRA 206
######################
|
Считываемые c разных вариантов гена
белки действительно различаются.
Теперь немного о MHC-I.
"MHC" расшифровывется как
"Major histocompatibility complex" и переводится как "Главный комплекс гистосовместимости".
Белки этого комплекса располагаются на клеточной мембране лимфоцитов.
Они обеспечивают взаимодействия и передачу "информации" между клетками, участвующими в иммунитете организма.
MHC class II опосредуют иммунизацию - специфичность иммунитета - к антигену.
Молекулы этого класса представляют антигены из окружающего клетку пространства внутрь.
MHC class I опосредуют разрушение клеток-хозяев, имеющих этот антиген.
Молекулы этого класса присутствуют на поверхности почти всех типов клеток.
MHC class III представляют собой элементы системы комплемента.
HLA-C входит в класс MHC class I и
является клеточным рецептором с тяжелой цепью (? heavy chain receptor).
Гены MHC располагаются в шестой хромосоме и называются HLA (Human leukocyte antigen, Лейкоцитарный антиген человека).
Существует множество аллелей рецепторов MYC class I.
Они формируют гаплотип (совокупность аллелей в одном геноме)
на локусе p21.3 хромосомы 6.
Общего вывода из работы сделать пока нельзя. Например, остается неясным, где располагаются
в приведенном примере локуса другие аллели HLA-C.
Моему экзону соответствует участок 116-206 белка 1C05_HUMAN (Q9TNN7) для аллели Cw*5.
Задание 4. Ссылки из записи банка Swiss-Prot на
записи банка EMBL.
Требуется найти все ссылки на банк EMBL из записи
Swiss-Prot о белке P35160 (Swiss-Prot AC) RESA_BACSU (Swiss-Prot ID).
Для получения записи о белке использую:
entret sw:P35160
Получила файл
resa_bacsu.entret.
В нем нахожу раздел
DR (database cross-reference),
выбираю все AC записей о белке из EMBL. Их оказывается всего 2.
Затем следую в SRS. Выполняю последовательно действия:
Library page -> EMBL (галочка)
-> Standard Query Form (клик)
Поле "Accession number" -> "AL009126 | L09228" (соответствие)
Окно "Choose 1 or more fields" -> "ID", "Molecule", "Data class", "Sequence Length",
"Entry Creation Date", "Description" (последовательное выделение, удерживая Ctrl)
-> Save (клик)
Далее сохраняю в виде таблицы в формате txt, разделение табуляцией.
Формирую таблицу:
| ID записи |
Тип молекулы |
Класс данных |
Длина последовательности |
Дата внесения в банк |
Описание |
| AL009126 | genomic DNA | STD | 4215606 | 18-JUL-2002 | Bacillus subtilis subsp. subtilis str. 168 complete genome. |
| L09228 | genomic DNA | STD | 28206 | 17-NOV-1993 | Bacillus subtilis spoVA to serA region. |
Получаю записи с помощью команд:
entret embl:AL009126 AL009126.embl
entret embl:L09228 L09228.embl
Итак:
AL009126.embl
L09228.embl
Краткие выводы. AL009126 - результат секвенирования
Bacillus subtilis subsp. subtilis str. 168 complete genome (полный геном организма Bacillus subtilis, штамм 168).
L09228 - результат секвенирования Bacillus subtilis spoVA to serA region (того же организма,
региона со spoVA до serA).
Результат секвенирования региона гораздо "старше" секвенирования полного генома. В нем
Главная страница
Страница семестра
© Галицына Александра, 2012





