Предсказание парных выравниваний
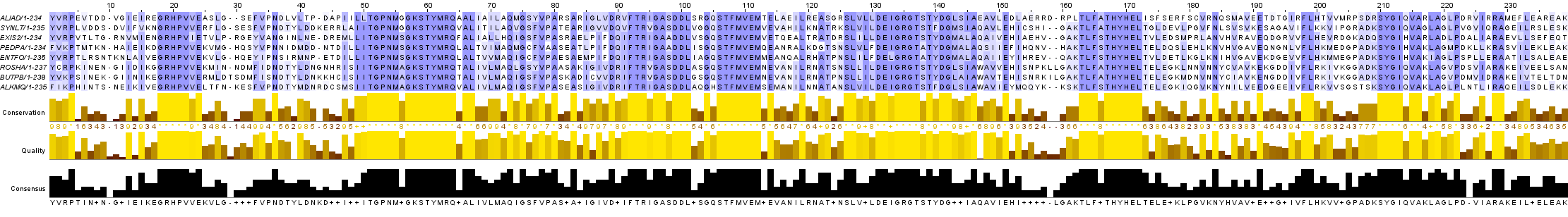
Было дано множественное выравнивание
Далее из этого множественного выравнивания было получено парное выравнивания последовательностей двух белков
( с индентификаторами ALIAD/1-234 и BUTPB/1-238).
При этом нужно было выбрать две наименее сходные последовательности. Это удалось сделать с помощью метода главных компонент.
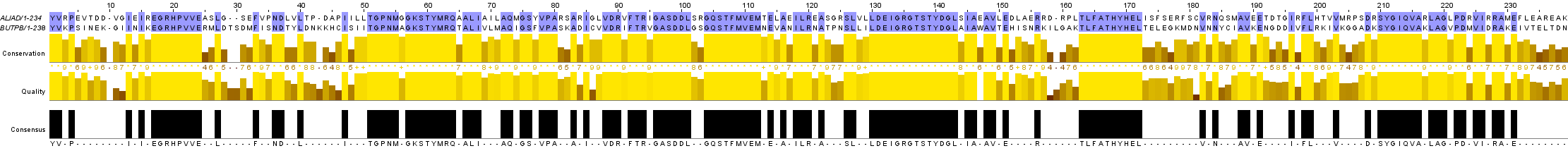
Сравнение выравниваний, сделанных программами water и needle с полученными из множественного.
Water строит локальное выравнивание, needle - глобальное
Алгоритм needle и water используют следующие параметры (некоторые установлены по умолчанию, другие вводятся самотоятельно):
- gapopen (штраф за открытие гэпа): 10
- gapextend (штраф за продолжение гэпа): 0.5
- datafile (матрица весов замен): EBLOSUM62 для белков, EDNAFULL для нуклеиновых кислот
- endweight (штраф за "концевые" гэпы)
- endopen (штраф за открытие "концевого" гэпа): 10
- endextend (штраф за продолжение "концевого" гэпа): 0.5
В задании нужно было построить 5 выравниваний выбранных двух последовательностей
(1. из множественного; 2. построенное needle; 3. построенное water; 4. needle с измененными параметрами 5. water с измененными параметрами)
Выбор изменениий параметров (штрафа за открsтие гэпа и штрафа за его длину) основывался на том, чтобы увидетьхоть какие-нибудь изменения.
Сравнение выравнивания, сделанного water и полученного из множественного.
В ходе сравнения выяснилось, что для "хорошего" выравнивания всех 4 последовтельностей
(без изменения внутреннего выравнивания) достаточно вставить один гэп.
Различия между выравниваниями наблюдаются на участке 9-12; 29-33 (см. рис)

Рис4. 2 различный участок 29-33
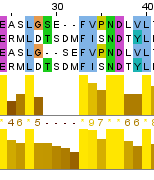
В needle выравнивание выглядет так же, но water "урезал" при добавлении дополнительного выравнивания исходное на 6 колонок.
Возможно, это связано с алгоритмом локального выравнивания.
Рис5. Замена конца последовательностей water'ого выравнивания на гэпы.

Мы посмотрели, как работает локальное и глобальное выравнивание на схожих последовательностях.
Теперь возмем два заведомо негомологичных белка с индентификаторами NP_148309.2 и YP_008011580

Таблица1. Сравнение выравниваний по численным хар-кам.
| Длина выравнивания (число колонок) | Число гэпов (сумма в двух последовательностях) | Длина гэпа | Число идентичных колонок | Процент идентичных колонок | Число сходных колонок | Процент сходных колонок | Процент идентичных и сходных колонок | |
| Парное выравнивание, полученное из множественного | 239 | 5 | 6 | 130 | 54.39 | 31 | 12.97 | 67.36 |
| Локальное выравнивание (water cо стандартными параметрами) | 232 | 3 | 4 | 130 | 56.03 | 32 | 13.79 | 69.82 |
| глобальное выравнивание (needle со стандартными параметрами) | 238 | 3 | 4 | 130 | 54.62 | 32 | 13.45 | 68.07 |
| Локальное выравнивание (water с измененными параметрами) | 257 | 12 | 23/28 | 139 | 54.08 | 34 | 13.23 | 67.31 |
| глобальное выравнивание (needle со измененные параметрами) | 244/245 | 3/5 | 6/11 | 133 | 54.29 | 35 | 14.29 | 68.58 |
| Локальное выравнивание негомологичных белков | 224 | 4/9 | 11/60 | 49 | 23 | 28 | 12.5 | 45.5 |
| глобальное выравнивание негомологичных белков | 243/502 | 4/10 | 11/75 | 51 | 10.6 | 28 | 5.6 | 16.2 |
В ходе выполнения заданий я пришла к следующим выводам:
- Для гомологичных последовательностей разница между глобальным и локальным выравнивание неочевидна.
- Однако для гомологичных последовательностей лучше использовать глобальное, чтобы не потерять ключевые фрагменты, которыми они отличаются.
- Для негомологичных последовательностей лучше использовать локальное выравнивание, так как гораздо важнее, чем они схожи (т.е. сразу убрать огромные негомологичные куски и работать с полезной информацией)
- Сравнивать множественное выравнивание и парное немножко сложно, так как их алгоритмы созданы для разных целей.
© 2014 Макарова Надежда