Чтение последовательностей по Сэнгеру.
Были даны два файла в формате .ab1, соответствующие прочтению прямой и обратнойцепочки анализируемой ДНК
Задание заключалось в нахождении и исправлении ошибок, допущенных программой при расшифровке детектирования сигнала на электрофорезе.
Для просмотра файлов была использована бесплатная версия программы Chromas lite. Сначала были получены две последовательности ( прямой и обратной, но перевернутой цепочек) в формате fasta. В Jalveiw они были выровнены вручную (см.рис1). Далее, основываясь на этом выравнивании, вручную "выравнивались" хроматограммы в Chromas. Из предварительного выравнивания видно, что 5'-конец последовательности лучше определен на перевернутой цепи, в то время как 3'-конец - на прямой.

Рис1. Выровненные вручную в Jalview прямая и обратная, но перевернутая последовательности ДНК. Нуклеотиды (A,T,G,C, N, где N - неизвестный нуклеотиды обозначен каждый своим цветом).
Определения нечитаемых концов.
Для начала определим границы читаемости хроматограммы с двух концов по координатам прямой последовательности.
Так как первые 17 нуклеотидов в прямой цепи не читаются, в обратной читаются с 11, но некоторых из их даже нет в прямой, нумерация пойдет в минус.
C 5'-конца хроматограмма не читается до -22 ( если считать по прямой) или до 11 (если по перевернутой обратной) (см. рис.2)
С 3'-конца хроматограмма не читается с 673 нуклеотида (по прямой цепи). (см. рис.3) Надо сказать, что такой выбор плохо читаемых концов был сделан в целях
верного определения последовательности. Из рис.2 и рис.3 видно, что качество хроматограммы позволяет определять последовательность и за пределами читаемости.
Но там закономерно появляются неоднозначные сложные места, которые я затрудняюсь точно определить.
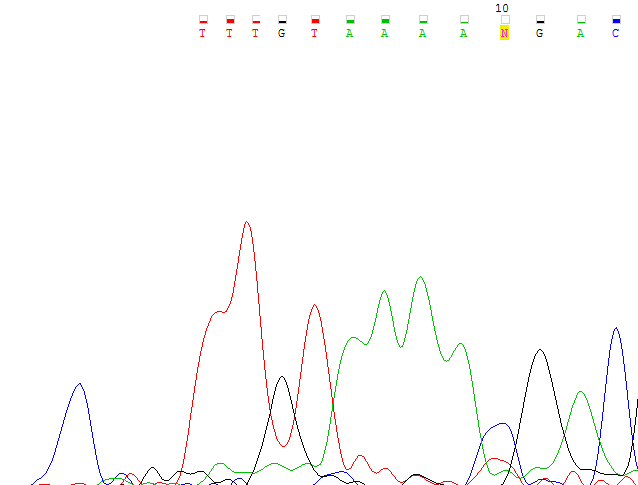
Рис.2 Плохо читаемый 5'-конец обратной последовательности (начиная с 11 (по обратной перевернутой последовательности) нуклеотида читается).
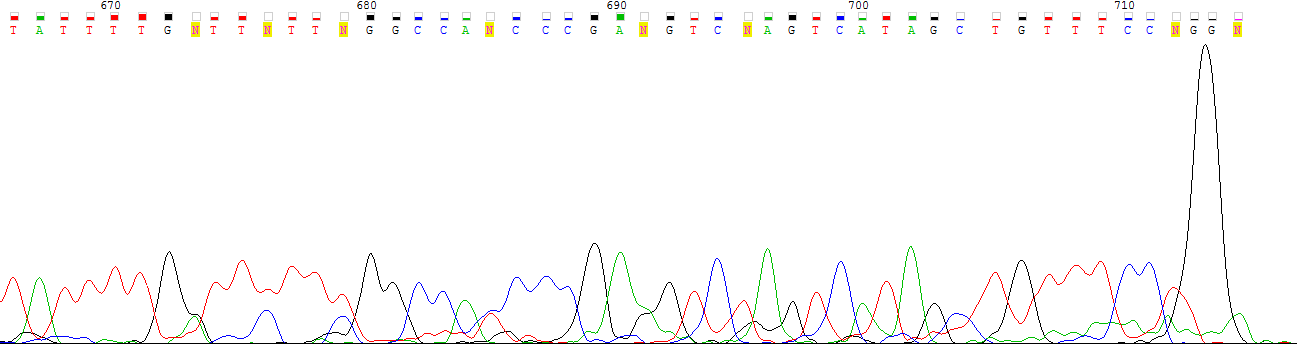
Рис.3 Плохо читаемый 3'-конец прямой последовательности. Плохо читается с 673 нуклеотида.
Визуальная характериcтика хроматограммы.
В данной хроматограмме уровень шума нисколько не мешает определять последовательность. В среднем сигнал превосходит шум в 5 раз.
Средняя сила сигнала и шума вдоль оси X равномерны, за исключением мест, где есть пятно краски и в плохо читаемых концах. (см. рис.4)
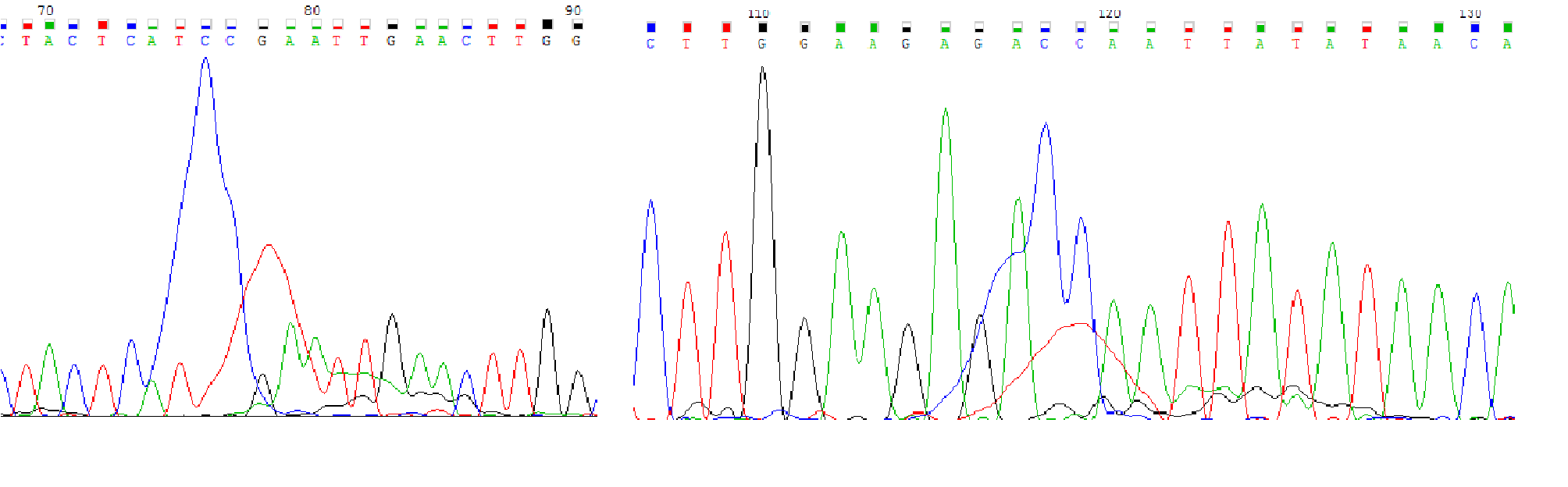
Рис.4 Пятна краски на хроматограмме. (Высота пиков очевидна разная: дело в маштабе; на первом пятне пик цитозина настолько велик, что другие програма маштабировала)
Различие между пиками очевидных ситуаций присутствует и отвечает некоторой закономерности ( а может и не закономерности) (см. таблицу 1) Исходя из наблюдений, можно сказать, что высота пика сигнала цитозина и тимина, а также разброс в их значениях в среднем почти одинаковы, аденина - чуть больше, а у гуанина, во-первых, уровень зарегистрированного сигнала выше и разность высот ощутемее. Возможно это связано с чувствительностью детектора. Еще мне показалось, что изображение пиков при детектировании нуклеотидов (когда какой-то нуклеотид в последовательности долго не появляется, а затем повторяется 3 раза (в частности), они образуют более менее одинаковый "профиль" (см. рис. 5 - пример с гуанином)
| Нуклеотид | Минимальная высота пика | Максимальная высота пика | Средний разброс высот пиков |
| С | 500 | 1300 | 650-1200 |
| Т | 577 | 1329 | 630-1250 |
| А | 612 | 1800 | 690-1300 |
| G | 420 | 2130 | 750-1500 |
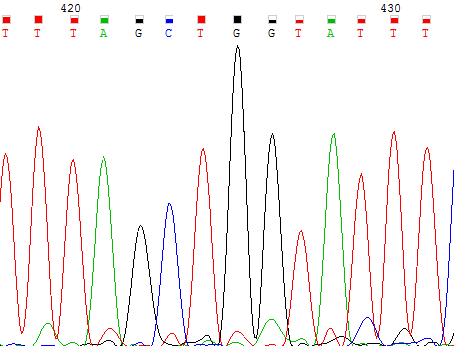
Рис.5 Характерный "профиль" пиков сигнала гуанина при близком расположении. (Сначала достаточно низкий пик, затем высокий, потом средний.
Правда, такая ситуация возникает, даже , когда один из пиков этом шум.
Надо сказать, что это хроматограмма хорошо сделана, так как уровень шума был достаточно низким. (см. рис 6) C 450 нуклеотида (по прямой) шум незначительно, но увеличивается.
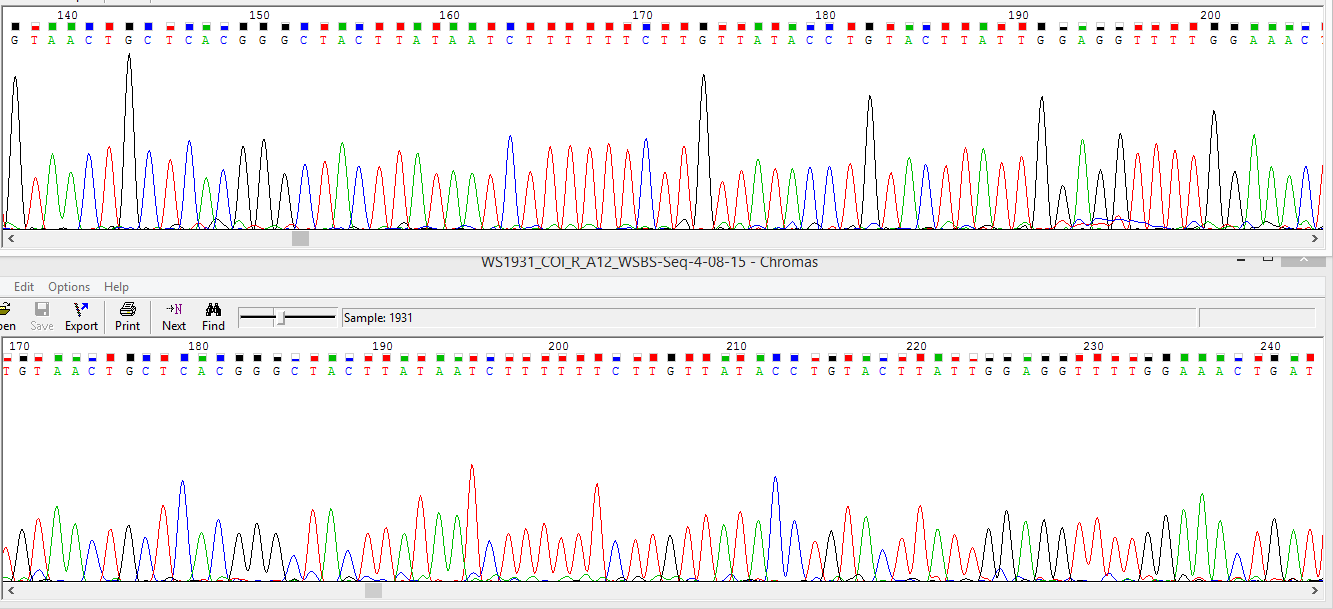
Рис.6 Изображение выровненных хороших хроматограмм с низким уровнем шума. Сверху - прямая цепь.
Нахождение проблемных мест в хроматограмме прямой цепи и проверка по обратной.
В основном, места, в которых возникали трудности при определении нуклеотида, оказывались на "концах" последовательностей. Что к сожалению, нельзя объяснить как полиморфизм, так как шум до этого места не был однородно низок.
Итак.-
Сложное место в 5' - конце: смотрела по обратной перевернутой цепи (см. рис.7). Нумерация по обратной цепи, так как в прямой этого участка нет, но он довольно хорошо читается. У 31, 33, и 40 нуклеотидов высота пика цитозина и тимина не сильно различаются (что отражено в таблице1 для всей последовательности, следовательно программа может легко принять одного за другого). Что касается 31 нуклеотида, то до него уровень шума был равномерно низок. Затем скачки шума повторились (33 нуклеотид и 40 нуклеотид) и не только цитозин-тимин, но и гуанин-аденин(37 нуклеотид). Надо заметить, что высота шумового пика тимина в 40 положении такая же, как и у сигнального в последующих двух. Предположение: в 31, 33, 37, 40 нуклеотидах просто загрязнение ДНК, но так как у нас нет возможности проверить по второй цепочке невозможно сделать однозначный вывод нуклеотиде в 31, 33 и 40 положении. (следовательно, в проверенную последовательность запишем, вместо N в 40 положении "Y" (обозначает С или T) - скорее всего это просто загрязнение, в 31 - тимин, в 33 - цитозин а в 37 - аденин (высота пика гуанина значительно ниже, чем соседние гуанины).
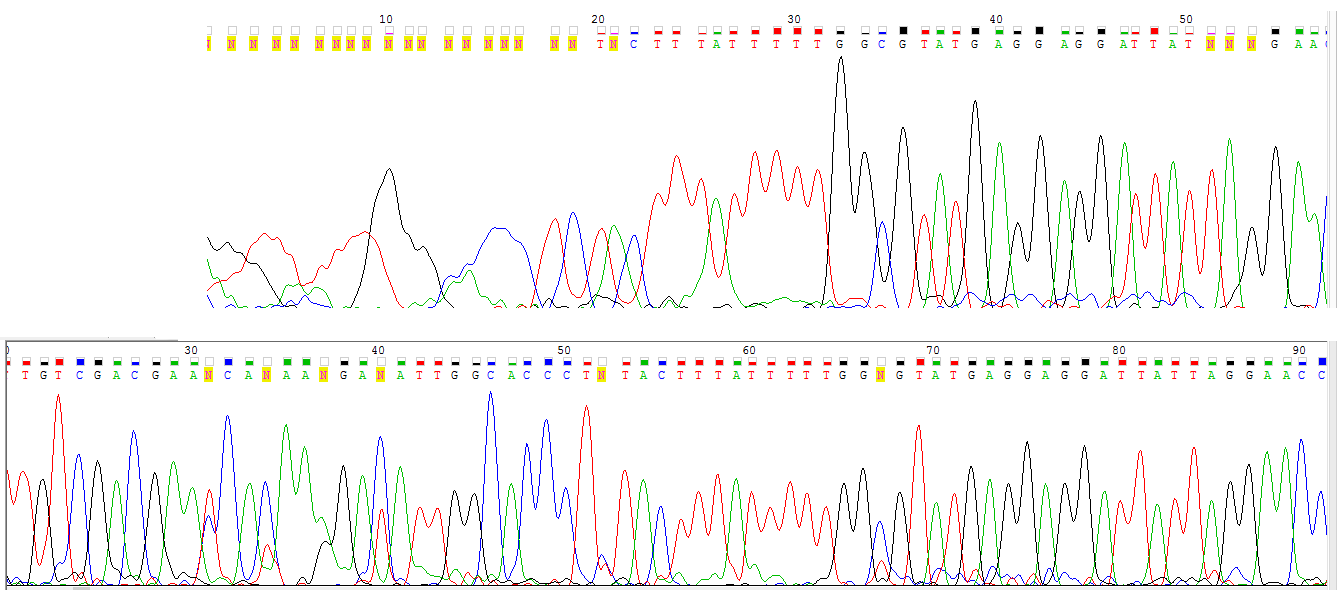
Рис.7 Изображение проблемного участка начала хроматограммы.
-
Теперь берем только прямую цепь. В 21 нуклеотиде не признала программа аденин (см.рис.7) Проверено по обратной цепи.(см. рис 7) Такого рода редактирование составляло большую часть работы. Программа почему-то иногда не распазнает очевидные сигнал.
-
Первое пятно (см. рис.8) определно программой верно. Проверено по обратной цепи. Второй пятно также правильно расшифровано.
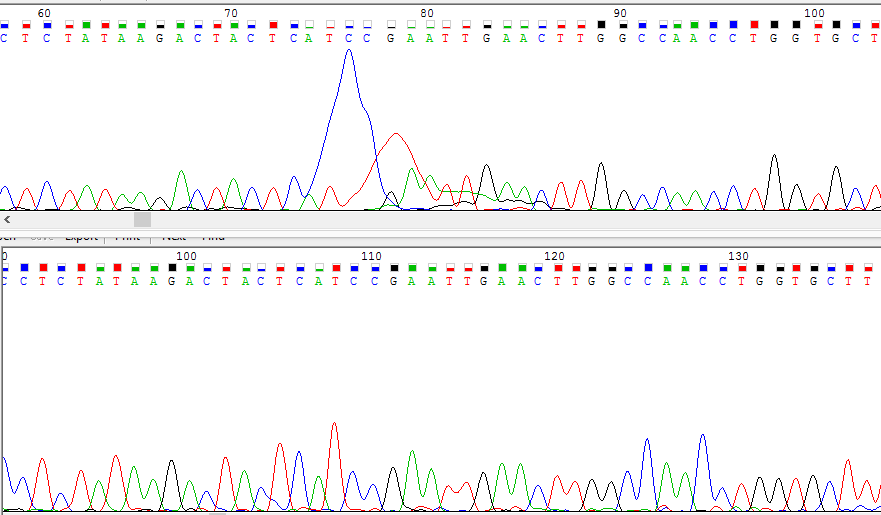
Рис.8 Изображение первого пятна, правильно расшифрованного программой, что ясно из сравнения прямой (находится сверху) и обратной цепей.
Далее хроматограмма прямой читается нормально. И трудностей не возникает. В обратной перевернутой цепи гораздо больше неопределенных мест не в начале, а в конце цепи. Во-первых, высота пиков неравномерна по последовательности. В следствии этого есть место с очень низким сигналом, к которому программа не чувствительна (см. рис.9). Но в сравнении с прямой цепью все определяется.
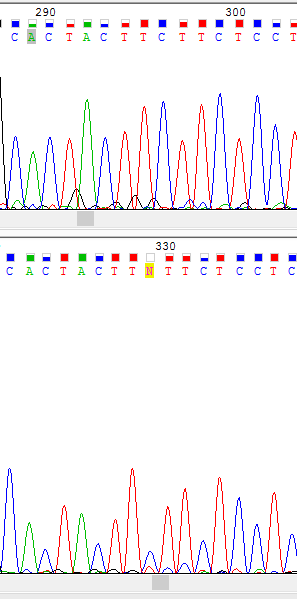
Рис.9 Изображение нерасшифрованного нуклеотида в положении 329. Но в сравнении с прямой цепью все определяется.
Во-вторых, есть два пятна краски, что помешало программе определить несколько нуклеотидов (см. рис.10)
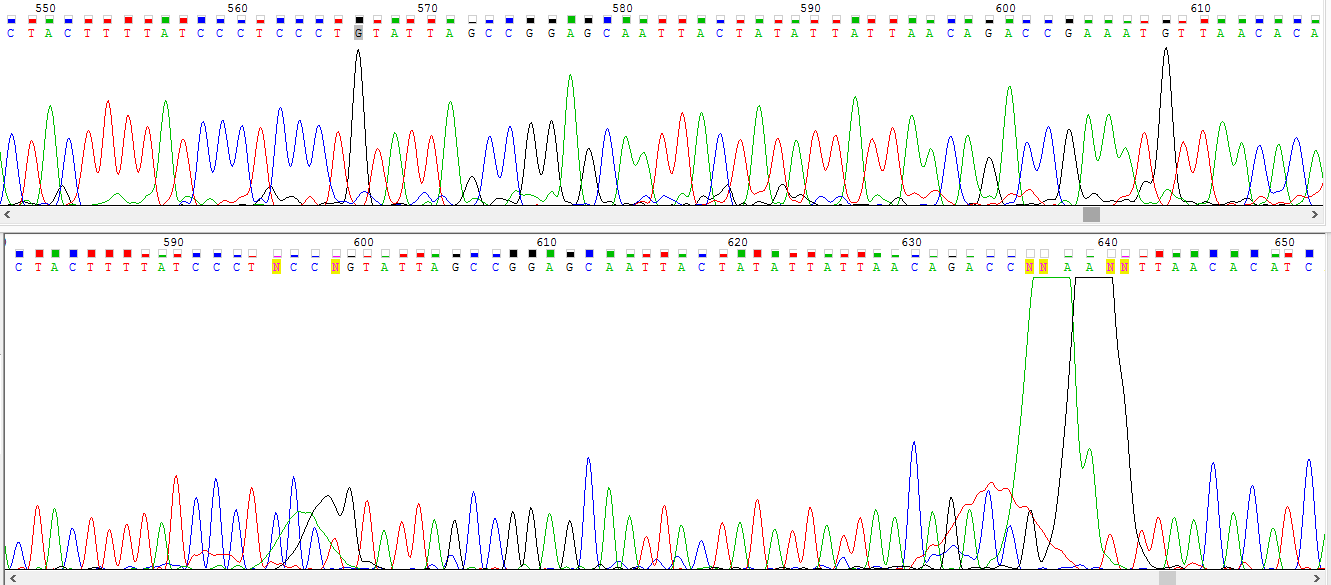
Рис.10 Изображение проблем с расшифровкой нуклеотидов в обратной перевернутой цепи.
Пример нечитаемой хроматограммы.
Чтобы понять, насколько можно доверять предыдущей хроматограмме, рассмотрим пример плохого представления детектирования сигнала. Здесь не читается ниодин пик. Программа пыталась сначала определять сигналы, но уже с 6-ого остановилась. Затем после двух пиков идет "пропасть". (см. рис.2) Интересно, почему программа не остановилась именно на этом участке, а прервала работу чуть раньше. По всей длине хроматограммы шум и сигнал на одном уровне, а иногда их вообще почти нет. Чтобы определить в чем причина плохой хроматографии (делеция нуклеотида, две разных ДНК), я пыталась найти какие-нибудь закономерности в последовательности пиков. Мне показалось, что в разных местах последовательности есть участки, где "профиль" повторяется через четыре нуклеотида. Также если сранить с предыдущей хроматограммой, то начало (5' конец) резко отличается по ширине пиков: в хорошей начало расплывчатое, пики сливаются образуя широкие "горы", а в плохой пики сразу различимы. Но это только наблюдения. Никаких выводов из них не следует. Предположение о причине получения: в пробе было несколько ДНК.
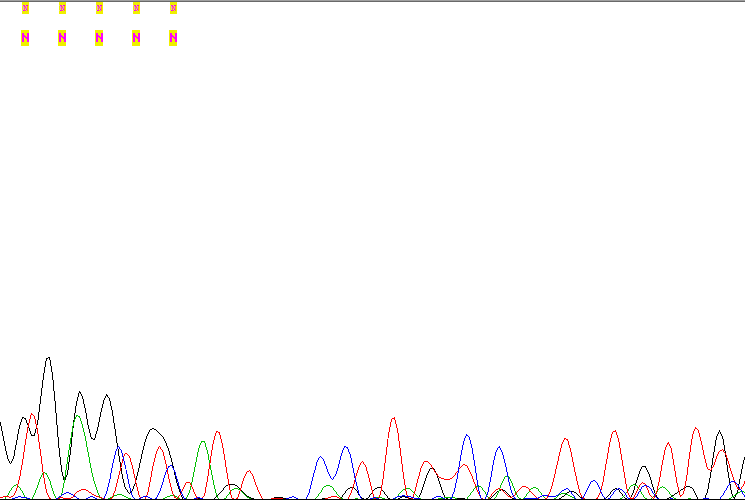
Рис.11 Изображение 5' конца неудачной хроматограммы.
В результате анализа была получена следующая "чистая" последовательность.
Также было построено выравнивание
прямой последовательности и комплементарной к обратной с измененными нуклеотидами.
© 2014 Макарова Надежда