Предсказание генов прокариот
Для анализа был взят геном Anaplasma marginale str. Florida . Это внутриклеточная бактерия крупного рогатого скота, переносится клещами.
Из семейства Rickettsiales. Не найдено вакцины. Полная таксономия представлена на рис.1. Содержит 1 кольцевую хромосому ( АС GenBank: NC_012026).

Рис. 1 Информация о таксономии Anaplasma marginale str. Florida, взятая с сайта NCBI.
Далее была скачана с сайта NCBI таблица с белок-кодирующими генами, аннотированными GenBank. C помощью сервиса RAST (Rapid Annotations using Subsystems Technology) была получена следующая таблица аннотированных генов. Задание заключалось в том, чтобы сравнить две таблицы аннотированных белок-кодирующих генов.
В таблице GenBank аннотировано 942 белок-кодирующих гена, в аннотациях RAST - 1193. Необходимо упомянуть, что в таблице из GenBank
были найдены гены одинаковых белков (см. таблицу 1)
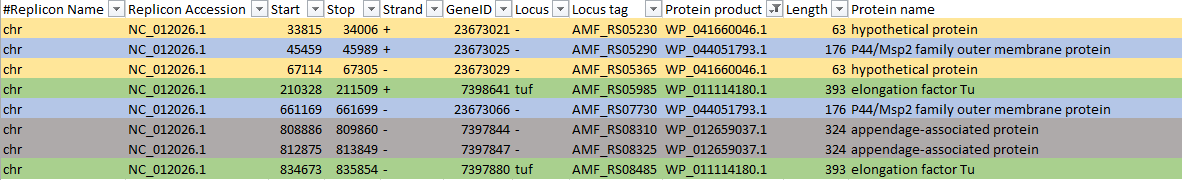
Таблица 1. Гены, кодирующие одни те же белки, но имеющие различное положение. Каждая пара отмечена своим цветом.
Для начала в таблице, выданной RAST, координаты начала и конца генов на комплементарной цепи были поменены местами. Далее с помощью скрипта были сравнены две таблицы аннотаций (каждая была преобразована в словарь структуры: название белка - [start, stop, strand]). Результаты представлены в таблице2.
| Число генов, у которых старт- и стоп- кодоны имеют одинаковые координаты в двух таблицах | Число генов с одинаковыми стоп-кодонами | Число генов, аннотированных RAST и неаннотированных GenBank | Число генов, аннотированных GenBank и неаннотированных RAST |
| 591 | 336 | 226 | 15 |
| файл с таблицей | файл с таблицей | файл с таблицей | файл с таблицей |
Таблица 2. Результат сравнения двух таблиц аннотаций генов для Anaplasma marginale str. Florida
Несовпадение аннотаций
Аннотация RAST в любом из трех примеров проверялась с помощью Blastx
- Сначала я сравнивала аннотации генов белков с определенными функциями (т.е. без hypothetical protein), у которых совпадал стоп кодон, но не совпадал старт. Но каждый раз оказывалось, что функция одинакова, но названия даны такие, что трудно понять, что они определяют одно и тоже. Часто встречалось, что RAST предсказывал ген, который немного больше аннотированного в GenBank, из-за того, что старт кодон был выбран по-другому. Тут, мне кажется, уместно сказать о проблеме определения N-конца белка: строя множественное выравнивание белков - гомологов, отобранных из базы данных RefSeq , я столкнулась с тем, что последовательности некоторых из них очевидно "обрублены" с N-конца, который , вероятно, был неправильно определен из-за выбора старт-кодона.
- Один не совсем понятный мне случай попался при сравнении следующих аннотаций (см. рис. 2):

Рис. 2 Информация о гене, кодирующем белок Integrase - локус (GenBank): AMF_RS08775
Интеграза — фермент, катализирующий интеграцию (включение) ДНК вируса в хромосому клетки-хозяина. Доменная структура белка, наложенная на ген показана на рис.3
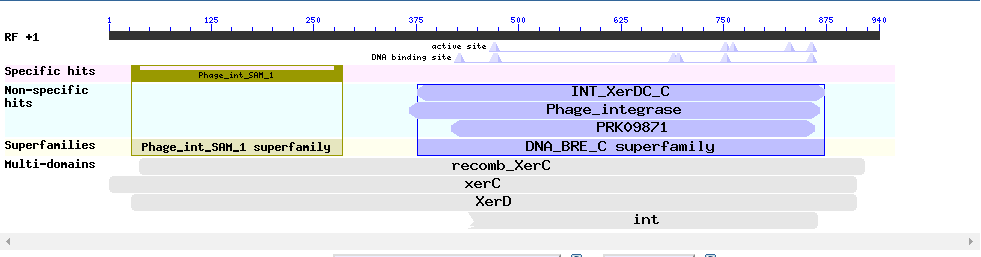
Рис. 3 Доменная структура белка Integrase, наложенная на нуклеотидную последовательность
Мне показалось странным, что в этот раз последовательность из GenBank была длиннее, последовательность RAST (см. рис. 4). Посмотрев на структуру нуклеотидной последовательности из GenBank, было определено, что в первых 12 нуклеотидах нет никаких регуляторных участков, что объяснило бы решение RAST не включать этот участок. Поэтому остается неясно, почему RAST не включил эту последовательность. При проверке по Blastx, в числе лучших находок присутствовали только Integrase. Что позволяет сделать вывод, что RAST ошибся.
Рис. 4 Выравнивание начального участка двух аннотированных последовательностей белков (RAST - верхняя, GenBank - нижняя). В красном прямоугольнике выделены аминокислоты, которые RAST не предсказал, но которые присутствуют в аннотации GenBank.
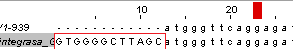
Рис. 5 Выравнивание начального участка двух аннотированных последовательностей гена (RAST - верхняя, GenBank - нижняя). В красном прямоугольнике выделены нуклеотиды, не включенные RAST при аннотации гена. Еще раз взглянув на графическое изображение структуры гена (рис. 6), становится понятно, почему аннотации генов разные. RAST дал такое название так, как определил старт-кодон гена там, где начинается регион xerC.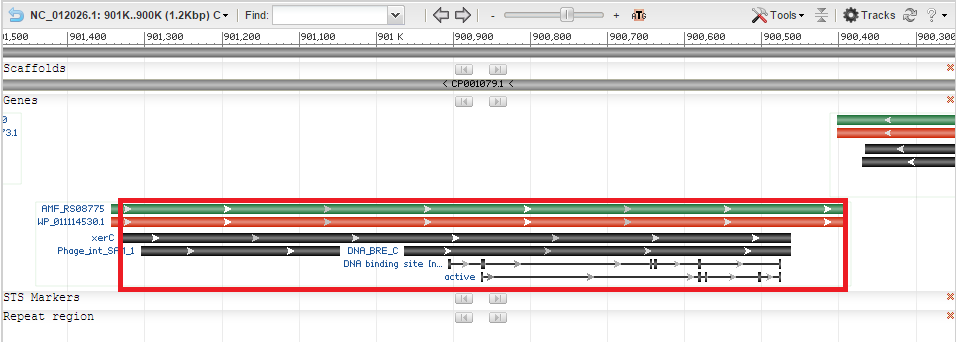
Рис. 6 Схема расположения участков гена Integrase. Красный прямоугольник выделяет границы гена, аннотированоого RAST. - Очень много раз попадалось, что гены, аннотированные в GenBank как hypothetical protein, в RAST определялись, как выполняющие определенные функции.
Я взяла гены, имеющие одинаковые координаты в двух аннотациях (см. рис 7)

Рис. 7 Информация о гене, находящемся в локусе (GenBank) AMF_RS09450. Желтым выделена функция от RAST, голубым - от GenBank.
Результаты поиска по blastx (см. Рис. 8) показали, что функцию данного белка не удастся установить. Ген содержит COG3898 (неохарактеризованных мембранных белков, функция которых неизвестна). Функция белка, закодированного на этом гене согласно RAST - Membrane-bound protoheme IX biogenesis protein, HemY не является достоверной.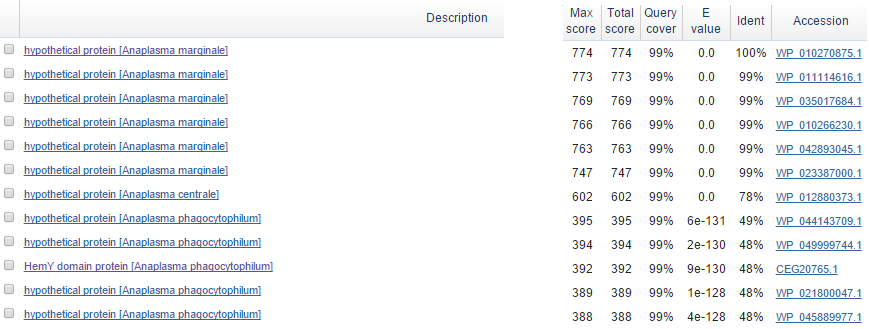
Рис. 8 Список лучших находок, выданных blastx.
- Так же было интересно посмотреть на белки, которые не нашел RAST, но которые есть в аннотациях GenBank. Из 15 найденных белков только для двух известна функция.
Первый был выбран для анализа. Информацию о белке представлена на рис. 9. В выдаче RAST в этом участке генома не аннотировано ничего.

Рис. 9 Информация о гене, аннотированном GenBank, но не предсказанным RAST
Сначала был сделан поиск гомологов по blastx. Ни одной находки из бактерий. Максимальное покрытие query 30%. Протранслировав данную последовательность в шести рамках и сравнив ее с белковой базой данной, BLAST нашел подходящими лишь эукариотические белки неизвестной функции. (см. рис. 11). Посмотрев на название query, все стало ясно: GI - 7398245 присвоен какой-то мРНК клонированной ДНК лягушки. Это объясняет то, что дальнейший поиск не давал никаких достоверных результатов. Видимо в базе данных GenBank ошибка: в базе Nucleotide одному и тому же GI соответствуют разные последовательности (лягушки Xenopus laevis - 2001 года, бактерии Anaplasma marginale str. Florida - 15.08.2015) Информация о гене Anaplasma marginale str. Florida с GI: 7398245 представлена на рис. 10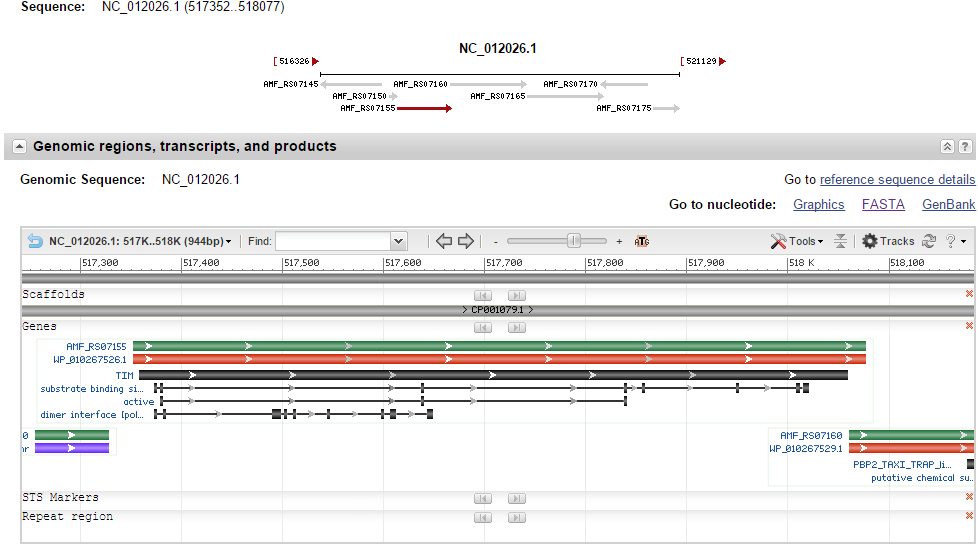
Рис. 10 Информация о структуре гена, аннотированного GenBank, но не предсказанного RAST
Прогнав последовательность, взятую уже из аннотированного генома, по BLASTX, существование гена Triosephosphate isomerase было доказано. Интересно, коррелирует ли отсутствие предсказания RAST и сбой в информации GenBank? Для решения этого вопроса я выбрала три GI белков неаннотированных в RAST из разных частей генома и просто искала их последовательности по Nucleotide, так как в прошлый раз именно так было обнаружено что GI присвоен другой последовательности. Оказалось что все три последовательности не находятся по GI однозначно. 23673089, 23673024, 7398662 - все эти гены не аннотированы RAST, и для всех есть другие последовательности с таким же GI.
© 2014 Макарова Надежда