Предсказание генов эукариот
Предсказание генов X5 с помощью AUGUSTUS
Выбор контига
Был выдан файл c контигами сборки генома неизвестного организма - X5.fasta. С помощью программы infoseq пакета EMBOSS была получена информация о длинах контигов (рекомендуемые маштабы: 20кb - 100кb). Я выбрала следующий контиг: scaffold_308 длиной 30351 пара нуклеотидов.
Выбор родственника из списка организмов, для которых уже произведено обучение сервиса.
Для начала был запущен BLASTX: поиск по транслируемой в шести рамках нуклеотидной последовательности против банка белков.
Из-за экзонно-интронной струтуры гена эукариот не имеет смысла искать схожие нуклеотидные последовательности, так как это вовсе не означает, наличия схожих генов.
Поэтому был сделан поиск по белкам.
Первый запуск BlASTX показал, что транслируемые последовательности нашего контига схожи с последовательностями белков грибов (царство Fungi),
так как их больше всего было в выдаче.
Чтобы посмотреть по-конкретнее, я ограничела поиск гомологов внутри Fungi.
Результат выдачи представлен на Рисунке 1.
Самые лучшие находки в большинстве принадлежат организмам из порядка Mucorales (на рисунке 1 подчеркнуты синим).
Остальные принадлежат различным таксонам и представлены в единственном экземпляре (Chytridiomycota, Mortierellomycotina, Ascomycota
(в выдаче их тоже много, но у них наблюдается резкое падение идентичности) . Присутствуют 3 находки из рода Rhizopus -
на рисунке только один из 7 и при увеличении числа последовательностей в алгоритме их количество возрастает.
Это стало причиной выбора организма Phizopus oryzae из списка Augustus, для которого уже проведено обучение.
(Программе нужно хотя бы примерно знать частоты встречаемости кодонов в различных генах,
опираясь на это и еще несколько других параметров программа будет определять положение генов в неизвестном, но родственном геноме).
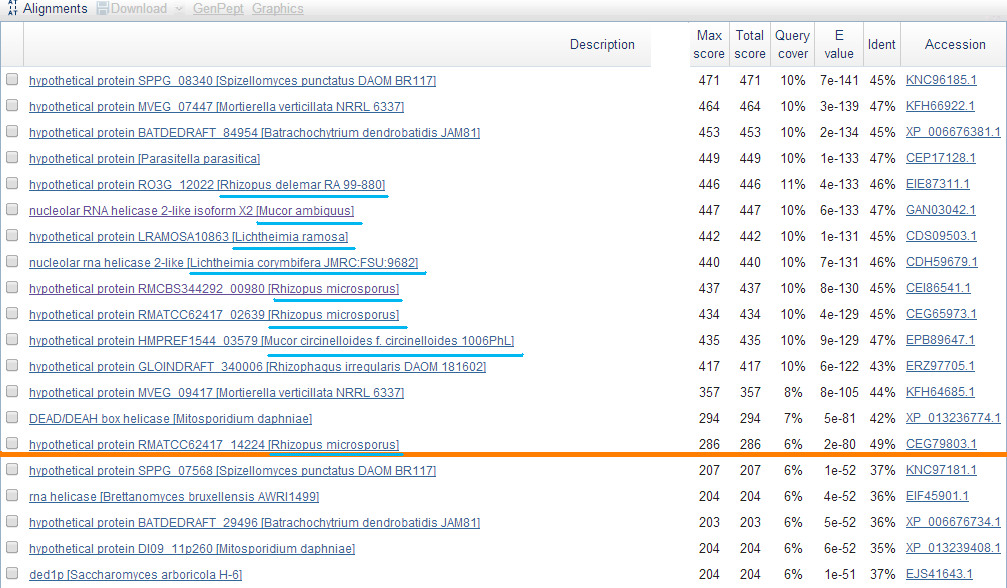
Рисунок 1. Список находок BlASTX. Из рисунка видно, что как минимум один белок точно закодирован на этом участке генома. Синим подчеркнуты представители порядка
Mucorales. Оранжевым выделен порого самой высокой идентичности.
Запуск и результаты программы AUGUSTUS
страница с результатомПосле указания Project identifier для организма rhizopus_oryzae , была запущена программа AUGUSTUS со стандартными параметрами (не предсказывать некодирующие области (Untranslated region - UTR), предсказывать гены на обеих цепях, без альтернативных транскриптов, предсказывать все возможные гены (в том числе и частичные послеодвательности), учитывать положение генов на комплементарной цепи)
В zip-архиве были получены следующие файлы:
- augustus.aa - содержит аминокислотные последовательности белков, которые закодированы в предсказанных генах.
- augustus.cdsexons - содержит нуклеотидные кодирующие последовательности экзонов.
- augustus.codingseq - содержит нуклеотидные кодирующие последовательности предсказанных генов.
- augustus.gbrowse - содержит таблицу с информацией о названии последовательности участка генома, об источнике предсказаний (программа), о типе последовательности (feature), которую выбрали как предсказанную о координатах начала и конца данной последовательности, о вероятности, о цепи, рамке считывания и индентификаторе последовательности. Отслежена согласно геномному браузеру GBrowse
- augustus.gff - помимо таблицы с идентичными колонками, что и в augustus.gbrowse (пока не проверено по браузеру в поле features не mRNA а transcript) содержит последовательности: нуклеотидную кодирующую и аминокислотную.
- augustus.gtf - содержит таблицу с информацией о предсказанных генах. колонки такие же как и в augustus.gff. Не содержит последовательностей.
Проверка предсказания с помощью BLASTp
- g11 Решила выбрать самый крупный белок. больше вероятность найти чего-нибудь. Но не особо вышло. E-value находок довольно плохой (min 3e-04),
процент идентичности не доходит и до 50. Однако как видно из рисунка 2 все-таки один домен нашелся. Ph-like домен.
Но так как среди находок нет представителей Mucorales и показатели довольно плохие, я считаю что гена такого белка у X5 нет.
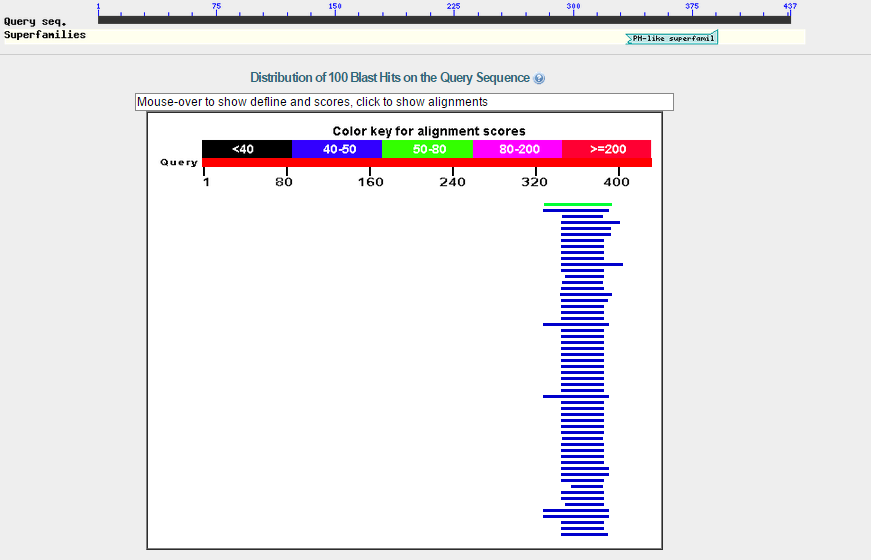
Рисунок 2 Графическое изображение находок Blastp для g11 - g10 В данном случае есть находки, которые покрывают query больше чем на 90%. Однако процент идентичности оставляет желать лучшего (небольше 40%).
Это значит, что можно установить функцию белка и обвинить Augustus в ошибке определения кодирующей последовательности. Итак, это точно протеин киназа.
Во всех выравниваниях хорошо ложится консерватеивный домен. Однако в целом выравнивания какие-то неубедительные. Еще нужно сказать, что по таксономии два претендента:
больше всего находок у Mucorales, однако покрытие у них 60 %. Самые первые и лучшие находки у Rozella allomycis дают 90%. Вывод: это ген протеин киназы, в котором Augustus
неверно определил экзон-интронную структуру (есть места, где он экзон определил как интрон и наоборот).
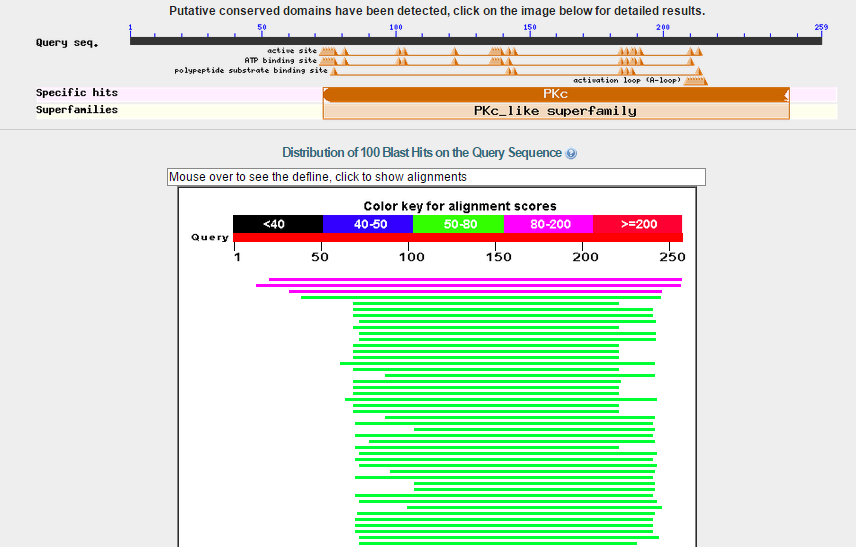
Рисунок 3 Графическое изображение находок Blastp для g10 - g6 Здесь опять та же ситуация. Ген неправильно определен. Однако на данном примере можно наглядно увидеть ошибки. На рисунке 4
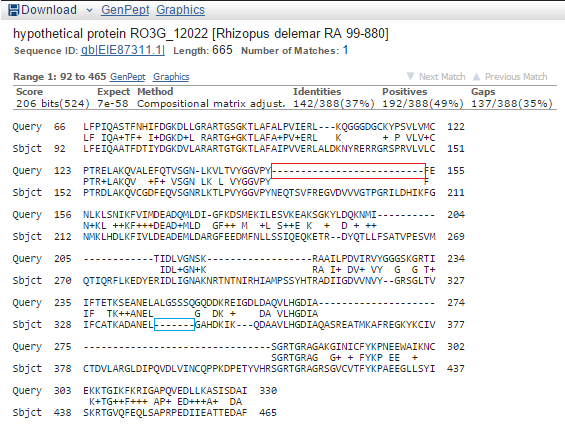
Рисунок 4 Выравнивание белка,закодированногот в g10 и hypothetical protein из Rhizopus delemar. Красным выделен участок белка, чья кодирующая последовательность является экзоном, но Augustus распознал ее как интрон. Синим - интрон, который распознан как экзон. - Далее я безуспешно пыталась найти пример точно предсказанного гена. Но все что осталось, это небольшие домены с низким процентом идентичности.
И тут я поняла, что нужно было брать контиг более подходящий для данной работы. На всякий случай я сделала еще раз задание но сдругим контигом.
Там был точно предсказанный ген. Но процент идентичности все равно оставался на уровне 50. (см. Рисунок 5)
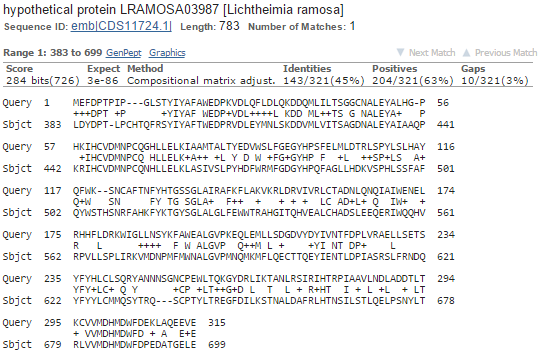
Рисунок 5 Выравнивание белка DUT, чей ген был верно предсказан.
Сравнение аннотации Refseq Augustus для одного гена человека
Для выполнения задания был выбран ген (GI: 1312) белка катехол-О-метилтрансферазы Catechol-O-methyl transferase (COMT) — фермент, играющий важную роль в распаде катехоламинов, в том числе дофамина, адреналина, норадреналина, так как присутствует диффузно во всех тканях. COMT катализирует присоединение к катехоламину метильной группы, донором которой служит S-Аденозилметионин. Участвует в процессе развития шизофрении.
Координаты гена: 19941740..19969975 прямая цепь, 22 хромосома
С помощью UCSC Genome Browse были получены две аннотации гена COMT. Графическое представление браузера представлено на Рисунке 6
Оставлено 3 трека (ту информацию, которую хотим увидеть): base position, RefSeq, Augustus.
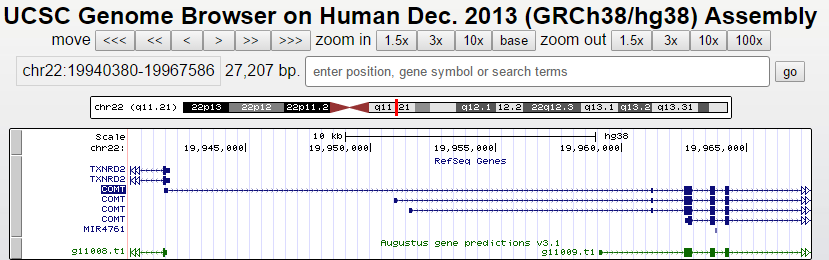
Рисунок 6 Изображение UCSC Genome Browse с аннотациями COMT
Далее по отдельности были получены предскзания экзон-интронной структуры, сделанные RefSeq и Augustus. Как видно из рисунка у белка есть несколько изоформ из-за альтернативного сплайсинга. Была оставлена одна самая полная.
Результаты сравнения представлены здесь и на Рисунке.
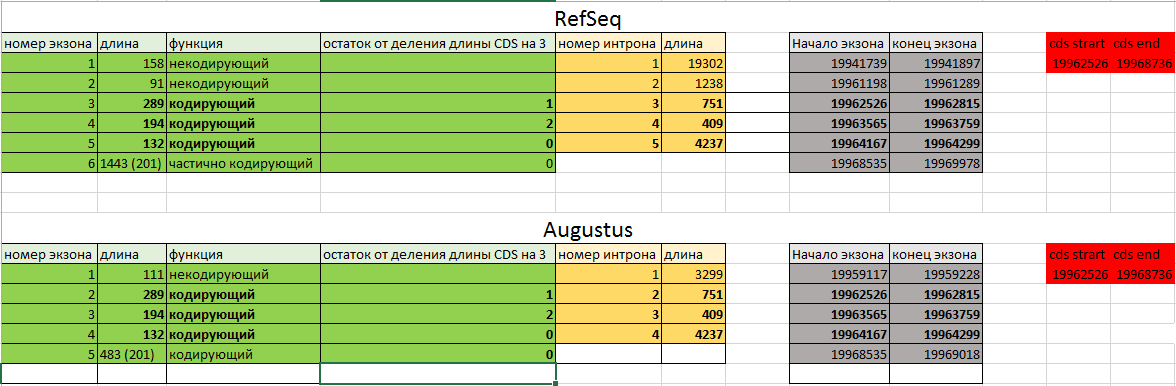
Рисунок 7 Сравнение предсказаний Augustus и RefSeq. Жирным шрифтом выделены одинаково предсказанные гены.
© 2014 Макарова Надежда