|
Третий семестр
Для выполнения заданий данного практикума был взят домен Laminin_N (ID в базе данных PFAM: PF00055).
Это домен представленный в семействе крупных адгезивных гликопротеинов. Они являются одним из основных компонентов базальной пластинки и влияют на дифференцировку клеток, миграцию и адгезию.
В программе JalView было построено выравнивание последовательностей данного домена. Последовательности раскрашены по консервативности ClustalX с порогом 15%. В рабочей директории был запущен скрипт python swisspfam_to_xls.py -p PF00055 -i /srv/databases/pfam/swisspfam.gz -z -o arch.txt Файл arch.txt был переведен в формат книги Excel, была сделана сводная таблица , отражающая количество архитектур определенного типа в белках из UniProt, обязательно содержащих домен Laminin_N. Были выбраны две архитектуры белков, содержащих данный домен (Рис.1): архитектура, содержащая только домен Laminin_N, и архитектура, содержащая 2 домена: Laminin_N и домен Laminin_EGF (домен схож по функциям с Laminin_N) . 
Из распределения по таксонам (Рис.2) в качетсве таксона было выбрано царство Metazoa, а в качестве подтаксонов типы Chordata и клада Ecdysozoa. 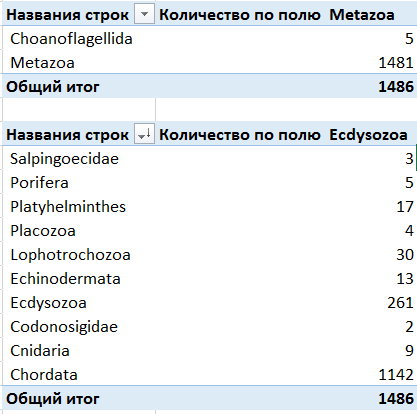
Белки, принадлежащие первой (1 домен) или второй (2 домена) архитектурам, обозначали соответственно 1 и 2, белки, принадлежащие Chordata и Ecdysozoa, соответственно _chor и _ecd .
Отредактированное выравнивание выбранных последовательностей можно посмотреть здесь , а
скобочную формулу построенного по нему дерева можно скачать по данной ссылке.
Дерево построено методом Neighbor joining с бутстреп поодержкой в 100 реплик и представлено на Рис.3.
ВыводыУже по выравнивнию (Рис.4) стало заметно, что белки, содержащие только домен Laminin_N сильно отличаются от белков с другой архитектурой. У белков только с Laminin_N этот домен укороченный. Причем как с N-конца, так и с C- конца. И там есть различия в таксонах. У Chordata он еще более ли менее сохранился, а вот у Ecdysozoa большая часть отсутствует (и это не порченные белки, так как дальше есть небольшая аминокислотная последоатльеность, невыравнивающаяся).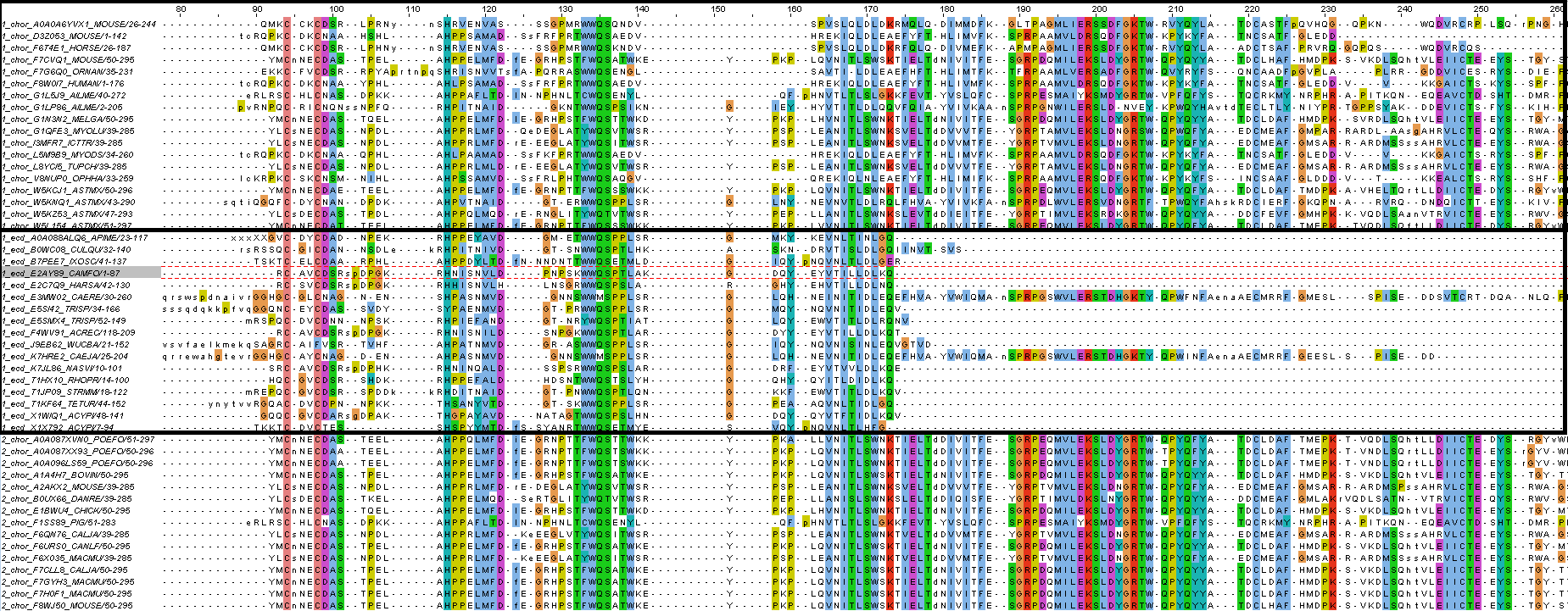
На дереве можно видеть большую кладу с усложненной архитектурой (то есть с двумя доменами) у таксона Chordata, которая есть на дереве в двух местах. Больше повторных клад не наблюдается. Клада же с одним единственным доменом четко отделена и разделаюется внутри на два таксона. Придерживаясь принципа парсимонии, так как последовательностей с усложненной архитектурой больше, то можно предположить, что изначально был многодоменный белок и на каком-то этапе эволюции домены белка разошлись и домен стал существовать в белке с урезанной доменной архитектурой, в нашем случае, в однодоменном белке. Еще стоит заметить, что по функциям эти домены не отличаются. Наверное, это может служить своеобразным критерием времени: домены внутри предкового белка разошлись относительно недавно и не успели сильно измениться. Теперь нужно разобраться с таксонами. В большой красной верхней кладе с усложненной архитектурой Chordata есть достаточно много зеленых прямоугольников упрощенной архитектуры Chordata. Что говорить о том, что дело было так: Chordata 2 домена -> Chordata 1 домен. Если бы эволюция в таксонах шла параллельно, тогда мы бы увидели маленькие желтые прямоугольники упрощенной архитектуры Ecdysozoa в голубой кладе усложненной архитектуры Ecdysozoa. Но мы такого не наблюдаем. Но наблюдаем желтые прямоугольники упрощенной архитектуры Ecdysozoa во второй красной кладе Chordata с усложненной архитектурой. Это значит, что схема такова Chordata 2 домена -> Ecdysozoa 1 домен. Это подверждается еще и тем, что клады упрощенных доменов из Chordata и Ecdysozoa объединены в кладу с красной ветвью с усложненной архитектурой Chordata Это возможно только при горизонтальном переносе (может быть косвенно?). Или что-то еще более интересное. Профиль семейства последовательностей белковДля построения было выбрано подсемейство Ecdysozoa с архитехтурой из двух доменов. Из выравнивания последовательностей домена, были удалены все последовательности, кроме выбранных. Далее для построения профиля использовались программы пакета HMMER3 Профиль по выравниванию был построен программой hmmbuild: hmm2build --amino out_hmm_2_chord 2_chord.stk На основе результатов поиска был получен файл Excel (на листе "FINAL_TAB_13" содержатся данные об E-value для каждой находки и столбец с правильными находками (представителями семейства). Затем с помощью функции ВПР были определены соответствия между полученными данными и предсказаниями (был создан столбец "наличие в представителях семейства", в котором отмечены последовательности (среди всех находок), участвовавшие в построении профиля (1), а также все остальные находки (0). На основании полученных данных была построена ROC-кривая (Рис.5). Ее изображение представлено на листе "ROC". 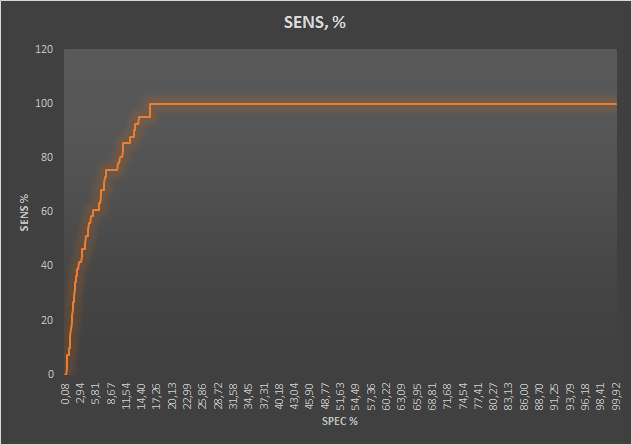
По ROC-кривой можно выбрать порог E-value = 5.8E-130. При этом значении наблюдается резкое изменение чувствительности. Она становится максимальна.
Дата последнего изменения: 14.09.16
© 2014 Макарова Надежда |
|||||||||||||||||||||