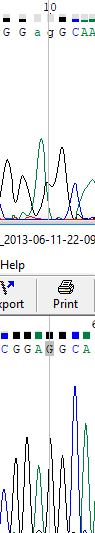
 Изображение 1: из-за наложения пиков друг на друга, неопределенность в 9 и 10 позициях прямой цепи (видимо слишком маленькое
расстояние расхождения фрагментов цепи), по обратной цепи однозначно подставляются аденин и гуанин.
Изображение 1: из-за наложения пиков друг на друга, неопределенность в 9 и 10 позициях прямой цепи (видимо слишком маленькое
расстояние расхождения фрагментов цепи), по обратной цепи однозначно подставляются аденин и гуанин.Для исследования были получены 2 файла с данными, полученными из капиллярного секвинатора. А имеено хроматограммы прямой и обратной цепей
одной и той же последовательности ДНК. Для обратной цепи была получена комплементарная последовательность для удобства сравнения ее с прямой(далее
под координатами нуклеотида в обратной цепи будет подразумеваться его номер в комплементарной последовательности). Сачала были определены нечитаемы участки для каждой цепи и сопоставлены их координаты.
Границы нечитаемых участков прямой последовательности:
5'конец:1-20(далее сигнал еще слабый, но пики различимы и при помощи
обратной цепи возможно прочитать последовательность).
3'конец: 380-381(далее сигнал совсем пропадает).
Границы нечитаемых участков обратной последовательности:
3'конец изначатьной, собственная нумерация:1-3.
5'конец изначальной: 356-388.
Сопоставление нечитаемых участкой (координаты участкой обратной цепи, относительно прямой): 1 читаемый нуклеотид прямой цепи, соответсвует
соответствует 47 отраженной обратной цепи, последний читаемый нуклеотид отображенной обратной цепи(352) соответсвует 306 прямой цепи.
Далее производилось исправление возможных ошибок определения нуклеотидов в прямой цепи, с обратной цепью в качестве шаблона. Примеры исправлений(на всех приведенных
ниже изображениях хроматограмма прямой цепи располагается сверху, а отображенная хроматограмма обратной цепи- снизу):
 Изображение 1: из-за наложения пиков друг на друга, неопределенность в 9 и 10 позициях прямой цепи (видимо слишком маленькое
расстояние расхождения фрагментов цепи), по обратной цепи однозначно подставляются аденин и гуанин.
Изображение 1: из-за наложения пиков друг на друга, неопределенность в 9 и 10 позициях прямой цепи (видимо слишком маленькое
расстояние расхождения фрагментов цепи), по обратной цепи однозначно подставляются аденин и гуанин.
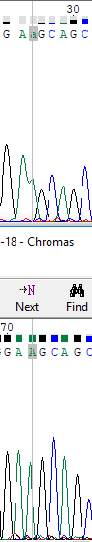 Изображение 2:слияние пиков на хроматограмме прямой цепи, из-за этого неопределенность в положении 26. С помощью хроматограммы
обратной цепи однозначно определяется два аденина, соответствующие слившимся пикам.
Изображение 2:слияние пиков на хроматограмме прямой цепи, из-за этого неопределенность в положении 26. С помощью хроматограммы
обратной цепи однозначно определяется два аденина, соответствующие слившимся пикам.
После 326 нуклеотида прямой цепи нет возможности пользоваться обратной цепьюкак шаблоном, так как на оставшемся участке обратная цепь не читаема. Но из общих соображений можно сделать некоторые заключения:
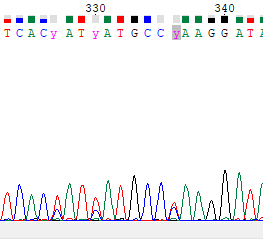 Изображение 3:фрагмент хроматограммы прямой цепи. В позициях 328, 330 и 332 наблюдается полиморфизм(пики почти одинаковой высоты).
Изображение 3:фрагмент хроматограммы прямой цепи. В позициях 328, 330 и 332 наблюдается полиморфизм(пики почти одинаковой высоты).
Затем аналогичная работа по корректировке последовательности была проведена для отображенной обратной цепи, с прямой цепью в качестве шаблона(начиная с 50 нуклеотида обратной цепи). Примеры исправлений(как и на изображениях приведенных выше, сверху расположена хроматограмма прямой цепи, снизу- отображенной обратной):
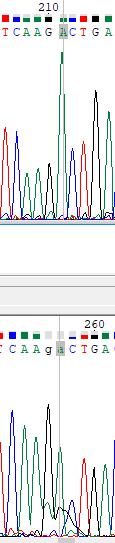 Изображение 4:пики на хроматограмме обратной цепи четкие, но в положениях 256 и 255 очень велик уровень "шума" поэтому данный участок требует проверки, по прямой цепи
достраивается гуанин и аденин соответственно.
Изображение 4:пики на хроматограмме обратной цепи четкие, но в положениях 256 и 255 очень велик уровень "шума" поэтому данный участок требует проверки, по прямой цепи
достраивается гуанин и аденин соответственно.
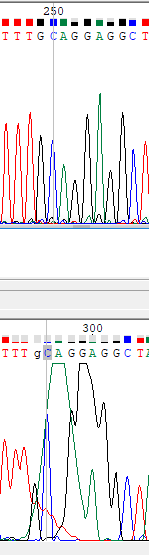 Изображение 5:в положении 296 пики сильно наслаиваются друг на друга, а дальше заметен резкий скачек, поэтому сложно проверить правильность данного фрагмента без прямой цепи. С помощью
прямой цепи однозначно достраивается гуанин в положении 296.
Изображение 5:в положении 296 пики сильно наслаиваются друг на друга, а дальше заметен резкий скачек, поэтому сложно проверить правильность данного фрагмента без прямой цепи. С помощью
прямой цепи однозначно достраивается гуанин в положении 296.
Посмотреть получившиеся последовательности в формате fasta:
Прямая цепь.
Отображенная обратная цепь.
Общее заключение о качестве хроматограммы:в целом, качество хроматограммы хорошеее, уровень шума очень низкий. К тому же начальные и конечные нечитаемые фрагменты довольно короткие. Сигнал четкий почти на всем протяжении хроматограммы(не считая нечитаемых участков), наиболее отчетливый сигнал в средней части каждой хроматограммы.
Было проведено выравнивание отредактированных прямой и обратной цепей. Выравнивание создано с помошью программы "needle". Jalview-проект (раскраска по нуклеотидам) с результатом выравнивания можно скачать по ссылке.
Как видно из выравнивания, последовательности полностью совпали, не считая концевых участков, каждый из которых читается только у одной из 2-х последовательностей. Но даже на этих участках определены все нуклеотиды, корме одного: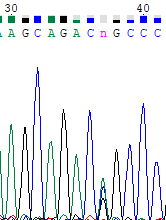 Изображение 6:фрагмент отображенной обратной цепи, позиции 37 соответствует сразу 3 пика, незначительно отличающихся между собой, но значительно превышающих уровень шума каждый. Участка прямой
цепи, соответсвущего этому, нет, поэтому точно определить, что должно стоять в позиции 37 нельзя.
Изображение 6:фрагмент отображенной обратной цепи, позиции 37 соответствует сразу 3 пика, незначительно отличающихся между собой, но значительно превышающих уровень шума каждый. Участка прямой
цепи, соответсвущего этому, нет, поэтому точно определить, что должно стоять в позиции 37 нельзя.
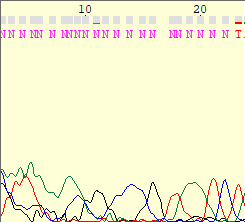 Изображение 7:начальный (со стороны 5'-конца) нечитаемый фрагмент хроматограммы прямой цепи. Первые 10 нуклеотидов
определить по данной хроматограмме невозможно, так как нет никаких преобладающих пиков в каждом положении. С 11 по 20 нуклеотидв пики очень
размытые и, скорее всего, многие из них представляют несколько соседних пиков слившихся в один, сложно сказать сколько именно нуклеотидов должны
соответствовать каждому пику, поэтому этот участок все еще не годится для точного определения последовательности. Фрагмент с 20 по 23 нуклеотид программа
отметила как нечитаемы, тем не менее там есть 3 невысоких, но вполне различимых пика. Участок 20-23 может быть определен по данной хроматограмме, но все же требует проверки
по обратной цепи.
Изображение 7:начальный (со стороны 5'-конца) нечитаемый фрагмент хроматограммы прямой цепи. Первые 10 нуклеотидов
определить по данной хроматограмме невозможно, так как нет никаких преобладающих пиков в каждом положении. С 11 по 20 нуклеотидв пики очень
размытые и, скорее всего, многие из них представляют несколько соседних пиков слившихся в один, сложно сказать сколько именно нуклеотидов должны
соответствовать каждому пику, поэтому этот участок все еще не годится для точного определения последовательности. Фрагмент с 20 по 23 нуклеотид программа
отметила как нечитаемы, тем не менее там есть 3 невысоких, но вполне различимых пика. Участок 20-23 может быть определен по данной хроматограмме, но все же требует проверки
по обратной цепи.
Третий семестр(осенний семестр 2018)