В файле приведены координаты атомов следующих молекул из SACCHAROMYCES CEREVISIAE:
Молекула-1: цитоплазматическая тирозил-тРНК синтетаза, молекула-2: Т-РНК (76-MER)..
Для исследования была выбрана цепь Y, представляющая тРНК (76-MER) со следующей последовательностью:
[501] 5' - CUCUCGGUAGCCAAGUUGGGAAGGCGCAAGACUGUAAAUCUUGAGGUCGGGCGUUCGACUCGCCCCCGGGAGACCA - 3' [576],
где 501 и 576 - номера первого и последнего нуклеотида.Следует отметить, что в последовательности на 3'-конце находится триплет CCA, к которому присоединяется аминокислота, с координатами атомов 4110-4172
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями ссылка на выходной файл .
В соответствии с полученными данными
акцепторный стебель состоит из участка 501-507 и комплементарного ему участка 566-572.
Т-стебель.........................................549-553 и комплементарного ему участка 561-565
D-стебель........................................510-513 и комплементарного ему участка 522-525
антикодоновый стебель..............542-544 и комплементарного ему участка 526-528
Скрипт для получения в RasMol изображения остова исследуемой тРНК
(акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым).
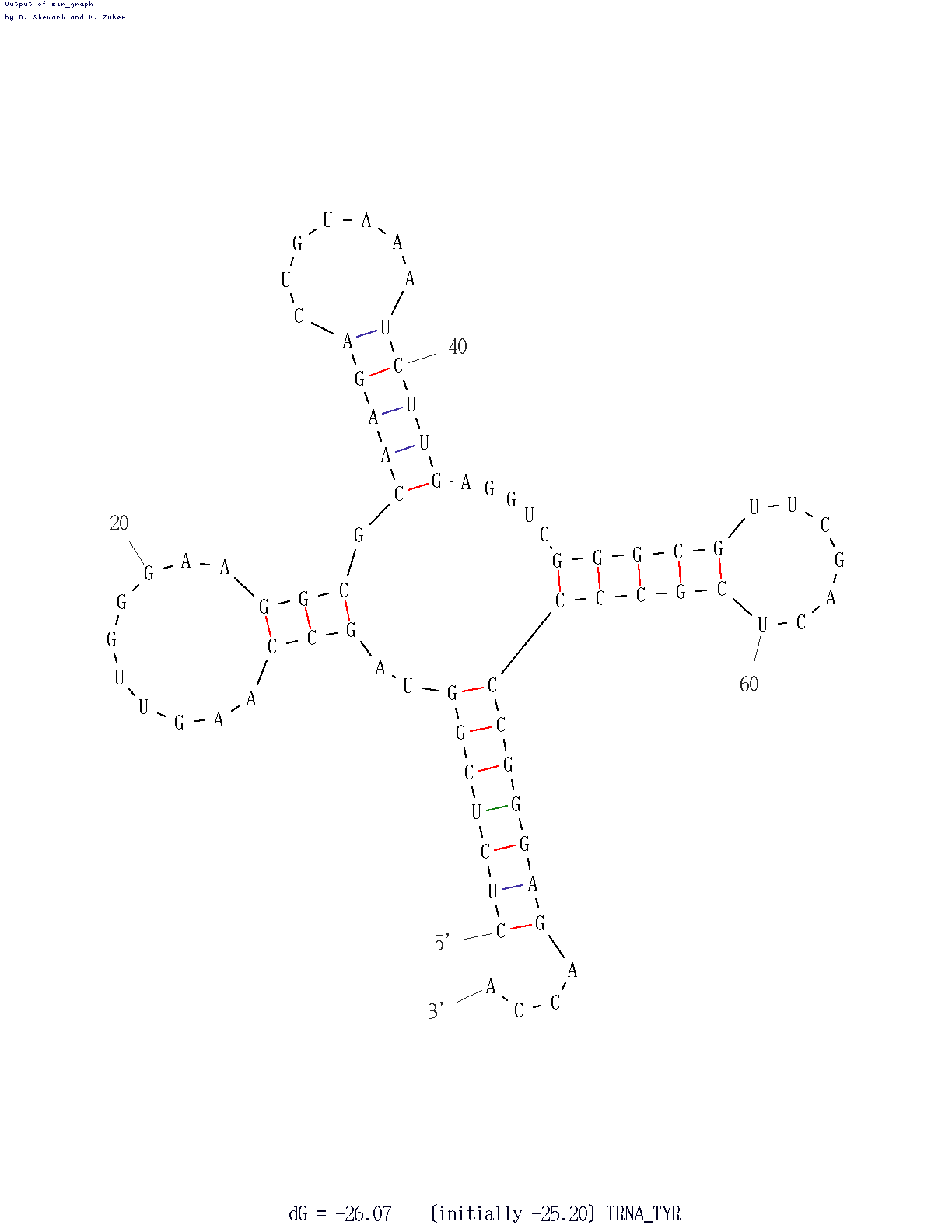
| Рис.1. Вторичная структура тРНК (76-MER) из SACCHAROMYCES CEREVISIAE | Скрипт для получения изображения |
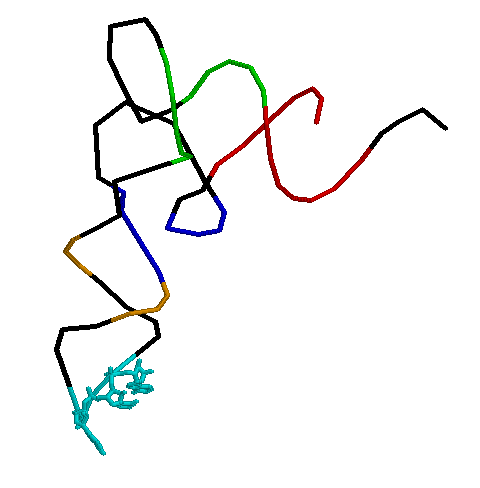 |
zap load 1dlc.pdb restrict none background white restrict backbone backbone 70 select backbone color black select 501-507:Y, 566-572:Y color red select 549-553:Y, 561-565:Y color green select 510-513:Y, 522-525:Y color blue select 542-544:Y, 526-528:Y color orange {select 531-534:Y color cyan wireframe 70 cpk 100 *антикодон AUG в антикодоновой петле} |
Из неканонических пар на рисунке представлена пара U504-G569
Следует также отметить
а)наличие вариабельной петли: 545..548;
b)отсутствие остатка тимидина в Т-петле, дигидроуридинов в D-петле
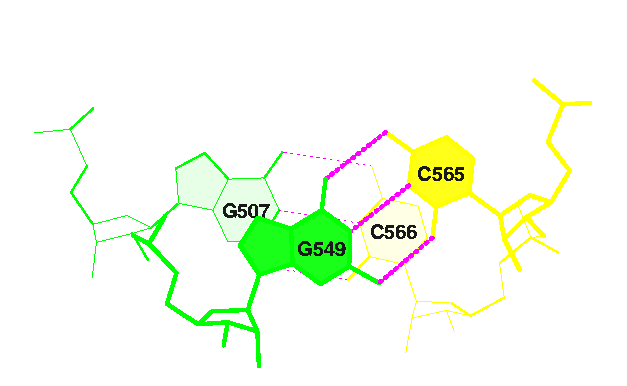
1. Cтекинг-взаимодействие между основаниями конца акцепторно стебля G507/C566 и начала
Т-стебля G549/C565: 2.98, a между антикодоновым и D-стеблем: 1.03, поэтому изображение
было получено для первого взаимодействия:
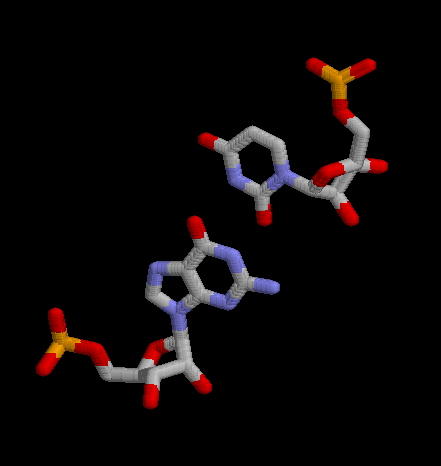
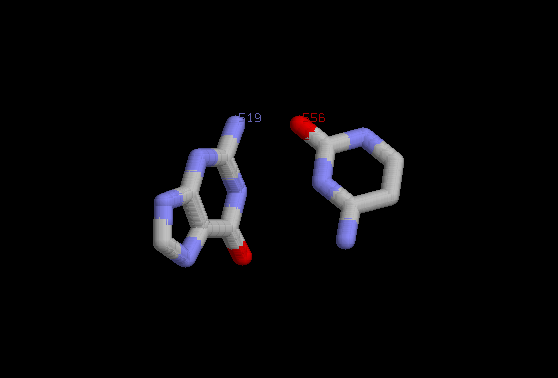
2. Дополнительные водородные связи между основаниями D- и Т-петель: одна неканоническая
пара PSU555-G518, и одна каноническая пара G519-C556, последняя из них представлена на картинке
С помощью программы einverted были найдены инвертированные участки,
начиная с установки значения 15 для параметра "Minimum score threshold",
при котором нашлись 5 пар нуклеотидов в Т-стебле и 5 пар в антикодоновом стебле,
остальные параметры не менялись. При уменьшении значения этого параметра вплоть до
1-го находились только те же 5 пар в Т-стебле.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' -501-507- 3' 5' -566-572- 3' Всего 7 пар |
Не предсказано ни одной пары | 5' -501-507- 3' 5' -566-572- 3' Всего 7 пар |
| D-стебель | 5' -510-513- 3' 5' -522-525- 3' Всего 4 пары |
Не предсказано ни одной пары | 5' -510-512- 3' 5' -523-525- 3' Всего 3 пары |
| T-стебель | 5' -549-553- 3' 5' -561-565- 3' Всего 5 пар |
5' -549-553- 3' 5' -561-565- 3' Всего 5 пар |
5' -549-553- 3' 5' -561-565- 3' Всего 5 пар |
| Антикодоновый стебель | 5' -542-544- 3' 5' -526-528- 3' Всего 3 пары |
5' -527-531- 3' 5' -539-543- 3' Всего 5 пар |
5' -527-531- 3' 5' -539-543- 3' Всего 5 пар |
| Общее число канонических пар нуклеотидов | 20 | 10 | 19 |