Реконструкция филогенетических деревьев
Занятие 2. Реконструкция филогенетических деревьев
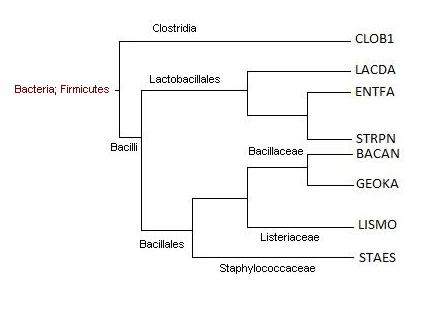
Таксономический сервис NCBI
Bacillus anthracis....................Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group
Clostridium botulinum.............Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Enterococcus faecalis.............Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus
Geobacillus kaustophilus.........Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus
Lactobacillus delbrueckii........Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus
Listeria monocytogenes..........Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria
Staphylococcus epidermidis...Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
Streptococcus pneumoniae.....Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus
Поиск в Swiss-Prot
Из списка функций белков была выбрана: Фактор элонгации трансляции 4/LEPA, по белкам соответствующего семейства будет
реконструировано филогенетическое дерево. Из Swiss-Prot были получены
последовательности белков с данной функцией из
отобранных бактерий и выравнены программой muscle и визуализированы в GeneDoc .

Pеконструкция дерева программой fprotpars
Программа выдала одно дерево. Cкобочная формула:
((((LEPA_GEOKA,LEPA_BACAN),(((LEPA_STRPN,LEPA_ENTFA),LEPA_LISMO),LEPA_STAES)),LEPA_CLOB1),LEPA_LACDA);
Изображение деревьев: слева - полученное программой fprotpars, а справа - правильное
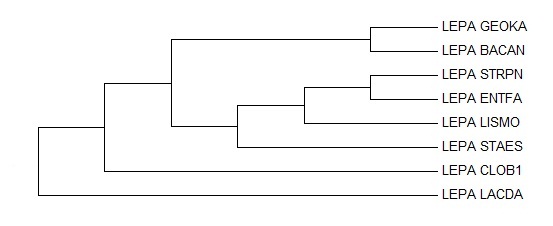
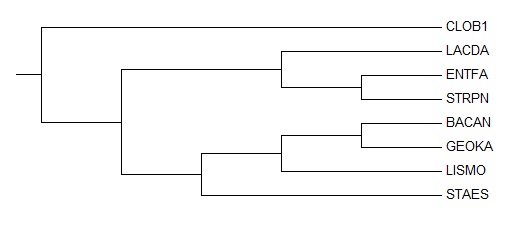
Cравнение полученного дерева с правильным
В правильном девере присутствуют ветви, отсутствующие в выдаче программы fprotpars:
1) {LACDA, ENTFA, STRPN} vs {CLOB1, BACAN, GEOKA, LISMO, STAES}
2) {CLOB1, LACDA, ENTFA, STRPN} vs {BACAN, GEOKA, LISMO, STAES}
3) {BACAN, GEOKA, LISMO} vs {CLOB1, LACDA, ENTFA, STRPN, STAES}
В выдаче программы fprotpars присутствуют ветви, отсутствующие в правильном девере:
1) {LEPA CLOB1, LEPA LACDA} vs {LEPA ENTFA, LEPA STRPN, LEPA BACAN, LEPA GEOKA, LEPA LISMO, LEPA STAES}
2) {LEPA ENTFA, LEPA STRPN, LEPA LISMO} vs {LEPA CLOB1, LEPA LACDA, LEPA BACAN, LEPA GEOKA, LEPA STAES}
3) {LEPA ENTFA, LEPA STRPN, LEPA LISMO, LEPA STAES} vs {LEPA CLOB1, LEPA LACDA, LEPA BACAN, LEPA GEOKA}
Эволюционные расстояния между последовательностями
Программой fprotdist была получена матрица расстояний
|
|
LEPA_LACDA
|
LEPA_CLOB1
|
LEPA_STAES
|
LEPA_BACAN
|
LEPA_GEOKA
|
LEPA_LISMO
|
LEPA_ENTFA
|
LEPA_STRPN
|
| LEPA_LACDA
|
0.000000
|
0.571728
|
0.544579
|
0.509776
|
0.488149
|
0.480280
|
0.459744
|
0.485074
|
| LEPA_CLOB1
|
0.571728
|
0.000000
|
0.504506
|
0.416720
|
0.387074
|
0.403618
|
0.451756
|
0.481146
|
| LEPA_STAES
|
0.544579
|
0.504506
|
0.000000
|
0.307129
|
0.291492
|
0.283476
|
0.293306
|
0.329237
|
| LEPA_BACAN
|
0.509776
|
0.416720
|
0.307129
|
0.000000
|
0.174603
|
0.199340
|
0.260083
|
0.278787
|
| LEPA_GEOKA
|
0.488149
|
0.387074
|
0.291492
|
0.174603
|
0.000000
|
0.224005
|
0.246539
|
0.272680
|
| LEPA_LISMO
|
0.480280
|
0.403618
|
0.283476
|
0.199340
|
0.224005
|
0.000000
|
0.188812
|
0.244084
|
| LEPA_ENTFA
|
0.459744
|
0.451756
|
0.293306
|
0.260083
|
0.246539
|
0.188812
|
0.000000
|
0.184502
|
| LEPA_STRPN
|
0.485074
|
0.481146
|
0.329237
|
0.278787
|
0.272680
|
0.244084
|
0.184502
|
0.000000
|
Проверка ультраметричности
Так как d(LEPA_LACDA, LEPA_CLOB1) = 0.571728 > d(LEPA_CLOB1, LEPA_STAES) = 0.504506,
то должно выполняться равенство: d(LEPA_LACDA, LEPA_STAES) - d(LEPA_LACDA, LEPA_CLOB1) = 0,
но в данном случае эта разность равна: 0.544579 - 0.571728 = -0,027149 и показывает
отклонение от ультраметричности.
Проверка аддитивности
Из трех сумм:
d(LEPA_LACDA, LEPA_CLOB1) + d(LEPA_STAES, LEPA_BACAN) = 0.571728 + 0.307129 = 0,878857
d(LEPA_LACDA, LEPA_STAES) + d(LEPA_CLOB1, LEPA_BACAN) = 0.544579 + 0.416720 = 0,961299
d(LEPA_LACDA, LEPA_BACAN) + d(LEPA_CLOB1, LEPA_STAES) = 0.509776 + 0.504506 = 1,014282
две должны быть равны и больше третьей: в данном случае две последние суммы больше первой,
но они не равны, и их разность составляет 0,052983 и показывает отклонение от аддитивности.
Реконструкции дерева
Программой fneighbor были оплуччены две реконструкции дерева по двум алгоритмам:
UPGMA, применяемая приусловии справедливости гипотезы молекулярных часов
(матрица расстояний не слишком далека от ультраметрической)(картинка слева),
и Neighbor-Joining (картинка справа), с длинами ветвей при обоих алгоритмах
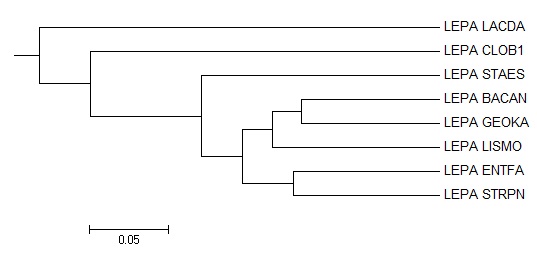
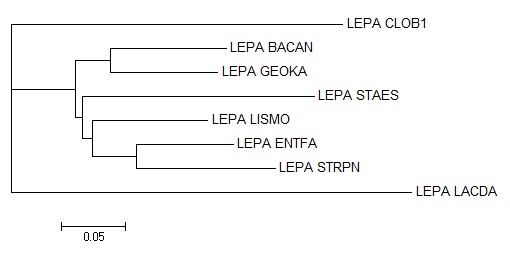
Общие ветви полученных реконструкций программой fneighbor :
1){LEPA CLOB1, LEPA LACDA} vs {LEPA ENTFA, LEPA STRPN, LEPA BACAN, LEPA GEOKA, LEPA LISMO, LEPA STAES}
(имеется в выдаче программы fprotpars, но но отсутствует в правильном дереве)
2){LEPA ENTFA, LEPA STRPN} vs {LEPA CLOB1, LEPA LACDA, LEPA BACAN, LEPA GEOKA, LEPA LISMO, LEPA STAES}
3){LEPA BACAN, LEPA GEOKA} vs {LEPA CLOB1, LEPA LACDA, LEPA ENTFA, LEPA STRPN, LEPA LISMO, LEPA STAES}
(2-я и 3-я имеются как в выдаче программы fprotpars, так и в правильном дереве)
Ветви, характерные выдаче программы fneighbor только с алгоритмом UPGMA:
1){LEPA BACAN, LEPA GEOKA, LEPA LISMO} vs {LEPA CLOB1, LEPA LACDA, LEPA ENTFA, LEPA STRPN, LEPA STAES}
(имеется еще только в правильном дереве)
2){LEPA CLOB1, LEPA LACDA, LEPA STAES} vs {LEPA ENTFA, LEPA STRPN, LEPA BACAN, LEPA GEOKA, LEPA LISMO}
(выдалось только этой программой с этим алгоритмом)
Ветви, выданные программой fneighbor только с алгоритмом Neighbor-Joining:
1){LEPA ENTFA, LEPA STRPN, LEPA LISMO} vs {LEPA CLOB1, LEPA LACDA, LEPA BACAN, LEPA GEOKA, LEPA STAES}
2){LEPA ENTFA, LEPA STRPN, LEPA LISMO, LEPA STAES} vs {LEPA CLOB1, LEPA LACDA, LEPA BACAN, LEPA GEOKA}
(имееются обе еще и в выдаче программы fprotpars)
Как правильное дерево, так и дерево, полученное программой fneighbor с алгоритмом UPGMA,
выдаются укорененными, в отличие от деревьев, полученных программами fprotpars и fneighbor ,
только с алгоритмом Neighbor-Joining,- они выдаются неукорененными.
©Андреянова Екатерина





