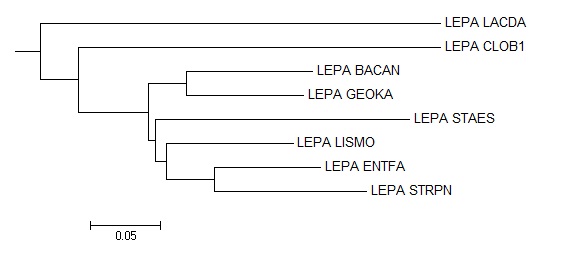
Данное укоренение получено с учетов масшабов предполагаемых эволюционных
расстояний в ветвь отделяющую LEPA LACDA от всех остальных, в то время
как в правильном дереве укоренение отделяет СLOB1 от остальных.
Деревья, построенные методом максимальной экономии, нельзя укоренить,
так как этот метод не выдает длин ветвей и, соответственно, нельзя
найти среднюю точку.
Процесс укоренения в среднюю точку не
имеет смысла с деревом, построенным методом UPGMA, так как этот метод
выдает уже укорененное дерево и нет смысла укоренять его заново.
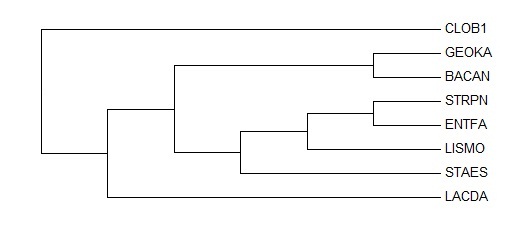
Вместе с добавленной внешней группой были выровнены выбранные последовательности
белков, и на основе полученного выравнивания построено дерево методом максимальной
экономии программой fprotpars. На выходе этой программы было такое же дерево, как
и без внешней группы, что указывает на сходство полученных в обоих случаях
выравниваний.
По сравнению с правильным деревом только две ветви совпадают ({STRPN,
ENTFA} vs {CLOB1, GEOKA, BACAN, LISMO, STAES, LACDA}; {GEOKA, BACAN} vs {CLOB1,
STRPN, ENTFA, LISMO, STAES, LACDA}), а остальные три нет.
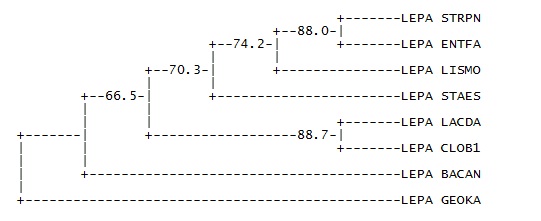
Реконструкция филогении по сравнению с результатом программы fprotpars на
исходном выравнивании привела к выдаче таких же ветвей. Однако некоторые верные
ветви не были включены в консенсусное дерево:
{STRPN,ENTFA,LACDA} vs {GEOKA,BACAN,LISMO,STAES,CLOB1} с поддержкой 8.00;
{GEOKA,BACAN,LISMO,STAES} vs {STRPN,ENTFA,LACDA,CLOB1} c поддержкой 2.00;
в выходном файле указано 20 ветвей, не включенных в консенсусное дерево, и 5
ветвей, включенных, из которых только две соответствуют правильной форме.