| Мнемоника бактерии | AC в записи EMBL | Координаты рРНК |
CLOB1 |
CP000726 |
complement |
LACDA |
CR954253 |
293362..294252 |
ENTFA |
AE016830 |
complement |
STRPN |
AE005672 |
733725..734243 |
BACAN |
AE016879 |
complement |
GEOKA |
BA000043 |
1220071..1220598 |
LISMO |
AL591977 |
82197..82706 |
STAES |
AE015929 |
complement |
Филогенетическое дерево отобранных бактерий было построено, используя последовательности РНК малой субъединицы рибосомы (16S rRNA) с помощью программы ffitch, которой на вход была подана матрица расстояний, полученная программой fprotdist на основе выравненных последовательностей:
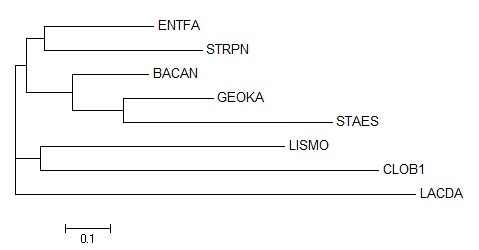
Проанализировав полученное дерево, можно сделать вывод, что качество
реконструкции по сравнению с деревьями, построенным по белкам, хуже,
так как только одна ветвь, совпадающая с правильным деревом и с
деревьями, полеченными другими методами, нашлась, но одна из двух
самых распространенных:
{ENTFA,STRPN} vs {CLOB1,LACDA,BACAN,GEOKA,LISMO,STAES}
Построение и анализ дерева, содержащего паралоги
В отобранных бактериях были найдены достоверные гомологи белка CLPX_BACSU с помощью программы BLASTP с порогом на E-value, равным 0.001, в результате чего было получено 16 гомологов нужных бактерий, на основании выравниваний последовательностей которых уже было построено дерево программой fprotpars:
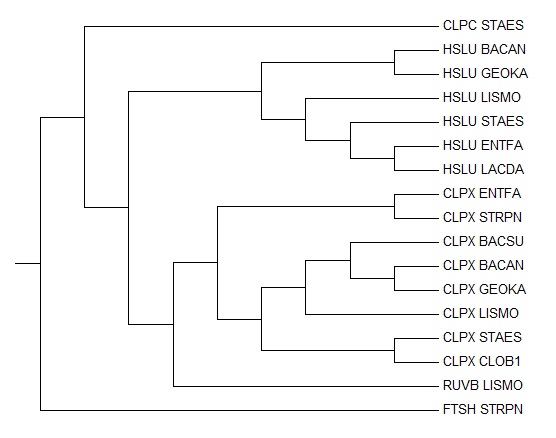
Среди найденных гомологичных белков следует отметить ортологи:
HSLU_BACAN и HSLU_GEOKA; CLPX_BACSU и CLPX_BACAN; CLPX_BACSU и CLPX_GEOKA,
а также паралоги:
HSLU_LISMO, CLPX_LISMO и RUVB_LISMO; FTSH_STRPN и CLPX_STRPN; HSLU_GEOKA и CLPX_GEOKA.
©Андреянова Екатерина