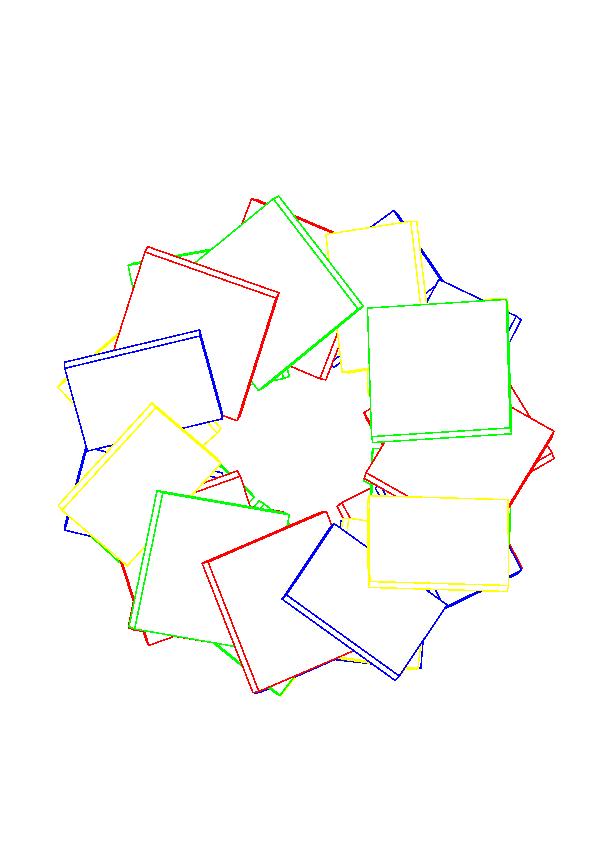
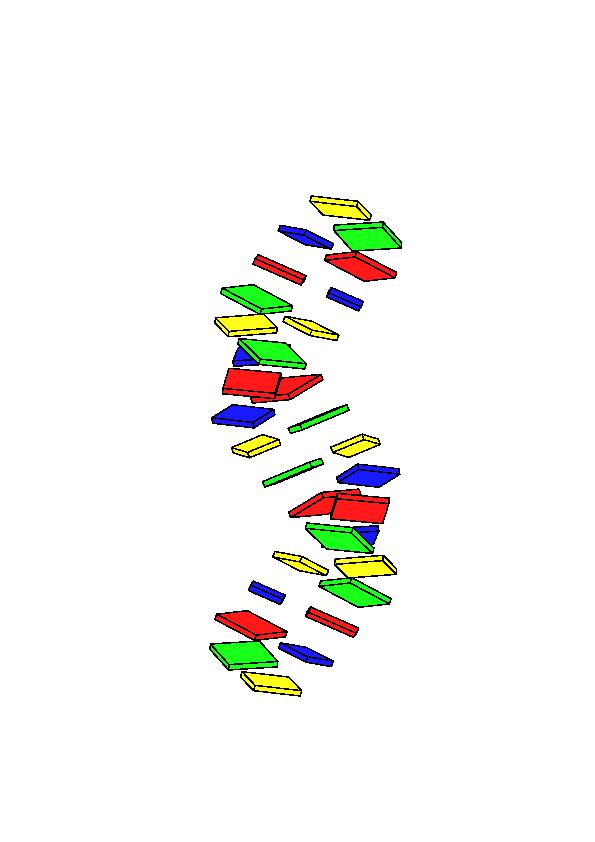
1.
Здесь можно найти файлы с А- и В-формами дуплекса ДНК,последовательность одной из нитей которого-4 раза повторенная последовательность "gatc": gatc_a.pdb и gatc_b.pdb2.
| A-форма | B-форма | Файл dnaN.pdb | |
| Тип спирали (правая или левая) | правая | правая | правая |
| Шаг спирали (Å) | 28,02 | 33,75 | 26,65 |
| Число оснований на виток | 12 | 11 | 12 |
| Ширина большой бороздки | 7.98 | 17.20 | 9.18 |
| Ширина малой бороздки | 16.8 | 11.69 | 15.86 |
3.
В последней колонке таблицы предыдущего пункта можно увидеть данные,полученные из файла:dna18.pdb
Расстояния измерялись в RasMol с помощью Pick Ident. Все расстояния измерялись по атомам фосфора. Визуально ДНК из этого файла напоминает
А-форму. Из-за характерного отверстия посередине,если смотреть с торца. Это также подтверждается числом
оснований на виток, и шириной малой и большой бороздок.
4.
С помощью команд find_pair -t XXXX.pdb stdout | analyze где XXXX.pdb - соответственно gatc-a.pdb, gatc-b.pdb,
и dna18.pdb, были получены файлы с расширением .out. В этих файлах можно найти информацию о торсионных углах
торсионные углы для gatc-a.pdb (А-форма ДНК)
Strand I
base alpha beta gamma delta epsilon zeta chi |
торсионные углы для gatc-b.pdb (В-форма ДНК)
Strand I
base alpha beta gamma delta epsilon zeta chi |
торсионные углы для dna18.pdb (мой кусок ДНК)
Strand I
base alpha beta gamma delta epsilon zeta chi |
Сравнивая торсионные углы А-и В-форм ДНК можно заметить,что углы gamma и epsilon достаточно сильно похожи по значениям,тогда как все остальные довольно четко различаются. Теперь посмотрим на торсионные углы ДНК из файла dna18.pdb: судя по ним с большой долей вероятности это именно А-форма ДНК.
5.
Следующие команды были применены для получения изображений с помощью команды pdb2img:
а) для поворота молекулы:
rotate_mol -b gatc_a.pdb gatc_a2.pdb
rotate_mol -b gatc_b.pdb gatc_b2.pdb
rotate_mol -b dna18.pdb dna182.pdb
б) для получения изображений:
pdb2img -c gatc_a.pdb gatc_a.ps
pdb2img -c gatc_a2.pdb gatc_a2.ps
pdb2img -c gatc_b.pdb gatc_b.ps
pdb2img -c gatc2_b2.pdb gatc_b2.ps
pdb2img -c dna18.pdb dna18.ps
pdb2img -c dna182.pdb dna182.ps
Затем изображения были конверированы в формат JPEG:
А-форма ДНК с торца: