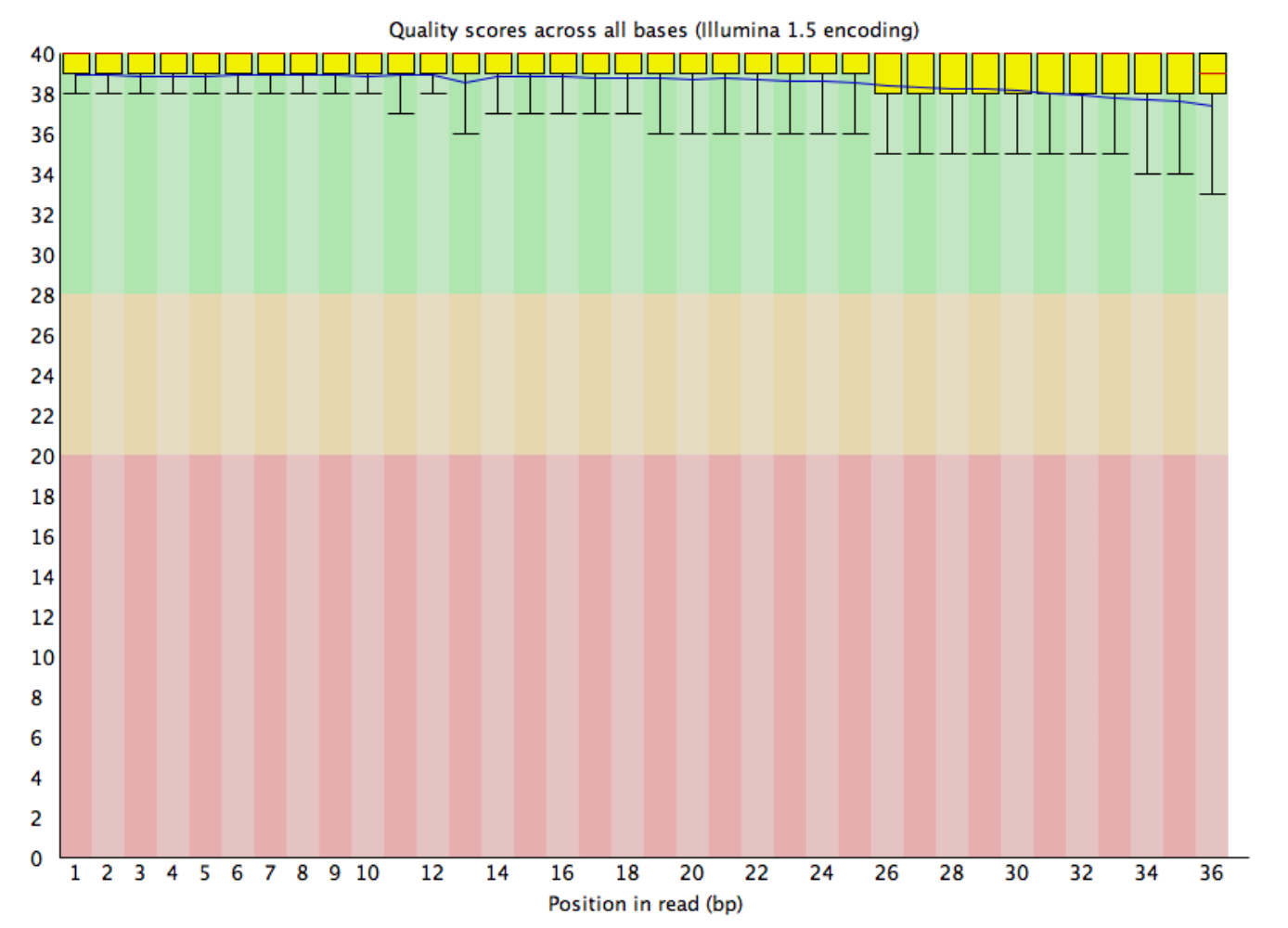
Работа велась с файлом chipseq_chunk30.fastq, содержащим риды Illumina, полученные в ходе ChIP-seq эксперимента. Контроль качества прочтений был осуществлен с помощью программы fastQC. Результат работы программы представлен на рис.1 В данном случае в очистке чтений не было необходимости.
Риды были откартированы на проиндексированный геном человека
при помощи алгоритма BWA-MEM. Анализ картирования
проводился при помощи samtools:
view для перевода в бинарный формат,
sort для сортировки выравнивания по координате начала рида в референсе,
index для индексирования,
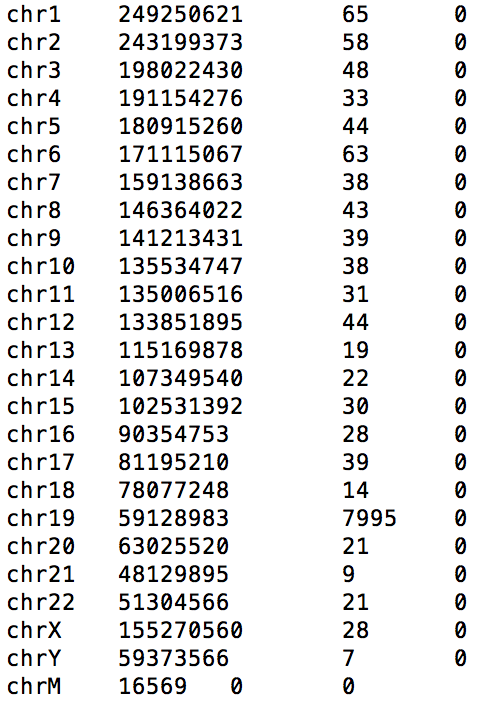
idxstats для получения информации о количестве прочтений на каждой из хромосом.
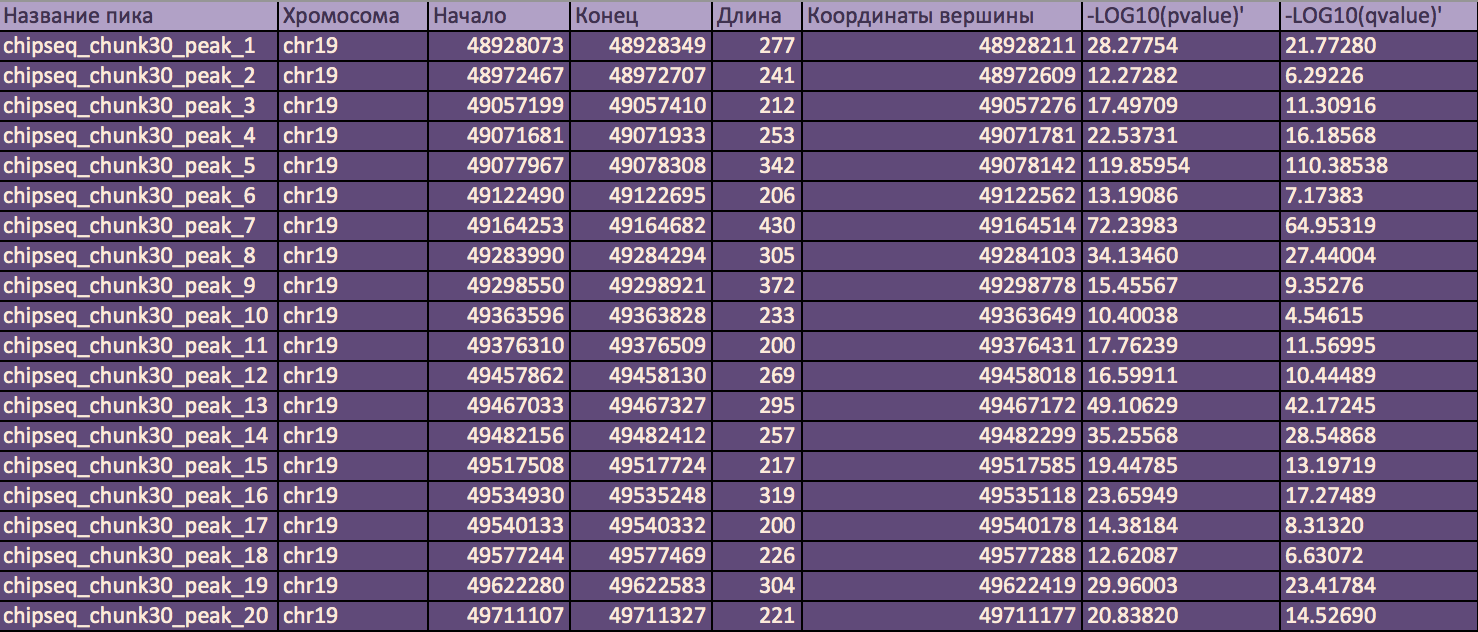
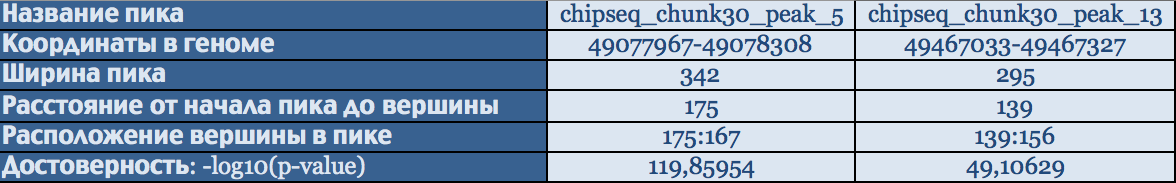
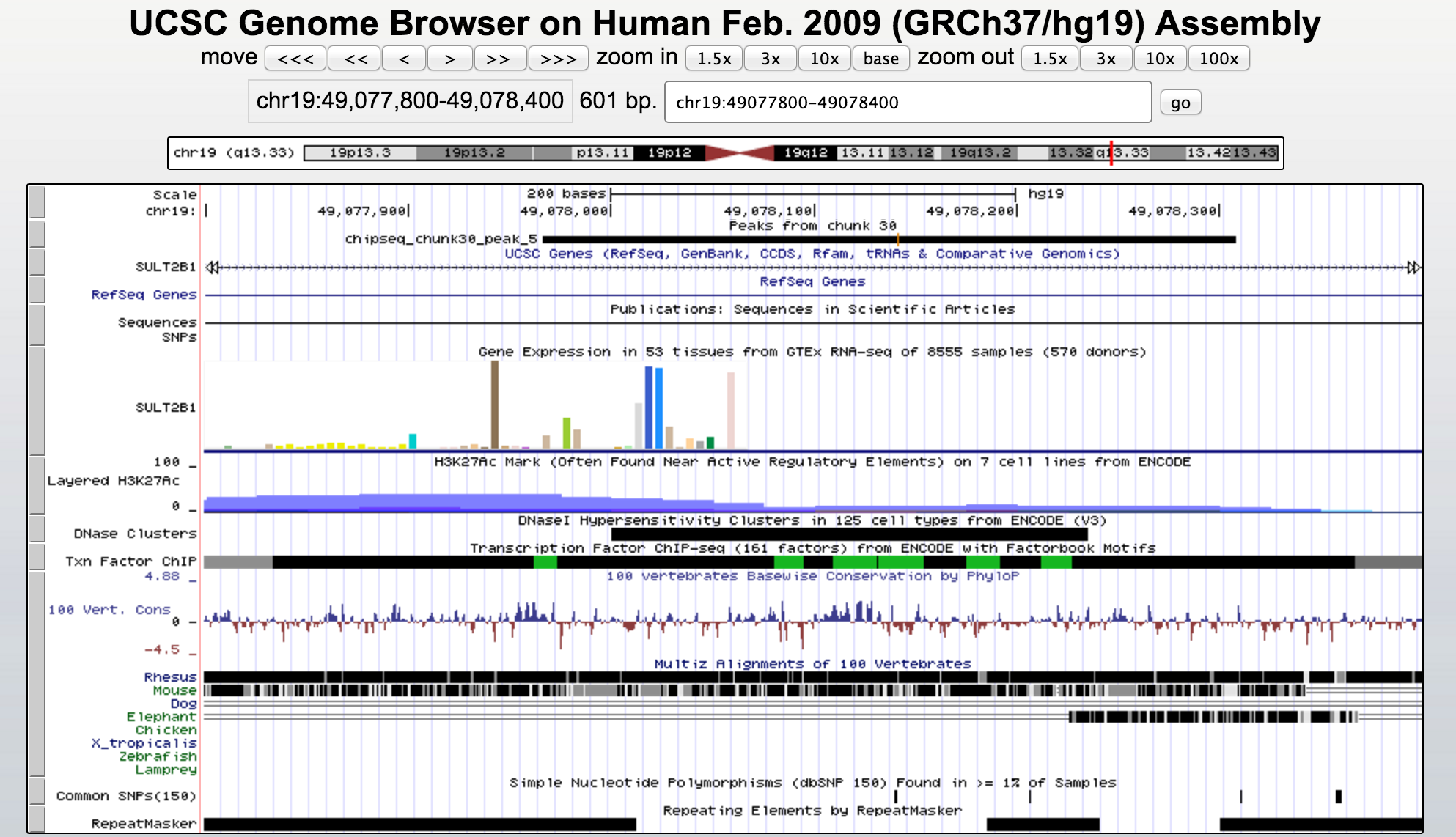
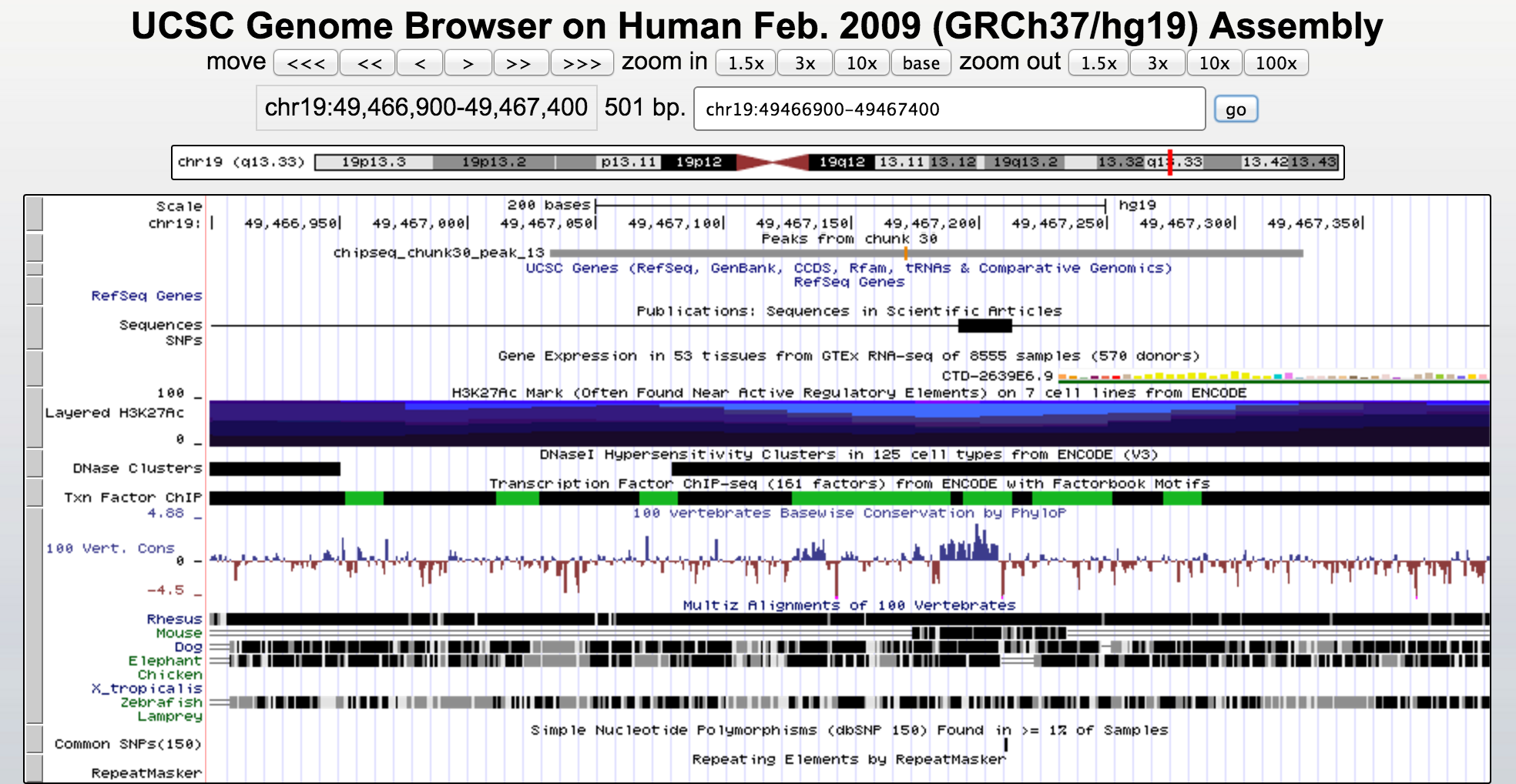
Визуализация пиков проводилась в геномном браузере UCSC Genome Browser.
Для того чтобы использовать файл [narrowPeaks] в качестве трэк-файла,
в него были добавлены следующие строки:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 30"
browser position chr19:48900000-49800000
На рис. 4 приведен соответствующий скриншот из браузера.