Поиск гомологов производился следующим образом:
Файлы из директории P:\y15\term4\Proteomes, содержащие полные протеомы (база UniProt),
были объединены в обин файл (cat file1 >> file2). Далее на основе объединенного файла протеомов была создана база данных,
по которой производился поиск гомологов:
makeblastdb -in proteoms.fasta -dbtype prot -out dbprot.fasta
blastp -query CLPX_BACSU.fasta -evalue 0.001 -db dbprot.fasta -out result
Были найдены 36 достоверных (E-value < 1e-3) гомологов белка CLPX_BACSU. В таблице указаны идентификаторы белков, их функции, а также значения Bit-Score и E-value полученных выравниваний. Sequences producing significant alignments
| ID | Function | Score (Bits) | E-Value |
| CLPX_BACSU | ATP-dependent Clp protease ATP-binding subunit ClpX | 840 | 0 |
| CLPX_GEOKA | ATP-dependent Clp protease ATP-binding subunit ClpX | 712 | 0 |
| A0A0H3GBK3_LISM4 | ATP-dependent Clp protease ATP-binding subunit ClpX | 683 | 0 |
| CLPX_CLOBH | ATP-dependent Clp protease ATP-binding subunit ClpX | 593 | 0 |
| CLPX_ENTFA | ATP-dependent Clp protease ATP-binding subunit ClpX | 580 | 0 |
| Q5FKR6_LACAC | ATP-dependent Clp protease ATP-binding subunit ClpX | 548 | 0 |
| CLPX_STRP1 | ATP-dependent Clp protease ATP-binding subunit ClpX | 547 | 0 |
| CLPX_STRPN | ATP-dependent Clp protease ATP-binding subunit ClpX | 541 | 0 |
| HSLU_LACAC | ATP-dependent protease ATPase subunit HslU | 104 | 1,00E-24 |
| HSLU_GEOKA | ATP-dependent protease ATPase subunit HslU | 101 | 1,00E-23 |
| HSLU_ENTFA | ATP-dependent protease ATPase subunit HslU | 100 | 2,00E-23 |
| CLPY_BACSU | ATP-dependent protease ATPase subunit ClpY | 100 | 4,00E-23 |
| A0A0H3GFY3_LISM4 | ATP-dependent protease ATPase subunit HslU | 97,8 | 2,00E-22 |
| Q5L436_GEOKA | ATP-dependent Clp protease ATPase subunit | 65,9 | 1,00E-11 |
| A0A0H2USJ7_STRPN | ATP-dependent Clp protease, ATP-binding subunit | 59,3 | 1,00E-09 |
| CLPC_BACSU | Negative regulator of genetic competence ClpC/MecB | 56,6 | 1,00E-08 |
| CLPE_BACSU | ATP-dependent Clp protease ATP-binding subunit ClpE | 54,7 | 3,00E-08 |
| A0A0H3GCZ0_LISM4 | ATP-dependent Clp protease ATP-binding subunit ClpC | 52,4 | 2,00E-07 |
| A0A0H3GFJ6_LISM4 | ATP-dependent Clp protease ATP-binding subunit ClpE | 51,6 | 3,00E-07 |
| A5I7N6_CLOBH | Negative regulator of genetic competence | 51,2 | 5,00E-07 |
| A5I766_CLOBH | ATP-dependent zinc metalloprotease FtsH | 49,3 | 1,00E-06 |
| A0A0H2UNL3_STRPN | Putative ATP-dependent Clp protease, ATP-binding subunit | 47 | 9,00E-06 |
| Q5FM98_LACAC | ATPase | 47 | 1,00E-05 |
| Q99XR9_STRP1 | Putative endopeptidase Clp ATP-binding chain C | 46,2 | 2,00E-05 |
| Q5FHW6_LACAC | ATP-dependent protease | 45,8 | 2,00E-05 |
| Q9A200_STRP1 | ATP-dependent zinc metalloprotease FtsH | 45,1 | 3,00E-05 |
| Q82YZ7_ENTFA | ATP-dependent Clp protease, ATP-binding subunit ClpC | 45,4 | 3,00E-05 |
| FTSH_STRPN | ATP-dependent zinc metalloprotease FtsH | 45,1 | 4,00E-05 |
| A5I7Q0_CLOBH | ATP-dependent zinc metalloprotease FtsH | 43,5 | 1,00E-04 |
| Q837W9_ENTFA | ATP-dependent Clp protease, ATP-binding subunit ClpE | 42,4 | 2,00E-04 |
| Q5L3T1_GEOKA | ATP-dependent zinc metalloprotease FtsH | 42 | 3,00E-04 |
| A0A0H3G8Q4_LISM4 | ATP-dependent zinc metalloprotease FtsH | 41,6 | 5,00E-04 |
| A0A0H3GH38_LISM4 | Holliday junction ATP-dependent DNA helicase RuvB | 40,8 | 5,00E-04 |
| Q839B1_ENTFA | ATP-dependent zinc metalloprotease FtsH | 40,8 | 7,00E-04 |
| Q5FMA3_LACAC | ATP-dependent zinc metalloprotease FtsH | 40,8 | 7,00E-04 |
| Q5FLA7_LACAC | ATP-dependent Clp protease, ATP-binding subunit | 40,8 | 7,00E-04 |
Ортологи - два гомологичных белка из разных организмов, разделение общего предка которых на линии, ведущие к ним, произошло в результате видообразования.
Паралоги - два гомологичных белка из одного организма.
Дерево найденных гомологов с обозначенными эволюционными событиями.
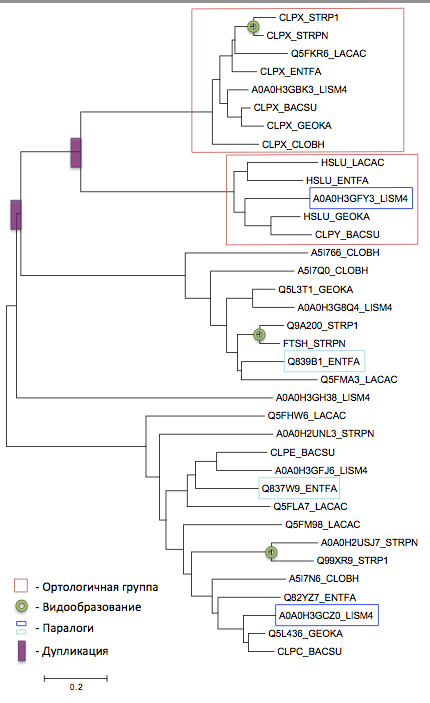 .
.