| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
BLAST
Поиск гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ
С помощью программы BLAST на сайте NCBI, я нашла гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ. В поле для запроса я ввела fasta-последовательность моего белка, поиск проводился в базе данных Uniprot/SwissProt с использованием алгоритма blastp (protein-protein BLAST). Всего было найдено 64 последовательности со значениями E-value от 1e-32 до 9.7. Информация о первых 13-ти находках представлена на рис. 1.
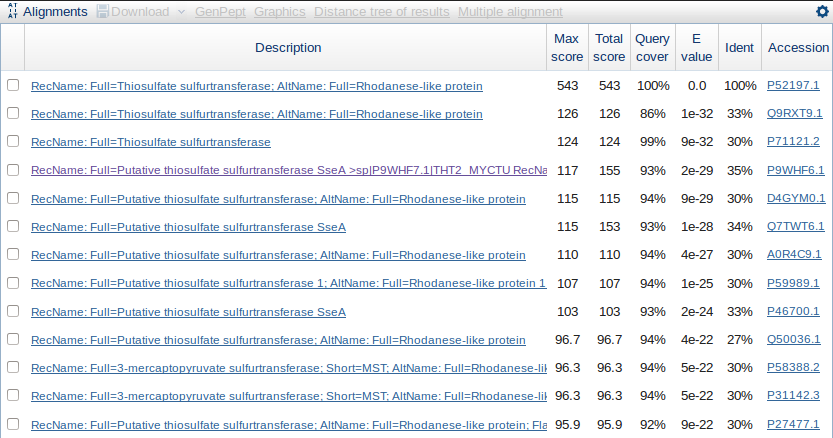
Рис. 1. Фрагмент выходной таблицы при поиске гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ в Uniprot/SwissProt при помощи BLAST.
Выходная таблица содержит следующие колонки:
- Description: название и краткое описание находки.
- Max score: наибольший вес выравнивания участка последовательности, в большинстве случаев равен Total score, но может и отличаться, если в последовательности находки найдено несколько участков совпадения с нашей последовательностью.
- Total score: общий вес выравнивания находки и исходной последовательности.
- Query cover: процент аминокислотных остатков исходной последовательности, покрытых последовательностью находки в выравнивании.
- E value: математическое ожидание находки с тем же и большим весом в банке случайных последовательностей; характеризует то, насколько случайно совпадение последовательности находки с исходной последовательностью, чем меньше E-value, тем ошибка менее вероятна.
- Ident: процент совпадающих аминокислот.
- Accession: идентификатор последовательности находки.
Первая последовательность со значением E-value 0.0 - это сама последовательность моего белка. Для описания я выбрала четвертую находку (белок Putative thiosulfate sulfurtransferase SseA), т. к. у неё достаточно небольшой E-value (2e-29), процент совпадающих аминокислот больше, чем у второй и у третьей находки и хотя Max score немного меньше (117), зато он не совпадает с общим весом выравнивания (155), т. е. в находке обнаружилось больше одного совпадения с исходной последовательностью. Покрытие последовательностью находки исходной последовательности составляет 93% (что меньше 100%, как и требовалось в задании). Параметры выбранного гомолога, содержащиеся в выходной таблице, также приведены в таблице 1.
Таблица 1. Параметры выбранного гомолога белка Rhodanese бактерии Azotobacter vinelandii DJ| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| RecName: Full=Putative thiosulfate sulfurtransferase SseA >sp|P9WHF7.1|THT2_MYCTU RecName: Full=Putative thiosulfate sulfurtransferase SseA | 117 | 155 | 93% | 2e-29 | 35% | P9WHF6.1 |
Выравнивания двух участков находки с исходной последовательнотью и некоторая дополнительная информация показана на рис. 2.
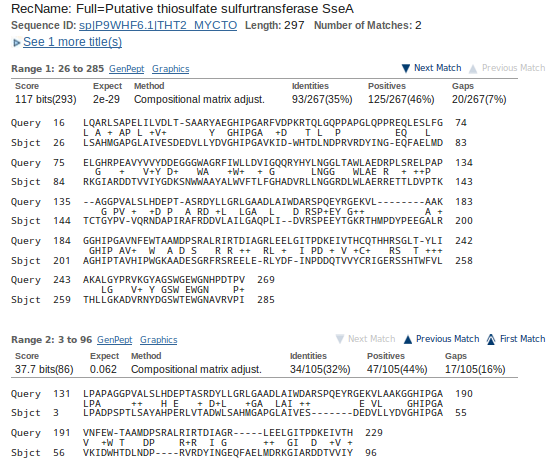
Рис. 2. Информация о выравниваниях найденной последовательности с исходной последовательностью.
Можно заметить, что информация о находке, приведенная в таблице 1, касается в основном только первого участка совпадения, как наилучшего, у второго участка параметры совсем другие:
- Score: 37.7
- E value: 0.062
- Ident: 32%
Карта локального сходства между query и выбранной находкой
Для построения карты локального сходства я отметила галочкой Align two or more sequences в интерфейсе запуска BLAST и ввела в поля последовательности исходного и найденного белка. В окне с результатом я открыла DotMatrix и сохранила картинку с картой локального сходства. Полученное изображение можно увидеть на рис. 3.
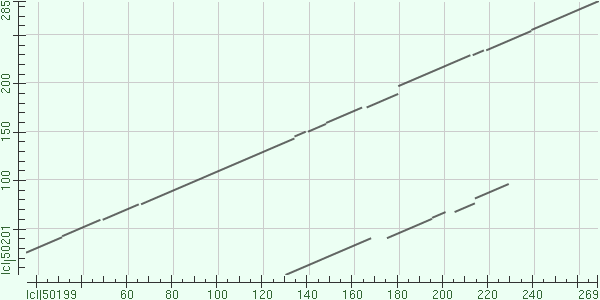
Рис. 3. Карта локального сходства белка Rhodanese бактерии Azotobacter vinelandii DJ и белка Putative thiosulfate sulfurtransferase SseA бактерии Mycobacterium tuberculosis CDC1551
По оси абсцисс находится нумеровка аминокислотных остатков исходного белка, по оси ординат - белка находки. На карте видно две прерывающиеся прямые. Более длинная соответствует первому гомологичному участку (с 26 по 285 аминокислоту исходной последовательности) в найденной последовательности, разрывов, соответствующих гэпам в выравнивании, достаточно много, но они небольшие. Маловероятно, что такой большой участок последовательностей оказался сходным по случайности, поэтому в гомологичности данного фрагмента сомневаться не приходится. Прямая, соответсвующая второму совпадающему участку (с 3 по 96 аминокислоту), намного короче, разрывы в ней немного значительнее (особенно первый разрыв). Хоть этот участок и является сходным, вполне возможно, что он не является гомологичным, т. к. мог совпасть по случайности (к тому же E-value данного участка равно 0.062, что больше, чем 0.001).
Поиск эукариотических гомологов последовательности белка белка Rhodanese бактерии Azotobacter vinelandii DJ в Uniprot/SwissProt
Для поиска эукариотических гомологов моего белка при составлении запроса в BLAST в поле я ввела "Organizm Eukaryota (taxid:2759)". В результате было найдено 26 последовательностей, из которых значение E-value меньше 0.001 имеют 14 находок. Самый лучший гомолог имеет значение E-value 3e-20, самый последний - 4e-05. Процент совпадения аминокислотных остатков с исходной последовательностью варьирует в пределах 24-29%. Фрагмент выходной таблицы запроса представлен на рис. 4.
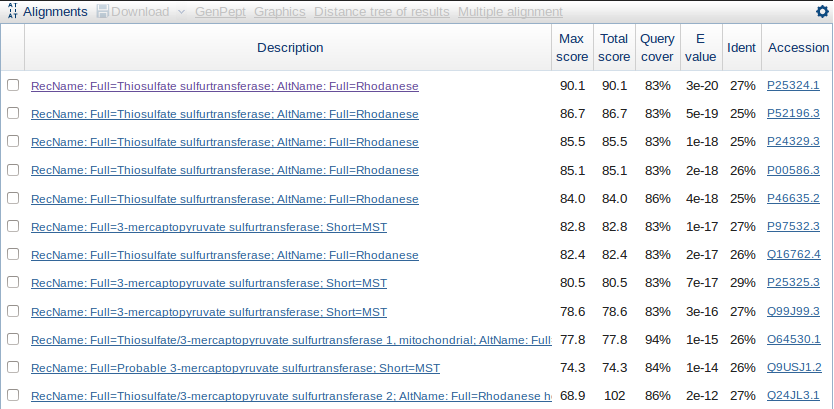
Рис. 4. Фрагмент выходной таблицы при поиске эукариотических гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ в Uniprot/SwissProt при помощи BLAST.
Для построения множественного выравнивания я выбрала первые 9 находок (отметив их галочками), прошла по ссылке Multiple alignment, в открывшемся окне нажала на Download и выбрала Fasta plus gaps. В результате я получила следующий файл, содержащий множественное выравнивание выбранных белков в fasta-формате. Наглядное представление данного выравнивания показано на рис. 5. Проект данного выравнивания в формате .jar можно скачать здесь.

Рис. 5 Множественное выравнивание первых девяти гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ, найденных в Uniprot/SwissProt при помощи BLAST. Раскраска ClustalX, консервативность 70%.
Длина полученного выравнивания составляет 298 аминокислотных остатков. В данном выравнивании было найдено 133 консервативные позиции (что составляет 44,6% от длины выравнивания) и 175 функционально консервативных позиций (58,7% от длины выравнивания). Эти цифры достаточно большие, особенно для множественного выравнивания, поэтому с высокой долей вероятности можно считать данные последовательности гомологичными.
Поиск эукариотических гомологов последовательности белка белка Rhodanese бактерии Azotobacter vinelandii DJ в Refseq
Для поиска эукариотических гомологов моего белка в базе данных Refseq я выполнила аналогичные действия, что и при поиске гомологов в Swissprot, поменяв в графе Database Uniprot/SwissProt на Reference proteins. В результате было найдено 100 последовательностей, притом самая первая имеет крайне низкое значение E-value (4e-132) и высокий процент сходства с исходной последовательностью (74%). Остальные находки имеют значение E-value от 2e-24 до 1e-16, идентичность с исходным белком варьирует в пределах 25-40%. В целом можно сказать, что по сравнению с поиском в Swissprot, количество находок увеличилось (что естественно, т. к. в Refseq в принципе больше белков), значения E-value оказались немного ниже, а identity - немного выше. Но вряд ли эти различия можно считать значительными, среди находок в Refseq и Swissprot примерно с одинаковой вероятностью могут оказаться истинные гомологи исходного белка. Фрагмент выходной таблицы запроса представлен на рис. 6.
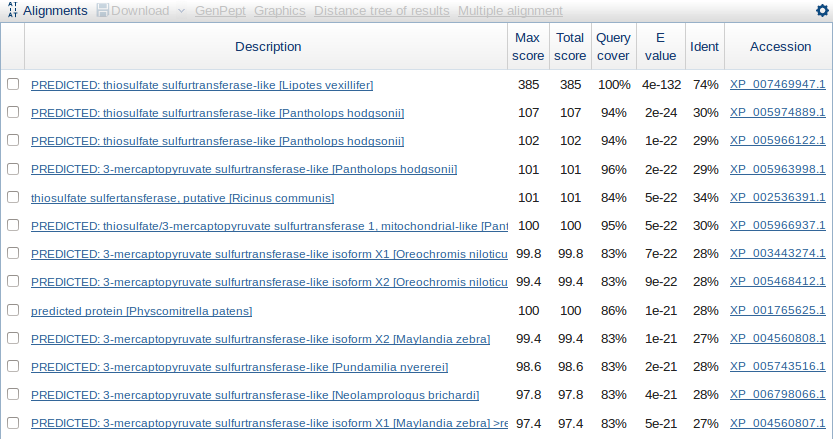
Рис. 6. Фрагмент выходной таблицы при поиске эукариотических гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ в Refseq при помощи BLAST.
Как и в прошлом задании, для построения множественного выравнивания я выбрала первые 9 находок. Файл с выравниванием в fasta-формате можно скачать здесь. Наглядное представление множественного выравнивания показано на рис. 7. Проект данного выравнивания в формате .jar здесь.

Рис. 7 Множественное выравнивание первых девяти гомологов белка Rhodanese бактерии Azotobacter vinelandii DJ, найденных в Refseq при помощи BLAST. Раскраска ClustalX, консервативность 70%.
Длина полученного выравнивания составляет 522 аминокислотных остатка. В данном выравнивании было найдено 27 консервативных позиций (что составляет 5,2% от длины выравнивания) и 56 функционально консервативных позиций (10,7% от длины выравнивания). Данное выравнивание намного хуже, чем аналогичное для белков из Swissprot. Возможно, это связано с тем, что в принципе информация, полученная из базы данных Refseq, не всегда достоверна. Но, скорее всего, главную роль сыграл эффект случайности, т. к. находки должны быть гомологичны исходной последовательности, но не обязательно между собой. Мне просто повезло, что в первом случае я выбрала 9 настолько близких белков. Видимо, в случае с белками из Refseq, мне повезло меньше. К тому же, сам банк Refseq и, соответственно, количество находок в нём больше, чем Swissprot/Uniprot, значит вероятность удачно выбрать гомологичные последовательности для выравнивания была меньше.
©Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016