| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Совмещение полипептидных цепей белков Sulfurtransferase (идентификатор PDB 1E0C) и Thiosulfate sulfurtransferase sseA (идентификатор PDB 3HZU)
С помощью сервиса PDBeFold я нашла белок, похожий на описываемый мной белок Rhodanese бактерии Azotobacter vinelandii. Для поиска была выбрана цепь А моего белка. После ввода индикатора белка, сервер выдал 31 совпадение.
Я выбрала белок Thiosulfate sulfurtransferase sseA (тиосульфат сульфуртрансфераза sseA) бактерии Mycobacterium tuberculosis (микобактерия туберкулёза или палочка Коха, наиболее изученный вид бактерий, среди способных вызывать туберкулёз у человека).
У выбранного мной белка Thiosulfate sulfurtransferase sseA следующие параметры сходства с белком Sulfurtransferase
- процент совпадающих аминокислотных остатков - 33% (соответственно, это принципиально другой белок, а не тот же самый)
- число сопоставленных Сα-атомов моего белка и найденного белка составляет 242/271=89% (меньше 90% но больше 70%)
- мера сходства структур (RMSD) исследуемого и найденного белка равна 1.89 ангстрем (в пределах 0.7 - 2.0)
- число гэпов больше нуля (13)
По всей видимости, данные белки являются белками разных видов одного рода (хотя, на сайте PDB исследуемый белок характеризуется как Sulfurtransferase, а найденный как Transferase, т. е. можно предположить, что белок сульфуртрансферазы бактерии Azotobacter vinelandii принадлежит к более мелкой систематической единице.
Судя по названию, оба белка связаны с транспортом серы, предположительно, они имеют сходные функции.
Чтобы проиллюстрировать совмещение цепей белков я выбрала участок 83-155 белка сульфуртрансферазы и участок 92-166 тиосульфат сульфуртрансферазы. На рис. 1. показано совмещение данных участков в структуре белков (выбранные участки выделены голубым, остальной белок - белым). Выбранный участок каждой из двух совмещенных цепей содержит 3 альфа-спирали и 3 тяжа бета-листов.
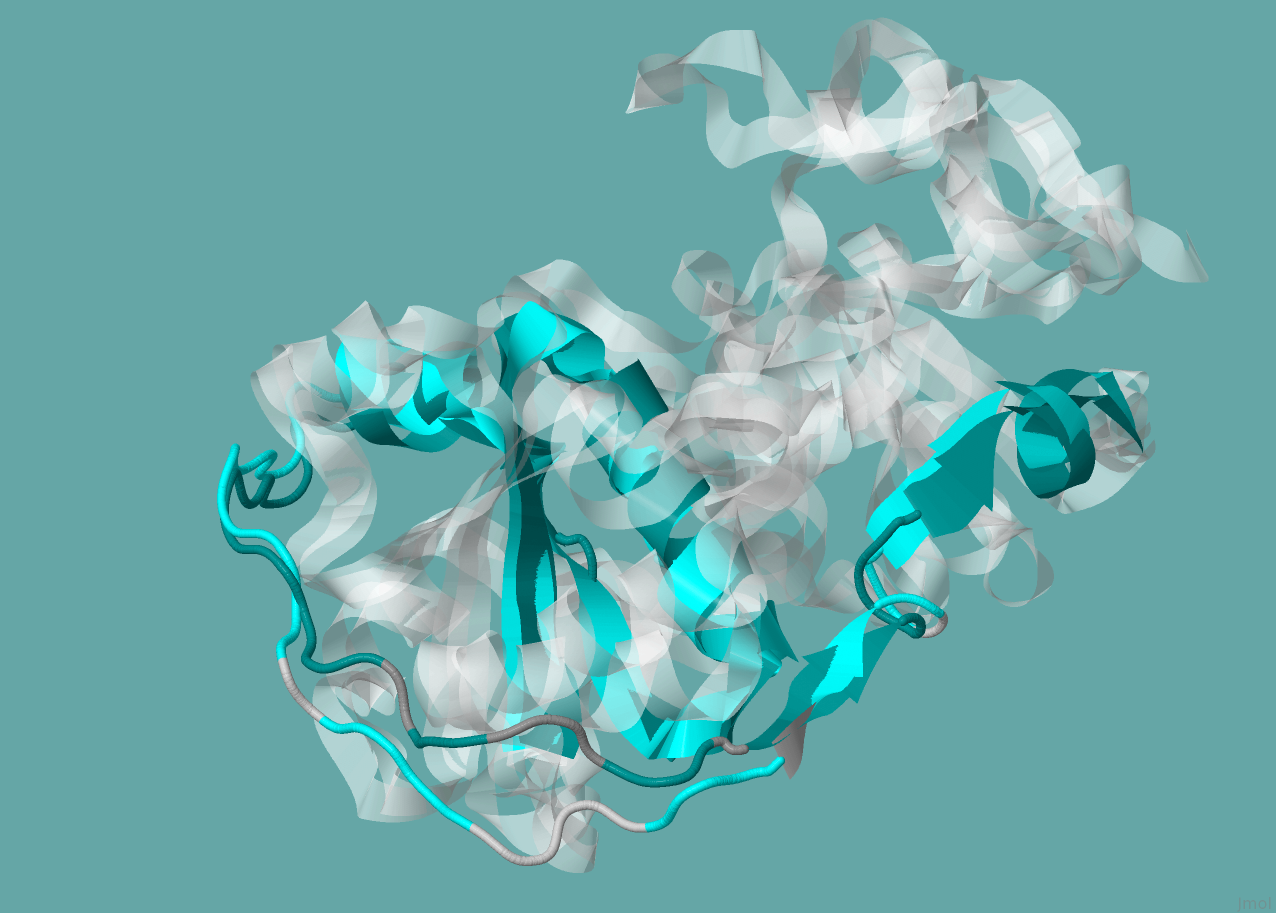
Рис. 1. Выбранный участок цепей белков Sulfurtransferase (идентификатор PDB 1E0C) и Thiosulfate sulfurtransferase sseA (идентификатор PDB 3HZU) в общей структуре данных белков
Для выделения геометрического ядра (т. е. наиболее хорошо совмещаемых пар Сα-атомов) я использовала следующие команды Jmol (был взят порог расстояния между атомами - 2Å):
define ch1 (83-155) and :A
define ch2 (92-166) and :B
select (within(GROUP,within(2, (ch2 and *.CA)) and (ch1 and *.CA))) and *.CA
select (within(GROUP,within(2, (ch1 and *.CA)) and (ch2 and *.CA))) and *.CA
Геометрическое ядро рассматриваемого участка совмещения составляет 65 пар Сα-атомов.
На рис. 2. детально показано совмещение цепей данных участков.
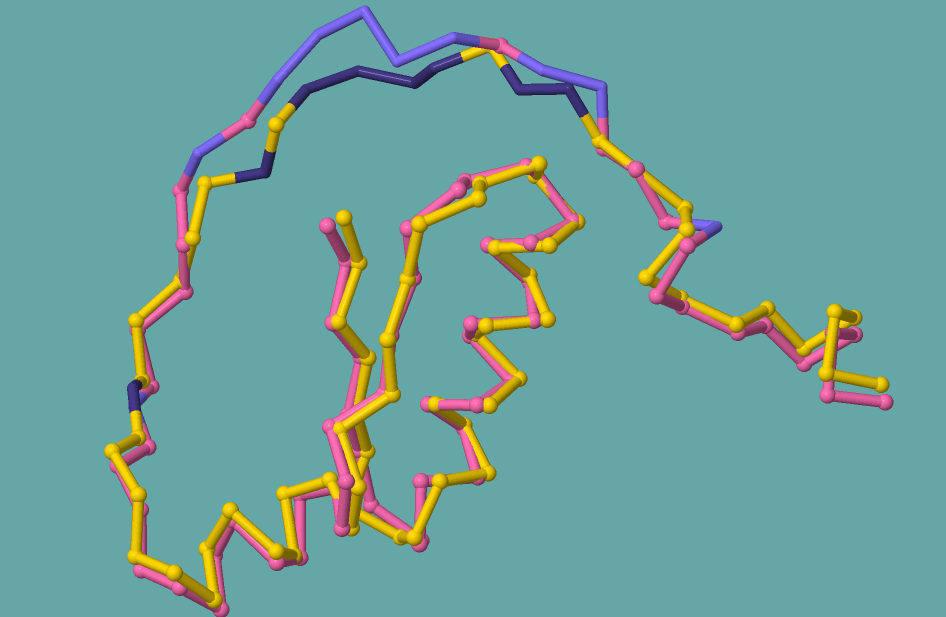
Рис. 2. Совмещение участков цепей белков Sulfurtransferase (идентификатор PDB 1E0C) и Thiosulfate sulfurtransferase sseA (идентификатор PDB 3HZU). Желтым цветом показана цепь исходного белка, розовым - найденного. Шариками выделены Сα-атомы геометрического ядра. Участки расхождения (гэпы) на цепи белка Sulfurtransferase показаны темно-синим цветом, на цепи белка Thiosulfate sulfurtransferase sseA - голубым цветом.
С помощью расширенного поиска PDB я нашла 32 структуры, схожие с белком Sulfurtransferase Azotobacter Vinelandii. При поиске я использовала следующие критерии:
- количество цепей: 1 (Number of Chains Search : Min Number of Chains=1 Max Number of Chains=1)
- организм, из которого получен белок относится к порядку Pseudomonadales (TaxonomyTree Search for Pseudomonadales)
- белок относится к трансферазам (EnzymeClassificationTree Search for 2: Transferases)
- Метод расшифровки - рентгеноструктурный анализ с разрешением 1.5 ангстрем и больше (Resolution is 1.5 and more)
- есть лиганды (Has free ligands=yes)
Таблицу в результатами поиска можно посмотреть здесь.
©Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016