| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Построение множественного выравнивания. Pfam
При помощи программ BLAST и PSI-BLAST я выбрала 8 гомологов белка Sulfurtransferase бактерии Azotobacter Vinelandii, поиск выполнялся в базе данных Refseq, т. к. в Swissprot/Uniprot не нашлось белков с ID = 50-60% (идентичность основной массы находок была в пределах от 20% до 40%). Однако, покрытие выбранных мной белков очень близко к 100% (находок с покрытием 70-90% и при этом с хорошими значениями ID и E-value не было). Файл с последовательностями выбранных белков можно скачать здесь.
Далее я построила множественное выравнивание данных последовательностей с помощью программы muscle на сервере kodomo. Для этого я использовала команду: "muscle -in seq.fasta -out alignment_muscle.fasta". В результате я получила следующий файл с выравниванием моих последовательностей. Наглядное представление данного выравнивания можно увидеть на рис. 1.
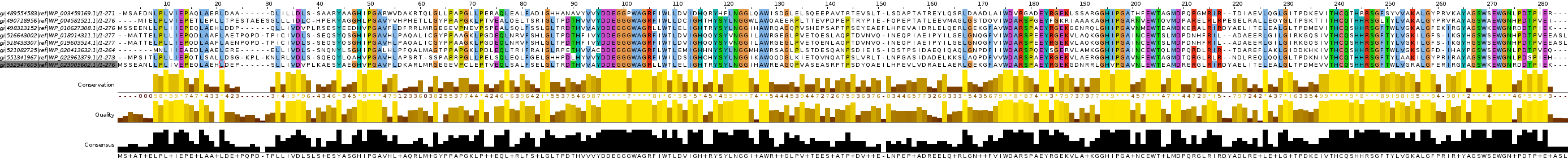
Рис. 1 Множественное выравнивание гомологов белка Sulfurtransferase бактерии Azotobacter vinelandii DJ, найденных в Refseq при помощи BLAST и PSI-BLAST. Получено с помощью muscle. Раскраска ClustalX, консервативность 70%.
Следующим этапом было построение множественного выравнивания этих же последовательностей с помощью программы mafft на сервере kodomo. Использованная команда: "mafft seq.fasta > alignment_mafft.fasta". Полученный fasta-файл можно скачать здесь. Наглядное представление данного выравнивания - на рис. 2.
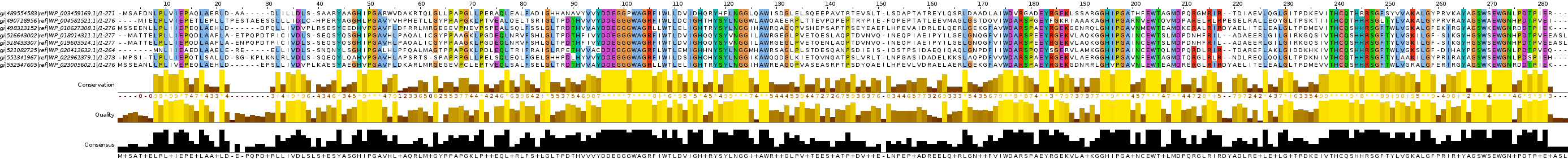
Рис. 2 Множественное выравнивание гомологов белка Sulfurtransferase бактерии Azotobacter vinelandii DJ, найденных в Refseq при помощи BLAST и PSI-BLAST. Получено с помощью mafft. Раскраска ClustalX, консервативность 70%.
Далее я сравнила два полученных выравнивания. Они оказались почти одинаковыми. Длина выравниваний совпадает, различия в расстановке гэпов незначительные и встречаются только в участках, где нет гомологичных блоков, поэтому для сопоставления выравниваний я не использовала никаких специальных программ, а просто скопировала их в одно окно Jalview, причём двигать выравнивания относительно друг друга тоже не потребовалось. Незначительные различия наблюдаются в участках с 2 по 6 позицию, с 23 по 30 и во позициях 61-62. В остальном выравнивания полностью одинаковые. Изображение со сравнением полученных выравниваний можно увидеть на рис. 3.

Рис. 3 Сравнение множественных выравниваний гомологов белка Sulfurtransferase бактерии Azotobacter vinelandii DJ. Сверху выравнивание, полученное с помощью muscle, снизу - с помощью mafft. Раскраска ClustalX, консервативность 70%.
Pfam
Я нашла последовательность моего белка на сайте pfam, использовав идентификатор 1e0c. По данным этого сайта, в последовательности моего белка 2 домена Rhodanese-like domain pfam-семейства Rhodanese (PF00581), один из них имеет координаты 12-123, второй такой же домен находится на участке 150-264. Я сохранила seed-выравнивание этого семейства. Файл с выравниванием можно скачать здесь. Наглядное представление - на рис. 4.

Рис. 4 Seed-выравнивание pfam-семейства Rhodanese (PF00581). Раскраска ClustalX, консервативность 70%.
Построение множественного выравнивания с помощью программы ClustalOmega
Я построила выравнивание гомологов моего белка с помощью программы ClustalOmega, доступной на сайте http://www.ebi.ac.uk/Tools/msa/clustalo/. Файл с выравниванием можно скачать здесь. Наглядное представление данного выравнивания - на рис. 5.

Рис. 5 Выравнивание гомологов белка Sulfurtransferase бактерии Azotobacter vinelandii DJ, полученное с помощью программы ClustalOmega. Раскраска ClustalX, консервативность 70%.
Далее я сравнила выравнивание, полученное с помощью ClustalOmega, с muscle-выравниванием. Длина выравниваний одинаковая, поэтому для их сравнения, как и в предыдущем задании, не потребовалось использование дополнительных программ. Незначительные различия расстановки гэпов наблюдаются в участках, которые не являются гомологичными (позиции 23-30, 61-62, 151-152, 218-220). В остальном выравнивания полностью совпадают. Изображение со сравнением полученных выравниваний можно увидеть на рис. 6.
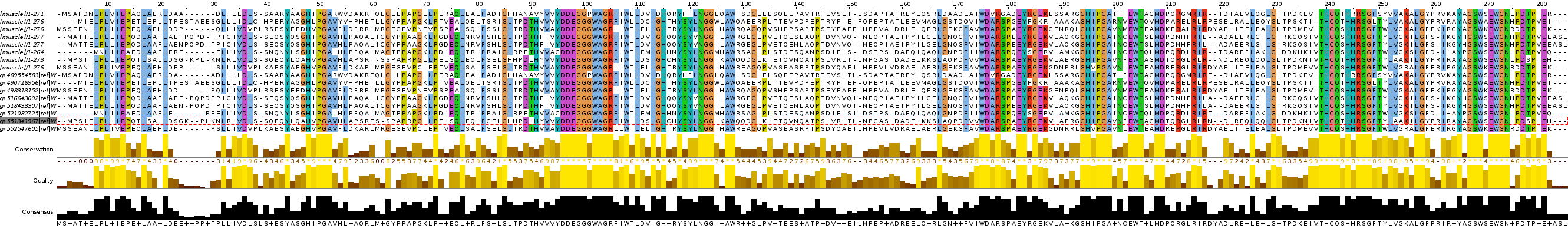
Рис. 6 Сравнение множественных выравниваний гомологов белка Sulfurtransferase бактерии Azotobacter vinelandii DJ. Сверху выравнивание, полученное с помощью muscle, снизу - с помощью ClustalOmega. Раскраска ClustalX, консервативность 70%.
Затем я применила к полученному выравниванию алгоритм оптимизации, реализованный в muscle. Для этого я использовала команду: "muscle -in alignment_clustalo.fasta -out refined_clustalo.fasta -refine". Улучшенное выравнивание можно скачать здесь. Я сравнила это выравнивание с исходным выравниванием, полученным с помощью ClustalOmega и выяснила, что они различаются опять же только в негомологичных участках (участки 23-29, 61-62, 151-152, 218-220). Я заметила, что изменились только участки, на которых выравнивание ClustalOmega отличается от muscle-выравнивания, причём на позициях 61-62, 151-152, 218-220 выравнивание стало полностью идентичным выравниванию muscle. Т. е., возможно, выравнивание muscle является более правильным, хотя, скорее всего, различия в почти негомологичных участках выравнивания не являются значительными. Поэтому все три выравнивания можно считать достаточно хорошими.
©Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016