| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Предсказание генов у прокариот
Предсказания генов проводились для данного контига микробиома кардиального отдела желудка валлаби.
Предсказания генов с помощью ORF Finder
ORF Finder (Open Reading Frame Finder) это сервис, который находит все рамки считывания в запрашиваемой последовательности. Этот сервис был запущен для последовательности данного контига. Использовался бактериальный генетический код (11 Bacterial Code). В результате были получены координаты всех возможных рамок считывания этой последовательности, для каждой из них производился поиск гомологов в Swissprot с помощью Blastp.
В качестве потенциальных генов выбраны последовательности, для которых нашлись более-менее достоверные гомологи (E-value находки меньше 0,001 и не менее 80% банковской последовательности покрыто выравниванием), предпочтение предоставлялось более длинным последовательностям. Информацию о предсказанных генах можно увидеть в таблице 1.
Таблица 1. Информация о генах контига микробиома кардиального отдела желудка валлаби, предсказанных с помощью ORF Finder
| Начало | Конец | Длина в а. о. | Цепь | Описание | Идентификатор гомолога | Организм | Вес выравнивания | Покрытие | E-value | Идентичность |
| 110 | <1 | >36 | - | Белок починки ДНК семейства RadC | WP_026666765.1 | Butyrivibrio sp. AE2005 | 40.8 | 64% | 0.017 | 57% |
| 776 | 171 | 201 | - | Белок устойчивости к алюминию семейства цистатионин бета-лиаз | WP_021922734.1 | Roseburia inulinivorans CAG:15 | 345 | 100% | 1e-114 | 81% |
| 1465 | 773 | 230 | - | Белок устойчивости к алюминию семейства цистатионин бета-лиаз | WP_021922734.1 | Roseburia inulinivorans CAG:15 | 349 | 92% | 1e-115 | 78% |
| 2443 | 1496 | 315 | - | тPHK диметилаллилтрансфераза | WP_022047040.1 | Roseburia sp. CAG:18 | 437 | 99% | 2e-150 | 64% |
| 2736 | 2458 | 92 | - | Белок семейства MutL, устраняющий несоответствия в ДНК | WP_031545457.1 | Lachnospiraceae bacterium AC2014 | 132 | 100% | 5e-34 | 67% |
Все гены, кроме первого, были предсказаны на основе указанных выше условий (соответствующие гомологи имеют хорошее e-value и покрытие и т. д.). Но гомолог первого предсказанного гена (как можно видеть в таблице 1) имеет e-value > 0.001 и покрытие 60% (меньше 80%). Предсказание данного гена производилось на основе выравниваний найденных гомологов. Покрытие составляет только 60%, т. к. скорее всего конец гена находится за пределами контига, поэтому с найденной открытой рамкой выравнивались только "начальные" участки последовательностей гомологов. Старт гена смещен с 164-й позиции на 110-ю (т. к. все выравнивания гомологов начинались с аминокислоты (D), следующей за метионином, кодируемым триплетом atg, начинающимся на 110-ой позиции).
Предсказания генов с помощью GeneMark
Предсказания генов того же контига произведены с помощью программы GeneMark с эвристическими параметрами 1999 года (Heuristic parameters, as in 1999 publication). В результате получен файл, содержащий координаты предсказанных генов и pdf-файл, содержащий график кодирующего потенциала. Полученный график можно увидеть на рис. 1. Информацию о предсказанных генах можно найти в таблице 2.
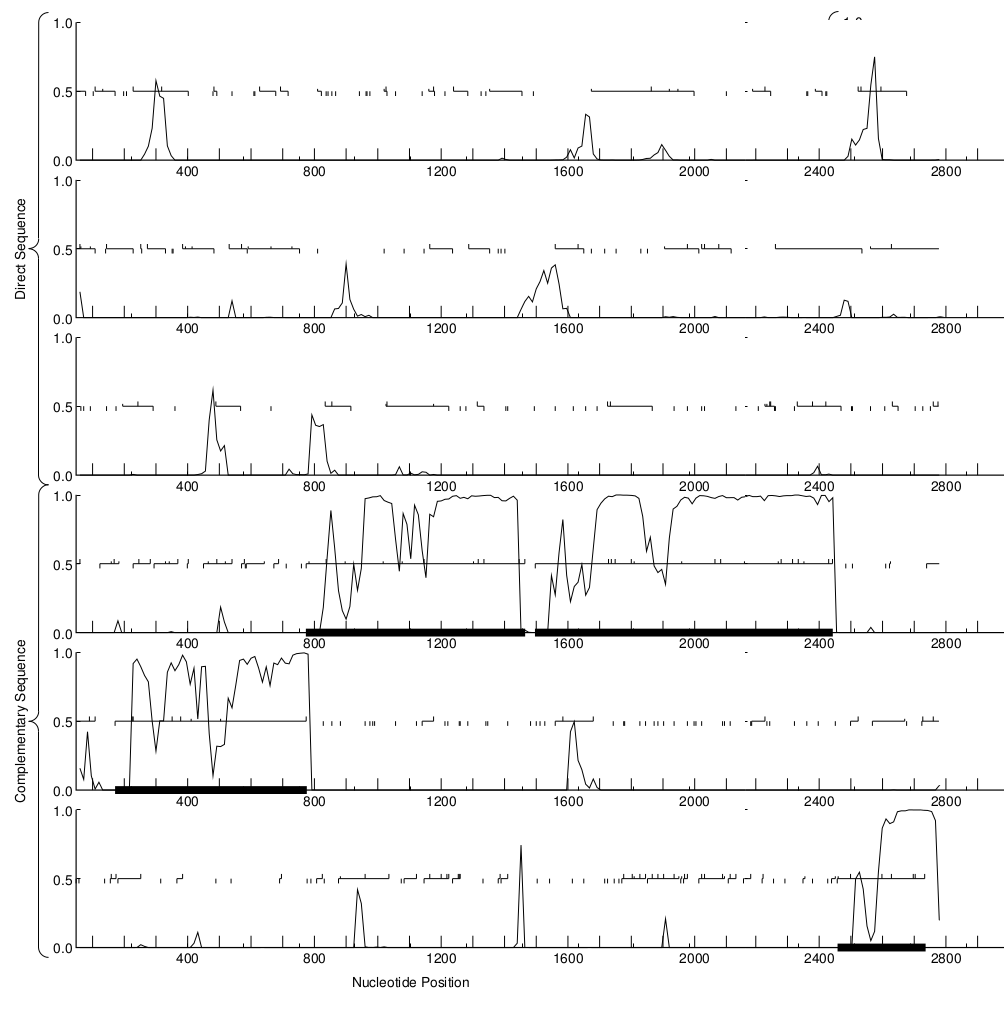
Рис. 1. График кодирующего потенциала контига микробиома кардиального отдела желудка валлаби. Получен с помощью программы GeneMark с эвристическими параметрами 1999 года (Heuristic parameters, as in 1999 publication)
Таблица 2. Информация о генах контига, предсказанных с помощью GeneMark с эвристическими параметрами 1999 года
| Начало | Конец | Длина в а. о. | Цепь |
| 776 | 171 | 201 | - |
| 1465 | 773 | 230 | - |
| 2443 | 1496 | 315 | - |
| 2736 | 2458 | 92 | - |
Сравнив данные, представленные в таблицах 1 и 2, можно увидеть, что предсказания, полученные с помощью GeneMark сходятся в предсказаниями ORF Finder за одним исключением - GeneMark не предсказал ген, конец которого находится за пределами контига.
Схематическое изображение расположения генов на комплементарной цепи исследуемого контига:
3'-----[<=110]-[171<=776}{773<=1465]-[1496<=2443]-[2458<=2736]-----5'Синим отмечены гены, одинаково предсказанные GeneMark и ORF Finder, красным отмечен ген, предсказанный только с помощью ORF Finder. Фигурными скобками показаны перекрывающиеся участки генов.
Программа GeneMark была запущена повторно, но на это раз с эвристическими параметрами 2010 года (Heuristic (MetaGeneMark) parameters, as in 2010 publication). В результате получено 2 файла: файл, содержащий координаты предсказанных генов и pdf-файл, содержащий график кодирующего потенциала. Полученный график можно увидеть на рис. 2. Информацию о предсказанных генах можно найти в таблице 3.
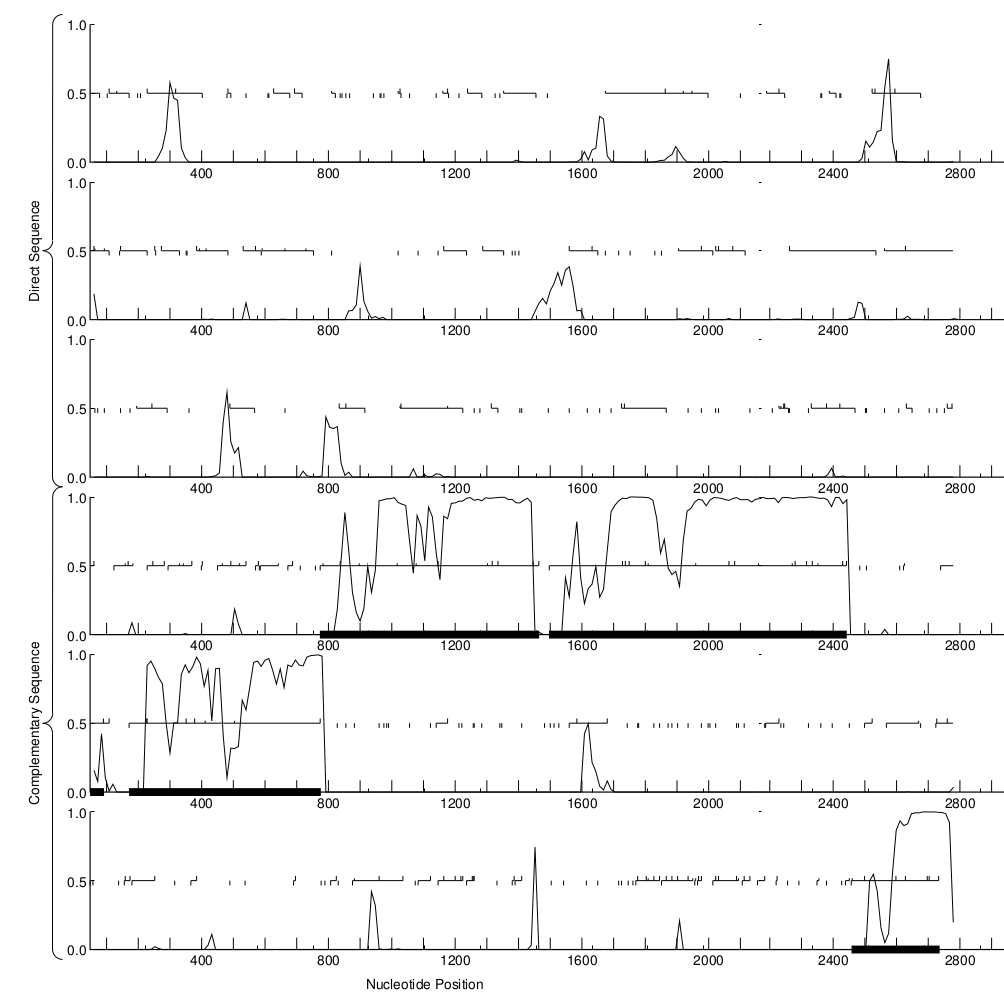
Рис. 2. График кодирующего потенциала контига микробиома кардиального отдела желудка валлаби. Получен с помощью программы GeneMark с эвристическими параметрами 2010 года (Heuristic (MetaGeneMark) parameters, as in 2010 publication)
Таблица 3. Информация о генах контига, предсказанных с помощью GeneMark с эвристическими параметрами 2010 года
| Начало | Конец | Длина в а. о. | Цепь |
| 92 | <3 | >30 | - |
| 776 | 171 | 201 | - |
| 1465 | 773 | 230 | - |
| 2443 | 1496 | 315 | - |
| 2736 | 2458 | 92 | - |
Можно видеть, что по сравнению с прошлым запуском GeneMark почти ничего не поменялось. Координаты предсказанных генов совершенно одинаковые. Единственное различие - GeneMark с эвристическими параметрами 2010 года предсказал тот самый ген, конец которого находится за пределами контига. Правда, началом этого гена он посчитал 92-ю позицию (в отличие от выбранной нами при работе с ORF Finder 110-ой позиции). Следовательно, раз GeneMark с новыми эвристическими параметрами смог предсказать ген, не найденный при предыдущем запуске, можно считать, что эвристические параметры 2010 года более чувствительные и запуск GeneMark с этими параметрами, вероятно, даёт более точные результаты.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016