| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
A- и В- формы ДНК. Структура РНК
С помощью программы fiber пакета 3DNA построены A-, B- и Z-формы дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc". Структура дуплекса в А-форме сохранена в файле gatc-a.pdb, структура дуплекса в В-форме - в файле gatc-b.pdb, структура дуплекса в Z-форме - в файле gatc-z.pdb.
Затем с помощью программы Jmol были получены изображения А-формы дуплекса ДНК. Они приведены на рис. 1 и рис. 2.
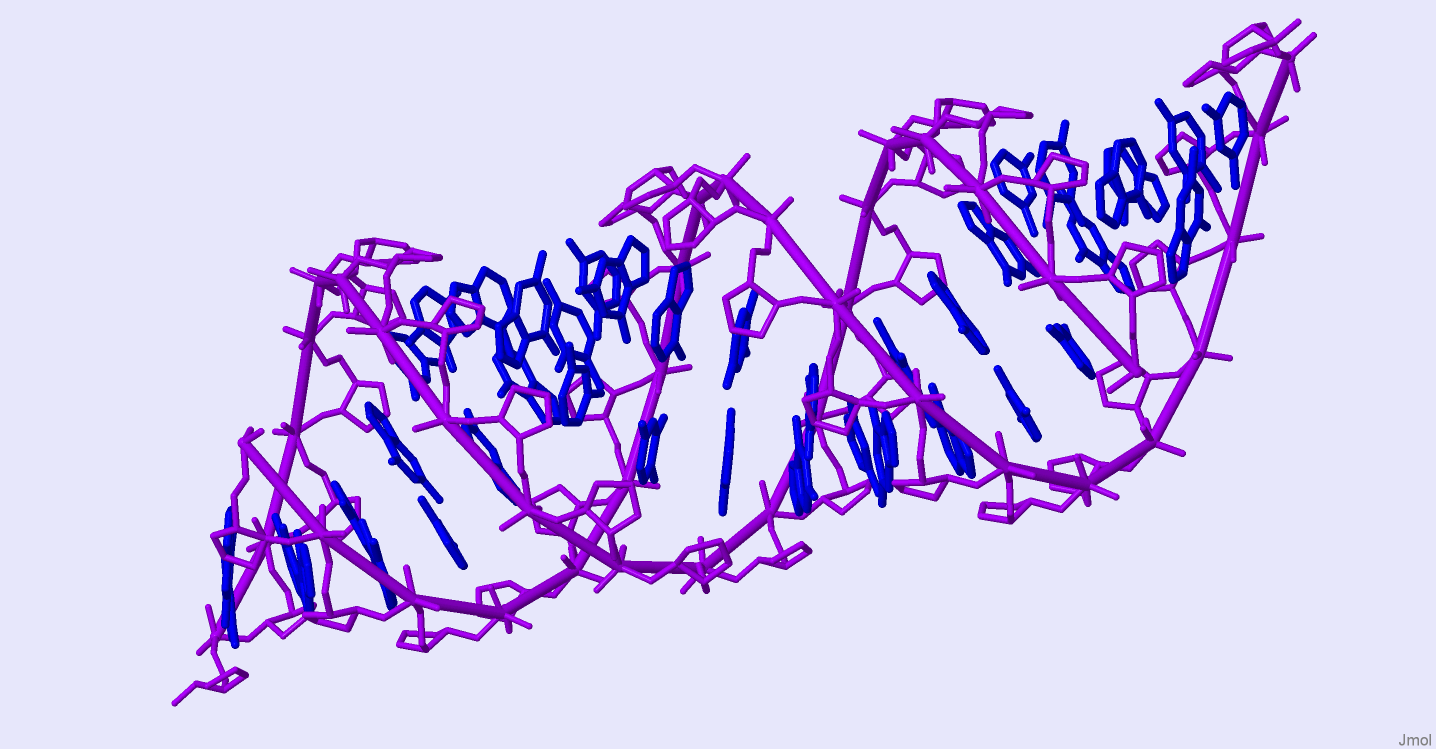
Рис. 1. Структура А-формы дуплекса ДНК. Фиолетовым выделен сахарофосфатный остов ДНК, синим выделены нуклеотиды. Изображение получено с помощью программы Jmol.
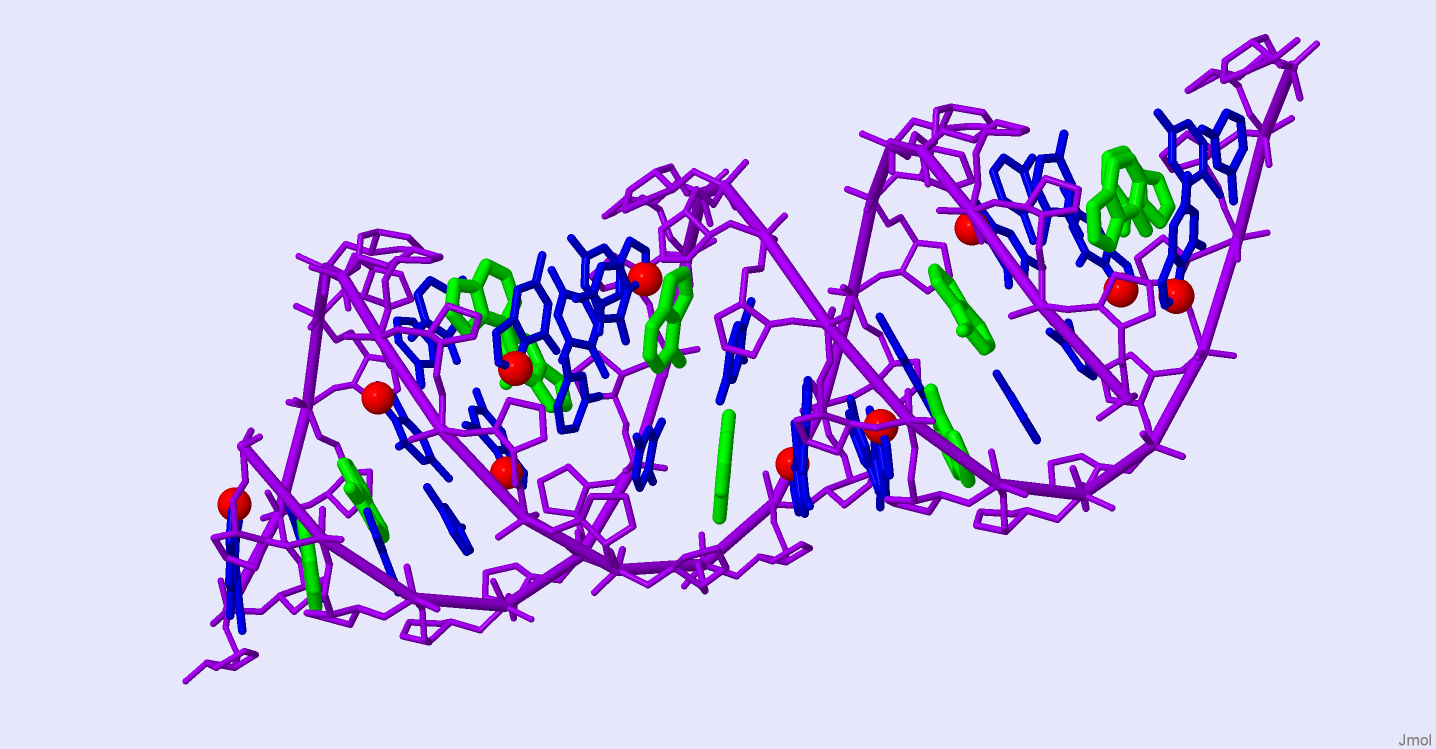
Рис. 2. Структура А-формы дуплекса ДНК. Зелёным выделены остатки аденина. Красным цветом выделены атомы N7 во всех гуанинах. Изображение получено с помощью программы Jmol.
Далее из базы данных PDB были скачаны pdb-файлы структур 1GTR и 1RH6. Содержащиеся в них цепи нуклеиновых кислот были проверены на визуальное наличие разрывов с помощью программы Jmol. В цепи тРНК из структуры 1GTR разрывов найдено не было. Двойная цепь ДНК из структуры 1RH6 также не содержит разрывов. Изображения, иллюстрирующие данные выводы, приведены на рис. 3.
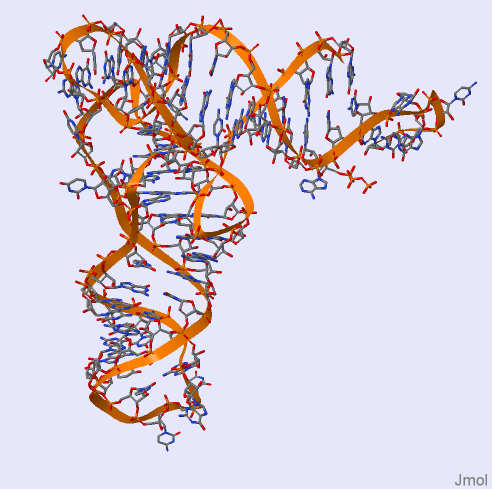
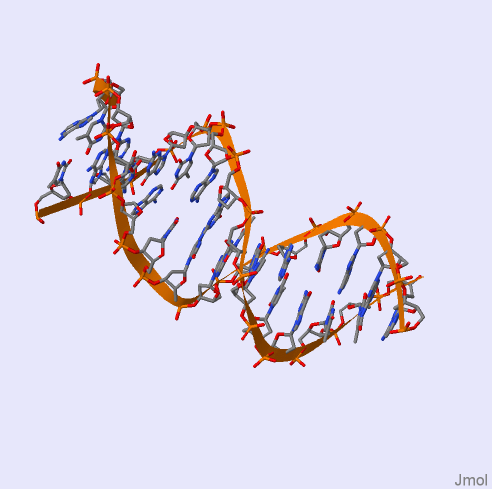
Рис. 3. Графические изображения структур нуклеиновых кислот в проволочной и ленточной модели. Слева - тРНК из pdb-файла 1GTR. Справа - ДНК из pdb-файла 1RH6. Окраска атомов - в соответствии с их химической природой (cpk). Изображения получены с помощью программы Jmol.
Сравнение структур 3-х форм ДНК с помощью средств JMol.
В структуре дуплексов ДНК имеются большие и малые бороздки. Их можно определить, посмотрев на скруктуру дуплекса. Изображение В-формы дуплекса ДНК с отмеченными бороздками представлено на рис. 4.
Далее был рассмотрен тимин в структуре дуплекса - определено направление его атомов (т. е. в сторону какой именно бороздки они направлены). Атомы N1, O2, С2 направлены в сторону малой бороздки и на рис. 5 обозначены синим цветом. Атомы N4, С4, С5, С6, С7 обращены в сторону большой бороздки и обозначены на рис. 5 красным цветом. Оставшийся атом - N3 не имеет строгого направления.
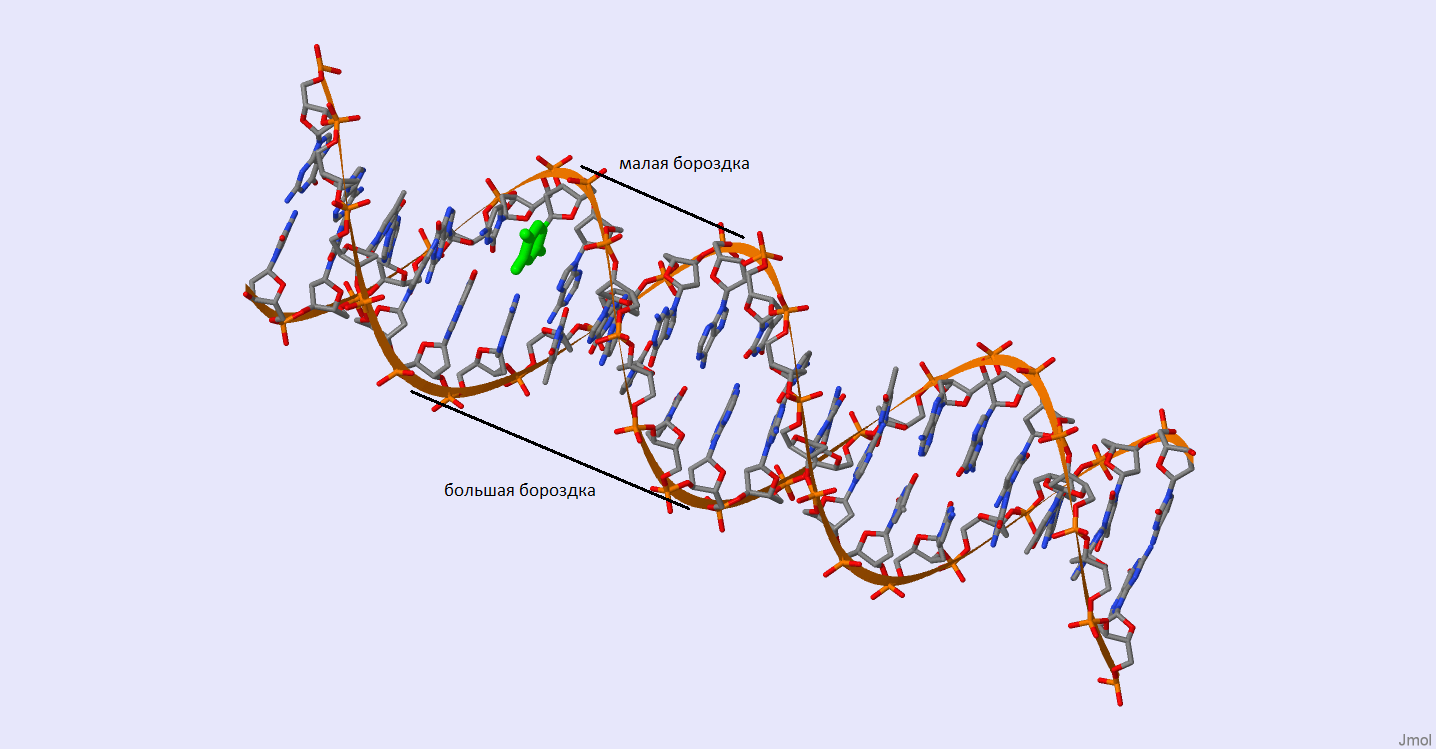
Рис. 4. Структура B-формы дуплекса ДНК в проволочной и ленточной модели. Подписаны большая и малая бороздки. Зелёным выделен рассматриваемый остаток тимина (35-ый тимин). Изображение получено с помощью программы Jmol.
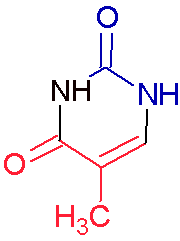
Рис. 5. Графическое представление структуры тимина. Красным выделены атомы, направленные в сторону большой бороздки В-формы дуплекса ДНК. Синим - в сторону малой. Изображение получено с помощью программы ChemSketch.
Для А-формы дуплекса ДНК направление атомов тимина такое же, как и в В-форме. В Z-форме тимина нет.
Затем нужно было сравнить основные параметры спиралей разных форм ДНК. Результаты сравнения показаны в таблице 1.
Таблица 1. Параметры структур А, В и Z-форм ДНК.
| А-форма | В-форма | Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (A) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (A) | 18,5 (13-32) | 20,58 (3-33) | 23,5 (10-26) |
| Ширина малой бороздки (A) | 9,63 (3-31) | 13,2 (13-33) | 8,7 (15-31) |
Далее были измерены торсионные углы выбранного нуклеотида (35-го тимина). Для этого была использована команда Measurements -> Click for torsion (dihedral) measurement (для выбора команды нужно сначала кликнуть правой кнопкой мыши по Jmol). Результаты измерений для двух форм ДНК и значения торсионных углов, приведённые в презентации, представлены в таблице 2.
Таблица 2. Торсионные углы тимина 35 А и В-форм ДНК.
| α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ζ (O3' - P) |
χ (C1' - N) |
|
| А-форма (из презентации) | 62 |
173 | 52 | 88 или 3 |
178 | -50 |
-160 |
| А-форма (gatc-a.pdb) | 64,1 |
174,8 |
41,7 |
79,1 |
-147,8 |
-75,1 |
-157,2 |
| В-форма (из презентации) | 63 |
171 | 54 | 123 или 131 | 155 | -90 | -117 |
| В-форма (gatc-b.pdb) | 85, 9 |
136,4 |
31,1 |
143,4 |
-140,8 |
-160,5 |
-98 |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Чтобы выполнить определить параметры структур нуклеиновых кислот, скаченные pdb-файлы были переведены в старый формат PDB (т. к. пакет 3DNA пока не может работать с новым форматом). Для этого были использованы комманды:
remediator --old ''1GTR.pdb'' > ''1GTR_old.pdb
remediator --old ''1RH6.pdb'' > ''1RH6_old.pdb
find_pair -t 1GTR.pdb stdout | analyze
find_pair -t 1RH6.pdb stdout | analyze
В результате было получено несколько файлов, в том числе наиболее нужные нам файлы 1GTR_old.out и 1RH6_old.out
Файлы 1GTR_old.out и 1RH6_old.out, полученные ранее, использовались для получения информации о торсионных углах и водородных связях.Средние значения торсионных углов были вычислены с помощью программы Excel. Крайние нуклеотиды не учитывались. Значения торсионных углов тРНК приведены в таблице 3. Самый "деформированный" нуклеотид - 7-ой гуанин 2-ой цепи, суммарное отклонение значений его торсионных углов от среднего оказалось наибольшим.
Таблица 3. Торсионные углы тРНК из структуры 1GTR.
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| 1 | G | --- | 150,2 | 59,6 | 82,1 | -144,4 | -76,7 | -168,3 |
| 2 | G | -69,4 | 167,1 | 60,8 | 79,6 | -161,1 | -73,6 | -166,1 |
| 3 | G | -62,7 | 163,7 | 61,5 | 80,8 | -150,7 | -73,8 | -164,7 |
| 4 | G | 152,7 | -165 | 177,6 | 80,7 | -146,7 | -69,8 | -170,8 |
| 5 | U | -68,7 | 171 | 59,6 | 79,5 | -168,3 | -78,1 | -162,8 |
| 6 | A | 169,6 | -176,7 | -179,4 | 159,1 | --- | --- | -143,3 |
| 7 | C | --- | 159,7 | 47 | 82,1 | -141,9 | -81,4 | -168,5 |
| 8 | G | -70,1 | 179,1 | 58 | 81,1 | -161,3 | -78,9 | -161,6 |
| 9 | A | -65,2 | 163,4 | 55,5 | 81,4 | -145,1 | -116 | -158,8 |
| 10 | G | 157 | -124,7 | 154 | 78,8 | -150,2 | -74,9 | -177,9 |
| 11 | G | -54,5 | 110,2 | 82,1 | 74,5 | -122,6 | -90,1 | 173,6 |
| 12 | U | -68,7 | 174,7 | 55,9 | 80,8 | -129,8 | -77,7 | -161 |
| 13 | U | -68,5 | 168,8 | 58 | 77,3 | --- | --- | -160 |
| 14 | A | --- | -178,7 | 61 | 80,9 | -141,1 | -64,2 | -173,4 |
| 15 | U | -64,7 | 160,9 | 62,8 | 82,6 | -139,2 | -62,3 | -168,3 |
| 16 | U | -69,1 | 170,3 | 55,2 | 78,7 | -148,6 | -75 | -161,8 |
| 17 | C | -69 | 171,3 | 54,7 | 79,9 | -164,1 | -109,4 | -163,5 |
| 18 | C | -180 | -86 | 97,3 | 71,4 | -133,8 | -83,6 | -165,1 |
| 19 | G | -61,7 | 155,4 | 63,6 | 73,3 | -167,3 | -70,1 | -169,2 |
| 20 | G | -60,3 | 154,3 | 61,5 | 73,5 | -140,3 | -82,6 | -165,5 |
| 21 | C | 56,9 | -85,8 | -137,9 | 98,1 | --- | --- | -167,7 |
| 22 | G | --- | 164,3 | 56,3 | 89,9 | -144,4 | -74,8 | 179,9 |
| 23 | C | -78,4 | -175,1 | 56,5 | 84,2 | -150,3 | -64,4 | -159 |
| 24 | C | -72 | 160 | 64,5 | 74,5 | -167 | -55,9 | -168,1 |
| 25 | A | -64,3 | 164 | 59,4 | 82,9 | -174,3 | -71,2 | -159,7 |
| 26 | A | -66,7 | 149,9 | 70,6 | 77,6 | -130,8 | -74,3 | -167,6 |
| 27 | G | -52,8 | 144,9 | 54,2 | 79 | --- | --- | -145,8 |
| 28 | G | --- | -166,3 | 61,4 | 155,2 | --- | --- | -74,8 |
| 1 | C | -71,3 | -179,4 | 58,2 | 84,8 | --- | --- | -146,2 |
| 2 | C | -75,3 | 177,7 | 58 | 81 | -155,7 | -78,8 | -157,2 |
| 3 | C | -70,7 | 171,3 | 58,2 | 80,9 | -155,3 | -72,2 | -165,6 |
| 4 | C | -68,8 | 159,2 | 63,2 | 78,8 | -154 | -76 | -157,9 |
| 5 | A | -75,3 | 170,7 | 61,5 | 83,5 | -140,6 | -82,8 | -161 |
| 6 | U | -62,4 | 160,7 | 60,1 | 78,8 | -151,9 | -77 | -163 |
| 7 | G | 155,7 | -134,4 | 167,5 | 79,8 | -126,7 | -79,8 | 175,2 |
| 8 | C | -63,4 | 140,6 | 57,2 | 77,2 | -152,1 | -107,9 | -157,3 |
| 9 | U | -121,3 | 99,8 | 146,4 | 93,9 | -93,4 | -95,8 | 176,1 |
| 10 | C | 51 | 83,3 | 44,4 | 74,2 | -138 | -58,3 | -171,2 |
| 11 | C | --- | 175,8 | 50,6 | 78,5 | -171,8 | -132,3 | -169 |
| 12 | A | --- | -124 | 66,3 | 148,6 | --- | --- | -69,3 |
| 13 | G | --- | 159,7 | -63,4 | 150,1 | --- | --- | -96,7 |
| 14 | U | -60,4 | 161,2 | 56,8 | 79,6 | --- | --- | -152,7 |
| 15 | U | 175,3 | -82,5 | 100,7 | 67,9 | -164,6 | -68,6 | -161 |
| 16 | A | -72,3 | 175 | 56,4 | 78,2 | -152,4 | -115,1 | -168,2 |
| 17 | G | 51,2 | 67,4 | 46,6 | 69,5 | -167,2 | -82,3 | -172 |
| 18 | G | -105,7 | 75,4 | 149,6 | 91,1 | -123 | -144,3 | 170,4 |
| 19 | C | -60,2 | 164,5 | 57,4 | 86,6 | -132,4 | -48,2 | -161,1 |
| 20 | C | -65,6 | 167,6 | 55,4 | 80,8 | -150,8 | -87 | -157,2 |
| 21 | A | -67,8 | 162,3 | 56,6 | 76,6 | -157,2 | -67,6 | -165 |
| 22 | C | -60,2 | 159,4 | 57,5 | 79,7 | -151,1 | -60,5 | -160,4 |
| 23 | G | -61,4 | 168,2 | 60,6 | 79,1 | -153 | -70,7 | -165,9 |
| 24 | G | --- | 165,1 | 58,6 | 78,4 | -155,7 | -78,1 | -170,2 |
| 25 | A | --- | -138,1 | 58,9 | 78,3 | --- | --- | -92,8 |
| 26 | A | --- | -118,4 | -175,9 | 81,2 | --- | --- | -152,7 |
| 27 | C | --- | 169,8 | 58,2 | 151,7 | --- | --- | -129,6 |
| 28 | C | --- | 169,7 | 56,1 | 84,9 | --- | --- | -172,5 |
| среднее | -28,9 | 78,8 | 54,8 | 86,8 | -115,3 | -62,7 | -125,1 |
Значения торсионных углов ДНК приведены в таблице 4. Самый "деформированный" нуклеотид - 12-ый тимин первой цепи.
Таблица 4. Торсионные углы ДНК из структуры 1RH6.
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| 1 | T | --- | -98,5 | 78,5 | 95,9 | -174,2 | -86,8 | -124,7 |
| 2 | A | -65,7 | 176,3 | 46,4 | 108,3 | 179 | -88,7 | -118,6 |
| 3 | T | -62,9 | 173,3 | 55,3 | 106,2 | 174,8 | -85,2 | -127,2 |
| 4 | G | -60,1 | -174,3 | 43,7 | 137,9 | -179,5 | -105,6 | -113,7 |
| 5 | T | -52,4 | -174,6 | 40,7 | 134 | -176,8 | -97,9 | -102,2 |
| 6 | A | -57,6 | 173,3 | 35,3 | 156,8 | -119,7 | -155,2 | -88,1 |
| 7 | G | -79,4 | 128,9 | 51,9 | 69,1 | 167,5 | -65,5 | -155,5 |
| 8 | T | -67,3 | -169,2 | 62,4 | 142,1 | -170,4 | -97,5 | -117,5 |
| 9 | C | -69,1 | 168,4 | 56,2 | 118,1 | -173,6 | -103,7 | -130,8 |
| 10 | T | -69,8 | 179,9 | 61,1 | 124,5 | -171,5 | -101,6 | -115 |
| 11 | G | -71,5 | 175,9 | 60,4 | 140,5 | 175,6 | -106,2 | -106 |
| 12 | T | -64,2 | -178,3 | 58,5 | 128,1 | 172,8 | -99,1 | -112,3 |
| 13 | T | -62,5 | 172,2 | 60,5 | 105 | -164,3 | -84,3 | -134,2 |
| 14 | G | -67,2 | -178,8 | 52,3 | 144,3 | --- | --- | -88 |
| 1 | A | -41,8 | 127 | 85,9 | 130,3 | --- | --- | 163,3 |
| 2 | T | 15,3 | 124,6 | 13,2 | 144,1 | -169,6 | -164,6 | -83,4 |
| 3 | A | -45,9 | 177 | 33,3 | 125,1 | 179,9 | -124,7 | -111,5 |
| 4 | C | -51,2 | 164,7 | 39,8 | 120,5 | 178,6 | -117,4 | -106,2 |
| 5 | A | -46,4 | 177,1 | 53,7 | 149,7 | -174,4 | -102,4 | -95,7 |
| 6 | T | -62,8 | 177,1 | 51,1 | 93,8 | -164,4 | -95,8 | -140,6 |
| 7 | C | -77 | 154,6 | 63,7 | 66,6 | -176,2 | -74,3 | -152,3 |
| 8 | A | -63,9 | -158,2 | 37 | 150,5 | -169,2 | -83,1 | -106,8 |
| 9 | G | -55,7 | 157,9 | 65,4 | 111,3 | -176,4 | -104,7 | -136 |
| 10 | A | -70,1 | 176,3 | 52,8 | 130,8 | 179,8 | -92,4 | -101,4 |
| 11 | C | -43,7 | 169,8 | 43,1 | 125,8 | -170,6 | -104,2 | -116,1 |
| 12 | A | -44,9 | 173,2 | 43,3 | 144,9 | -175,1 | -112,6 | -96,1 |
| 13 | A | -51,3 | 144,9 | 50,3 | 137 | 178,7 | -119,4 | -96,1 |
| 14 | C | --- | -98,6 | 58,4 | 141,5 | -139,6 | -160,9 | -94,9 |
| среднее | -57,5 | 95,5 | 49,1 | 123,8 | -39,4 | -103,6 | -115,1 |
Определение структуры водородных связей.
Чтобы понять, как определить номера нуклеотидов, образующих стебли во вторичной структуре заданной тРНК, попробуем разобраться в принципах строения тРНК. Наглядная схема вторичной структуры тРНК приведена на рис. 6.
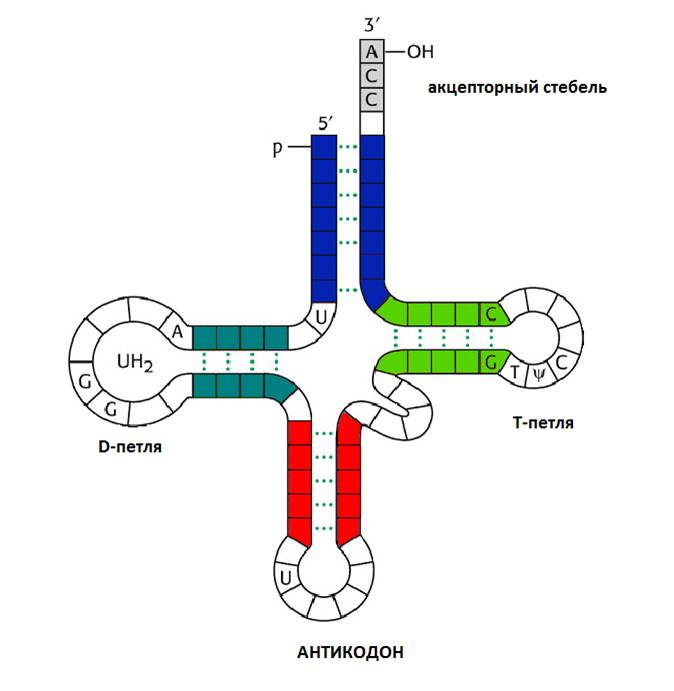
Рис. 6. Вторичная структура тРНК.
Акцепторный стебель (7 пар нуклеотидов) формируется за счет спаривания 5'- и 3'-концевых нуклеотидов (на 3'-конце неспаренные CCA для прикрепления аминокислоты).D-петля стабилизирована 4 парами нуклеотидов и содержит дигидроуридин.
Антикодоновая петля стабилизирована 5 парами нуклеотидов и содержит антикодон.
T-петля стабилизирована 5 парами нуклеотидов и содержит ТΨС последовательность, где Ψ - псевдоуридин.
Номера нуклеотидов, образующих водородные связи, приведены ниже, они также, как и значения торсионных углов, получены из файла 1GTR_old.out. Красным цветом выделен акцепторный стебель, синим Т-стебель, фиолетовым D-стебель и зелёным антикодоновый стебель.
Strand I Strand II Helix
1 (0.012) B:...2_:[..G]G-----C[..C]:..71_:B (0.011) |
2 (0.010) B:...3_:[..G]G-----C[..C]:..70_:B (0.009) |
3 (0.011) B:...4_:[..G]G-----C[..C]:..69_:B (0.011) |
4 (0.011) B:...5_:[..G]G-----C[..C]:..68_:B (0.012) |
5 (0.013) B:...6_:[..U]U-----A[..A]:..67_:B (0.011) |
6 (0.014) B:...7_:[..A]Ax----U[..U]:..66_:B (0.013) |
7 (0.013) B:..49_:[..C]C-----G[..G]:..65_:B (0.010) |
8 (0.012) B:..50_:[..G]G-----C[..C]:..64_:B (0.011) |
9 (0.015) B:..51_:[..A]A-----U[..U]:..63_:B (0.016) |
10 (0.008) B:..52_:[..G]G-----C[..C]:..62_:B (0.010) |
11 (0.010) B:..53_:[..G]G----xC[..C]:..61_:B (0.009) |
12 (0.011) B:..54_:[..U]U-**-xA[..A]:..58_:B (0.010) |
13 (0.017) B:..55_:[..U]Ux**+xG[..G]:..18_:B (0.009) x
14 (0.010) B:..37_:[..A]A-*---U[..U]:..33_:B (0.014) |
15 (0.016) B:..38_:[..U]U-*---U[..U]:..32_:B (0.016) |
16 (0.013) B:..39_:[..U]U-----A[..A]:..31_:B (0.012) |
17 (0.010) B:..40_:[..C]C-----G[..G]:..30_:B (0.010) |
18 (0.011) B:..41_:[..C]C-----G[..G]:..29_:B (0.013) |
19 (0.008) B:..42_:[..G]G-----C[..C]:..28_:B (0.011) |
20 (0.009) B:..43_:[..G]G-----C[..C]:..27_:B (0.011) |
21 (0.015) B:..44_:[..C]Cx*---A[..A]:..26_:B (0.012) |
22 (0.011) B:..10_:[..G]G-----C[..C]:..25_:B (0.010) |
23 (0.012) B:..11_:[..C]C-----G[..G]:..24_:B (0.012) |
24 (0.011) B:..12_:[..C]C----xG[..G]:..23_:B (0.011) |
25 (0.012) B:..13_:[..A]A-**+xA[..A]:..45_:B (0.012) |
26 (0.011) B:..14_:[..A]A-*--xA[..A]:..21_:B (0.014) |
27 (0.008) B:..15_:[..G]Gx**+xC[..C]:..48_:B (0.009) x
28 (0.009) B:..19_:[..G]G-----C[..C]:..56_:B (0.011) +
- В соответствии с полученными данными:
- акцепторный стебель состоит из участка 3 - 7 и комплементарного ему участка 66 - 70
- Т-стебель состоит из участка 50 - 53 и комплементарного ему участка 61 - 64
- антикодоновый стебель состоит из участка 38 - 44 и комплементарного ему участка 26 - 32
- D-стебель состоит из участка 11 - 12 и комплементарного ему участка 23 - 24
Неканонические пары составляют (отмечены в файле звёздочками) 12 U-A, 13 U-G, 15 U-U, 21 C-A, 25 A-A, 26 A-A, 27 G-C. Среди них есть как и некомплементарные пары, так и комплементарные, но с неканоническими водородными связями.
Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру составляют 12 U-A, 13 U-G, 25 A-A, 26 A-A, 27 G-C, 28 G-C (это комплементарные пары, не имеющие отношения к стеблям).
Изображение заданной структуры тРНК с окрашенными стеблями приведено на рис. 7.

Рис. 7. Структура тРНК из файла 1GTR.pdb. Красным цветом выделен акцепторный стебель, синим Т-стебель, фиолетовым D-стебель и зелёным антикодоновый стебель.
Возможные стекинг-взаимодействия.
Чтобы определить пары нуклеотидов, для которых в наибольшей степени возможно стекинг-взаимодействие, была проанализирована информация о перекрывании нуклеотидов, полученная из файла 1GTR_old.out:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 4.05( 2.85) 0.00( 0.00) 0.09( 0.00) 0.63( 0.00) 4.76( 2.85)
2 GG/CC 3.65( 2.31) 0.00( 0.00) 0.35( 0.00) 0.06( 0.00) 4.06( 2.31)
3 GG/CC 3.96( 2.48) 0.00( 0.00) 0.52( 0.00) 0.00( 0.00) 4.49( 2.48)
4 GU/AC 6.66( 3.82) 0.00( 0.00) 0.00( 0.00) 4.45( 2.93) 11.11( 6.75)
5 UA/UA 0.62( 0.00) 0.00( 0.00) 1.16( 0.94) 0.13( 0.00) 1.91( 0.94)
6 AC/GU 2.76( 1.60) 0.00( 0.00) 0.00( 0.00) 6.27( 3.25) 9.03( 4.85)
7 CG/CG 0.00( 0.00) 0.00( 0.00) 5.60( 2.70) 0.00( 0.00) 5.60( 2.70)
8 GA/UC 4.18( 2.38) 0.00( 0.00) 0.00( 0.00) 2.65( 0.35) 6.82( 2.73)
9 AG/CU 3.53( 3.27) 0.00( 0.00) 0.23( 0.00) 0.33( 0.00) 4.09( 3.27)
10 GG/CC 3.60( 2.09) 0.00( 0.00) 0.92( 0.00) 0.00( 0.00) 4.52( 2.09)
11 GU/AC 6.60( 3.77) 0.00( 0.00) 0.00( 0.00) 4.43( 1.73) 11.04( 5.50)
12 UU/GA 4.81( 2.33) 0.00( 0.00) 0.00( 0.00) 6.06( 3.01) 10.87( 5.35)
13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
14 AU/UU 4.84( 1.19) 0.00( 0.00) 0.00( 0.00) 3.97( 1.95) 8.80( 3.14)
15 UU/AU 2.33( 0.94) 0.00( 0.00) 0.00( 0.00) 4.93( 3.75) 7.25( 4.68)
16 UC/GA 0.11( 0.01) 0.00( 0.00) 0.00( 0.00) 2.57( 1.07) 2.68( 1.07)
17 CC/GG 0.65( 0.02) 0.00( 0.00) 0.30( 0.00) 3.18( 1.73) 4.12( 1.75)
18 CG/CG 0.00( 0.00) 0.00( 0.00) 5.88( 2.66) 0.00( 0.00) 5.88( 2.66)
19 GG/CC 3.83( 2.44) 0.00( 0.00) 0.57( 0.00) 0.07( 0.00) 4.47( 2.44)
20 GC/AC 6.92( 3.76) 0.00( 0.00) 0.00( 0.00) 5.28( 3.35) 12.21( 7.11)
21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.52( 0.00) 0.69( 0.21) 1.21( 0.21)
22 GC/GC 4.19( 1.51) 0.00( 0.00) 0.00( 0.00) 6.41( 3.26) 10.61( 4.77)
23 CC/GG 0.05( 0.00) 0.00( 0.00) 0.52( 0.00) 3.10( 1.58) 3.66( 1.58)
24 CA/AG 1.89( 0.97) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 1.89( 0.97)
25 AA/AA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 5.05( 4.27) 5.05( 4.27)
26 AG/CA 4.22( 1.87) 0.00( 0.00) 0.00( 0.00) 5.49( 2.42) 9.71( 4.29)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Очевидно, что наибольшее перекрывание (суммарно 6.75) наблюдается для пар 20 GC/AC. Из файла stacking.pdb с помощью комманды "ex_str -20 stacking.pdb st20.pdb" был вырезан файл со структурой нужных нам пар. Затем с помощью команды "stack2img -cdolt st20.pdb st20.ps" было получено изображение стэкинг-взаимодействия пар 20 GC/AC (приведено на рис. 8). Далее, чтобы проверить взаимное расположение пар нуклеотидов, было получено изображение этих пар с помощью программы Jmol (оно приведено на рис. 8).
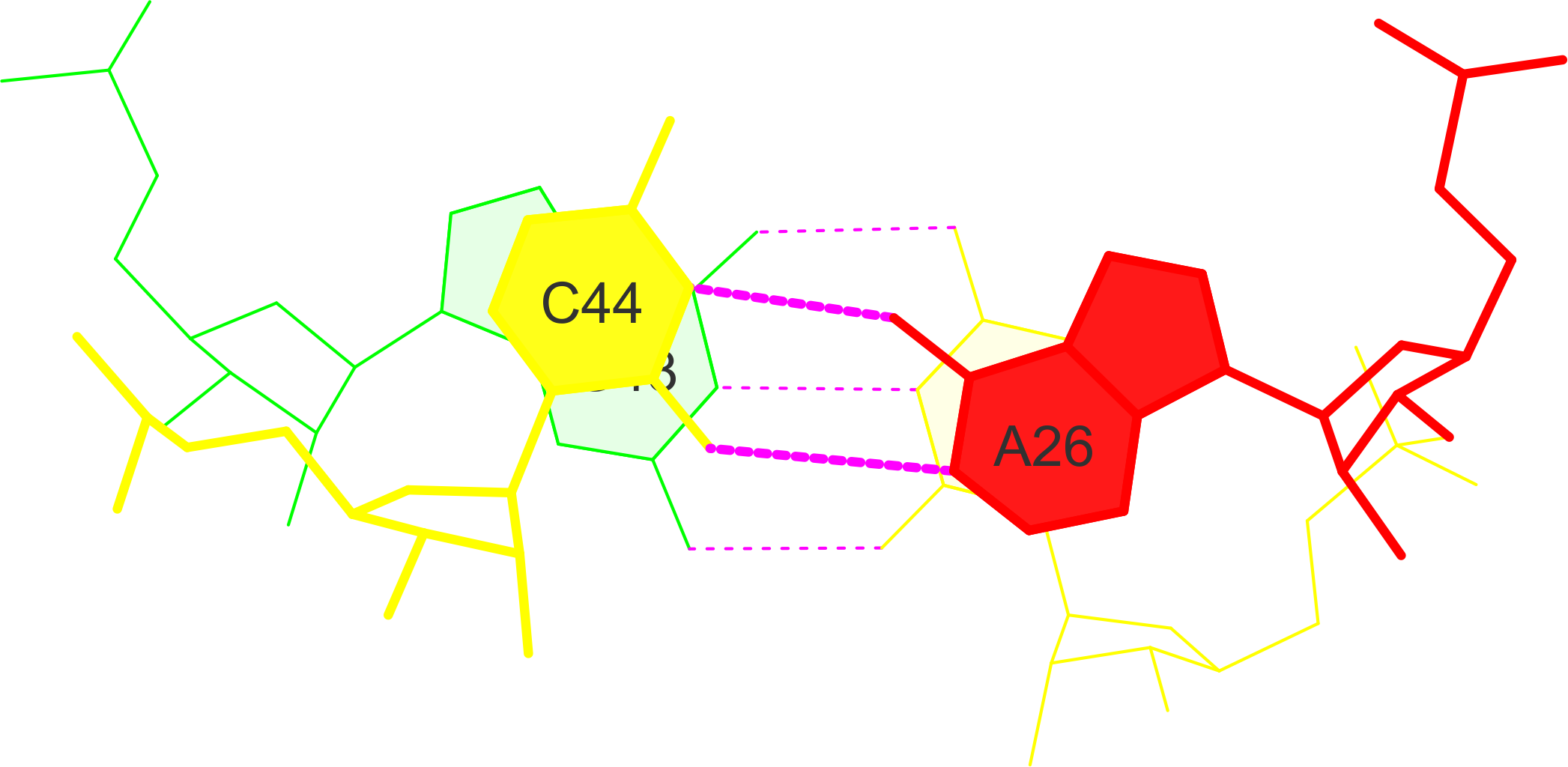
Рис. 8. Стэкинг-взаимодействие пар 20 GC/AC.
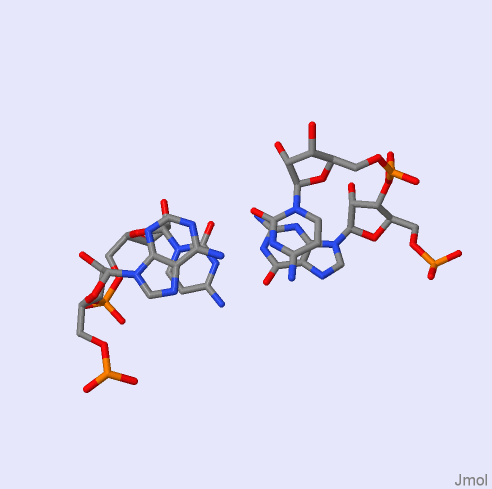
Рис. 9. Взаимное расположение пар 20 GC/AC. Изображение получено с помощью программы Jmol.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016