| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
EMBOSS
Программа getorf пакета EMBOSS
С помощью команды entret пакета EMBOSS получен файл d89965.entret с записью D89965 банка EMBL.В данной записи содержится последовательность мРНК из 448-ми нуклеотидов, содержащюю участок с геном (Rat Stomach Serotonin receptor-related gene), кодирующим белок RSS серой крысы (Rattus norvegicus). Кодирующий участок последовательности (CDS) имеет координаты 163..435.
С помощью программы getorf получен набор трансляций всех открытых рамок считывания, содержащихся в данной последовательности. Нужные нам рамки считывания одновременно: определены при использовании стандартного генетического кода, имеют длину не менее 30 аминокислотных остатков, начинаются со старт-кодона (то есть с начала последовательности) и заканчиваются стоп-кодоном (до конца последовательности). Для их получения использовалась следующая команда:"getorf -minsize 90 -table 0 -find 1 embl:d89965". Программа getorf создала файл d89965.orf, содержащий набор трансляций открытых рамок считывания, отвечающих вышеприведённым условиям.
Приведённой в поле FT кодирующей последовательности (CDS) частично соответствует третья открытая рамка считывания (D89965_3):
D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Координаты начала этих последовательностей совпадают (163). Координаты конца отличаются на 3 нуклеотида, возможно, потому что CDS включает в себя стоп-кодон, а найденная с помощью getorf рамка считывания - нет.
Запись EMBL ссылается на запись Swiss-Prot с идентификатором P0A7B8. Файл hslv_ecoli.entret с соответствующей записью Swiss-Prot был получен с помощью команды: "entret sw:P0A7B8". Чтобы найти рамку считывания, соответствующую последовательности, содержащейся в Swiss-Prot, запущена программа needle: "needle sw:P0A7B8 d89965.orf" (в качестве первого входа указан идентификатор соответствующей последовательности в Swiss-Prot, а в качестве второго – файл с транслированными последовательностями открытых рамок считывания (выдача getorf)). Когда программа needle получает в качестве одного из входов не одну, а несколько последовательностей, она выполняет все возможные парные выравнивания.
После анализа полученного файла hslv_ecoli.needle, содержащего 5 выравниваний (для каждой транстированной рамки - выравнивание с последовательностью белка из Swiss-Prot), приходим к выводу, что последовательности Swiss-Prot соответствует пятая рамка D89965_5 (выравнивания с остальными транслированными рамками считывания содержат гэпы).
Запись P0A7B8 банка Swiss-Prot содержит последовательность белка бактерии Escherichia coli, тогда как последовательность соответствующей записи EMBL является последовательностью мРНК серой крысы. По всей видимости, эксперимент по секвенированию мРНК крысы был проведён с ошибкой. Образец мРНК крысы был загрязнён посторонней мРНК кишечной палочки (что вполне вероятно, т. к. анализировали мРНК из желудка крысы). Расшифровав ген кишечной палочки (думая, что он тоже принадлежит серой крысе), экспериментаторы по ошибке включили его рамку считывания в последовательность мРНК крысы.
Файлы-списки
С помощью команды "seqret sw:adh*_* adh.fasta" скачаны все доступные в Swiss-Prot последовательности алкогольдегидрогеназ. Полученный файл - adh.fasta.
С помощью программы infoseq, запущенной с параметрами -only и -usa, получен файл-список usa.txt, содержащий универсальные адреса (USA) этих последовательностей. Для этого использовалась следующая команда:"infoseq adh.fasta -only -usa -out usa.txt".
Из списка USA последовательностей алкогольдегидрогеназ сформирован меньший список, содержащий адреса только тех последовательностей, которые взяты из организмов: DROTE, BOVIN, DROMU, METM6, DROMN, OCTVU, DROWI. Для этого сначала был создан файл-список organisms.txt, содержащий список идентификаторов этих организмов. Затем с помощью команды "grep -f organisms.txt usa.txt > org_usa.txt" был сформирован файл-список адресов (USA) последовательностей из нужных нам организмов - org_usa.txt.
С помощью программы seqret получен fasta-файл с последовательностями алкогольдегидрогеназ организмов: DROTE, BOVIN, DROMU, METM6, DROMN, OCTVU, DROWI. Использовалась команда:"seqret @org_usa.txt seq.fasta". Полученный файл - seq.fasta.
Случайная модель для оценки достоверности выравнивания
Проведена оценка достоверности вывода о гомологии последовательностей алкогольдегидрогеназ Drosophila melanogaster (последовательность ADH1_DROMU.fasta) и Bos taurus (последовательность ADHX_BOVIN.fasta). Для этого с помощью программы shuffleseq (команда "shuffleseq ADH1_DROMU.fasta") сгенерированы 100 случайных перемешиваний последовательности алкогольдегидрогеназы Drosophila melanogaster. Файл, содержащий "перемешанные" последовательности - shuffled_dromu.fasta.
С помощью программы water были построены выравнивания последовательности ADHX_BOVIN с последовательностью ADH1_DROMU и с каждой "перемешанной" последовательностью. Файл-выравнивание последовательности ADHX_BOVIN с исходной последовательностью ADH1_DROMU - adhx_bovin.water. Выравнивание "реальных" последовательностей имеет вес 49,5. Файл, содержащий выравнивания последовательности ADHX_BOVIN с "перемешанными" последовательностями - adhx_bovin_shuffled.water. Файл scores.txt, содержащий список весов этих выравниваний, получен с помощью команды:"grep '# Score:' adhx_bovin_shuffled.water | sed 's/# Score: //g' > scores.txt"
C помощью MS Excel построена гистограмма распределения весов 101-го парного выравнивания (приведена на рис. 1).
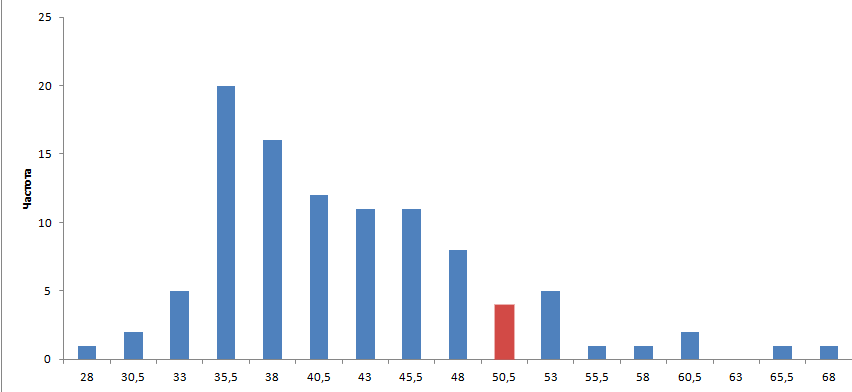
Рис. 1. Гистограмма распределения весов выравниваний аминокислотной последовательности алкогольдегидрогеназы Bos taurus с последовательностью алкогольдегидрогеназы Drosophila melanogaster и с сотней "перемешанных" последовательностей (полученных случайным перемешиванием последовательности алкогольдегидрогеназы Drosophila melanogaster). Красным отмечен столбик, соответствующий диапазону, в который входит выравнивание "реальных" последовательностей. Гистограмма получена с помощью программы MS Excel.
Вес выравнивания исходных последовательностей попадает в диапазон 48-50,5. Он достаточно отличается от большинства весов выравниваний с "перемешанными" последовательностями, поэтому гомологию соответствующих белков можно считать возможной.
Аналогичные действия были проведены для нуклеотидных последовательностей, соответствующих генам белков ADHX_BOVIN и ADH1_DROMU (соответствующие нуклеотидные последовательности были взяты из банка EMBL). Случайные последовательности генерировались для гена алкогольдегидрогеназы Drosophila melanogaster. Полученная гистограмма распределения весов 101 выравнивания нуклеотидных последовательностей представлена на рис. 2.
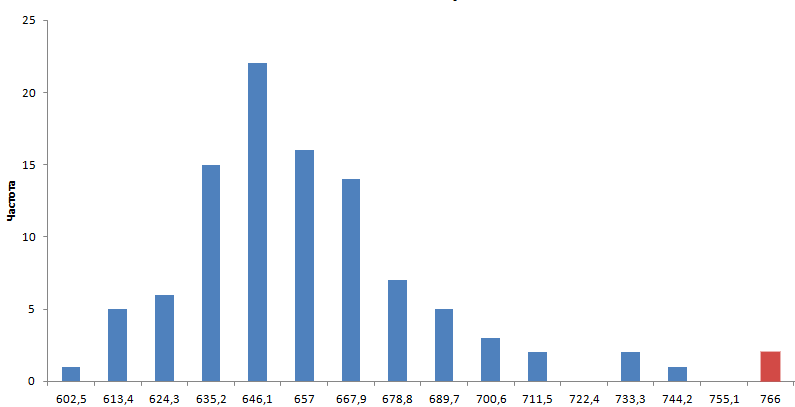
Рис. 2. Гистограмма распределения весов выравниваний нуклеотидной последовательности гена алкогольдегидрогеназы Bos taurus с последовательностью гена алкогольдегидрогеназы Drosophila melanogaster и с сотней "перемешанных" последовательностей (полученных случайным перемешиванием последовательности гена алкогольдегидрогеназы Drosophila melanogaster). Красным отмечен столбик, соответствующий диапазону, в который входит выравнивание "реальных" последовательностей. Гистограмма получена с помощью программы MS Excel.
Вес выравнивания исходных нуклеотидных последовательностей (761) попадает в последний диапазон 755,1-766. Т. е. он достаточно сильно отличается от весов выравниваний с "перемешанными" последовательностями.
Для выбранных алкогольдегидрогеназ вес выравнивания реальных (не "перемешанных") нуклеотидных последовательностей сильнее отличается от весов выравниваний последовательности алкогольдегидрогеназы Bos taurus с "перемешанными", чем вес выравнивания соответствующих аминокислотных последовательностей. Это происходит потому, что вариантов нуклеотидных последовательностей, получающихся при перемешивании, больше, чем соответствующих аминокислотных, следовательно всего при перемешивании аминокислотной последовательности меньше шансов получить "хорошую", гомологичную последовательность с соотвествующим весом выравнивания, близким к весу выравнивания "реальных" последовательностей, чем при перемешивании нуклеотидной последовательности.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016