| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Предсказание генов у эукариот
Для фрагмента ДНК человека с помощью программ GENSCAN и UCSC Genome Browser определена экзон-интронная структура гена и описан его альтернативный сплайсинг.
GENSCAN
Исследуемая последовательность была подана на вход программе GENSCAN. Выбраны настройки для позвоночных (Vertebrate). В результате был предсказан один ген. Информация о его экзонах получена в виде таблицы. Часть этой информации можно увидеть в таблице 1. Были исключены записи, не соответствующие экзонам: PlyA (сайт полиаденилирования) и Prom (промотор).
Таблица 1. Информация об экзонах гена человека, предсказанного с помощью GENSCAN
| Начало | Конец | Цепь | Тип |
| 4590 | 4454 | - | конечный |
| 6474 | 6315 | - | внутренний |
| 7262 | 7084 | - | внутренний |
| 10158 | 9959 | - | внутренний |
| 14362 | 14251 | - | внутренний |
| 15630 | 15531 | - | начальный |
Genome Browser
Затем предсказание экзон-интронной структуры того же участка проводилось с помощью программы BLAT, которая доступна с сайта UCSC Genome Browser. Данная программа позволяет искать последовательности в геноме с учетом его возможной фрагментированности. Последовательность ДНК была помещена в текстовое поле формы, выбран поиск в геноме человека, сборка hg38 (Dec. 2013). Получен список найденных фрагментов генома, из них выбран фрагмент, имеющий 100% идентичность с исследуемой последовательностью и наибольший вес (18958).
Перейти к просмотру выбранного найденного фрагмента генома человека можно, нажав гиперссылку browser. Рассматривались участки гена, предсказанные на основе мРНК и EST (короткие фрагменты (клонированной) мРНК). Для отображения найденной последовательности и этих участков нужно поставить на "pack" переключатель Blat Sequence в группе Mapping and Sequencing Tracks, а также переключатели Human mRNAs и Spliced ESTs в группе mRNA and EST Tracks, остальные переключатели нужно поставить на "hide". Затем нужно нажать кнопку "refresh" (она находится внизу страницы).
На полученном отображении последовательностей мРНК и EST, соответствующих нашему фрагменту ДНК, найдено несколько примеров альтернативного сплайсинга. Кассетные экзоны найдены как среди мРНК (например, между последовательностями AY940729 и J02931), так и среди EST (например, между последовательностями DA029400 и BP220588). Другим примером может послужить альтернативный донорный сайт, образованный последовательностями DA029400 и BI668899. На рис. 1 можно увидеть эти и другие примеры альтернативного сплайсинга данной последовательности.
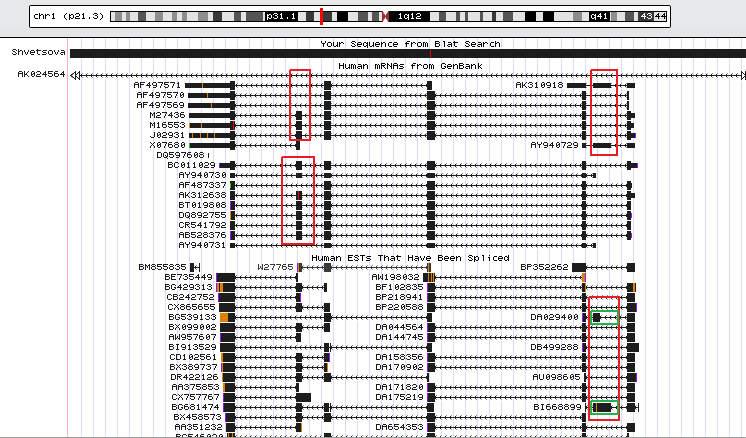
Рис. 1. Фрагмент отображения последовательностей мРНК и EST, соответствующих исследуемуму фрагменту ДНК человека. Получено с помощью Genome Browser и программы BLAT. В красные рамки обведены кассетные экзоны. В зеленые рамки обведены участки последовательностей, составляющие альтернативный донорный сайт.
BLASTX
Предсказание генов и экзон-интронной структуры проводилось для фрагмента ДНК киви (Actinidia chinensis). Геном киви не является хорошо аннотированным (в отличие от генома человека), поэтому для предсказания генов фрагмента генома этого растения невозможно использовать GENSCAN или Genome Browser. Поэтому разметка экзон-интронной структуры генов этого фрагмента производилась с помощью BLASTX (поиск гомологичных последовательностей белков, соответствующих участкам исследуемого фрагмента ДНК).
При поиске был исключён (Exclude) поиск по моделям и пробам среды (галочки возле Models (XM/XP) и Uncultured/environmental sample sequences). Поиск ограничен белками растений (Viridiplantae), исключён геном винограда Vitis vinifera. Поиск проводился по банку SwissProt, использовался стандартный генетический код.
На основе найденных гомологичных последовательностей предсказано 5 генов данного фрагмента. Использовались гомологи с E-value меньше 0.001, предсказания каждого гена составлялись на основе выравнивания с наилучшим гомологом в группе, соответствующей этому гену.
Предсказание предполагаемого гена галактуроносилтрансферазы (Probable galacturonosyltransferase) было составлено на основе лучшего из двух найденных гомологов, гена белка-шаперона - из 14-ти, белка EID1-like F-box - из 2-ух, предполагаемого гена сахаропин дегидрогеназоподобной оксидоредуктазы - на основе одного гомолога и, наконец предсказание гена белка, содержащего пентатрисопептидные повторы - на основе лучшего из более 50-ти найденных гомологов.
Участки перекрытия приписывались экзонам, в которых эти участки составляли наилучшее выравнивание с соответствующим фрагментом белка. Экзон гена галактуроносилтрансферазы, содержащий стоп-кодон, был разделен на два. Для гена белка-шаперона предсказано две возможные изоформы.
Информацию о предсказанной экзон-интронной структуре предполагаемых генов можно увидеть на таблицах 2-6.
Таблица 2. Информация об экзонах гена галактуроносилтрансферазы (Probable galacturonosyltransferase) киви, предсказанного с помощью BLASTX
| Начало | Конец | Цепь | Тип |
| 2538 | 2750 | + | начальный |
| 4900 | 5061 | + | внутренний |
| 7979 | 8427 | + | внутренний |
| 8431 | 8560 | + | внутренний |
| 9328 | 9627 | + | внутренний |
| 10261 | 10539 | + | конечный |
Таблица 3. Информация об экзонах двух возможных изоформ гена белка-шаперона (Chaperone protein) киви, предсказанного с помощью BLASTX
| Начало | Конец | Цепь | Тип |
| 23428 | 23646 | + | начальный |
| 24175 | 24414 | + | внутренний |
| 25187 | 25384 | + | внутренний |
| 26608 | 26760 | + | внутренний |
| 26995 | 27309 | + | внутренний |
| 27383 | 27757 | + | внутренний |
| 28053 | 28976 | + | внутренний |
| 29539 | 29859 | + | конечный |
| Начало | Конец | Цепь | Тип |
| 25181 | 25384 | + | начальный |
| 26590 | 26757 | + | внутренний |
| 26995 | 27309 | + | внутренний |
| 27407 | 27643 | + | внутренний |
| 28071 | 28979 | + | внутренний |
| 29548 | 29748 | + | конечный |
Таблица 4. Информация об экзонах гена белка EID1-like F-box киви, предсказанного с помощью BLASTX
| Начало | Конец | Цепь |
| 34945 | 34214 | - |
Таблица 5. Информация об экзонах гена сахаропин дегидрогеназоподобной оксидоредуктазы (Probable mitochondrial saccharopine dehydrogenase-like oxidoreductase) киви, предсказанного с помощью BLASTX
| Начало | Конец | Цепь | Тип |
| 98053 | 97448 | - | конечный |
| 96106 | 95468 | - | начальный |
Таблица 6. Информация об экзонах гена белка, содержащего пентатрисопептидные повторы (Pentatricopeptide repeat-containing protein) киви, предсказанного с помощью BLASTX
| Начало | Конец | Цепь |
| 105880 | 103619 | - |
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016