| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Анализ качества и очистка чтений
Анализ качества чтений
Анализ и очистка чтений проводились для файла Ath_tae_CTTGTA_L003_R2_007.fastq с чтениями генома резуховидки. Данный файл весит слишком много, поэтому выложить его на сайт нет возможности.
С помощью программы FastQC проведён контроль качества скачанных чтений. Использовалась команда: "fastqc Ath_tae_CTTGTA_L003_R2_007.fastq". Отчёт программы можно увидеть здесь.
В отчёте программы FastQC можно найти информацию о исследуемых чтениях. В скачанном файле используется кодировка Sanger / Illumina 1.9. Всего файл содержит 4000000 чтений. Каждая постеловательность имеет длину в 101 нуклеотид. Чтения содержат 35% глутамина и цитозина.
Очистка чтений
Последовательности всех возможных адаптеров взяты из папки /P/y13/term3/block4/adapters и объединены в общий файл adapters.fa.
Далее проведена очистка чтений с помощью программы Trimmomatic. Удалены адаптеры (опция ILLUMINACLIP:adapters.fa:2:7:7), с каждого конца прочтения отрезаны нуклеотиды с качеством ниже 20 (опция TRAILING:20), удалены прочтения длиной меньше 50 нуклеотидов (опция MINLEN:50). При запуске программы указан phred33 формат fastq (для Sanger / Illumina 1.9).
В итоге, для запуска Trimmomatic использовалась команда: "java -jar /usr/share/java/trimmomatic.jar SE -phred33 Ath_tae_CTTGTA_L003_R2_007.fastq Ath_tae_CTTGTA_L003_R2_007_out.fastq ILLUMINACLIP:adapters.fa:2:7:7 TRAILING:20 MINLEN:50". В результате получен новый файл с чтениями - Ath_tae_CTTGTA_L003_R2_007_out.fastq, который снова проанализирован с помощью FastQC (команда "fastqc Ath_tae_CTTGTA_L003_R2_007_out.fastq"). Отчёт по очищенным чтениям пожно увидеть здесь.
Отчёт FastQC отличается от аналогичного для файла с неочищенными чтениями. Теперь файл содержит 3872176 чтений (количество уменьшилось не сильно, что говорит о том, что чтения изначально были довольно хорошими). Длина чтений теперь варьирует от 50 до 101, т. к. сначала чтения были обрезаны, а потом удалены те из них, которые имели длину меньше 50. Процентное содержание гуанина и цитозина осталось неизменным.
Качество нуклеотидов (т. е. точность определения нуклеотида в каждой позиции) в целом возросло, но не очень значительно. Это отражает график Per base sequence quality. Сравнение исходного и "улучшенного" графиков можно увидеть на рис. 1. До обработки Trimmomatic на графике были нуктеотиды, минимальная оценка качества которых попадала в красную зону, после улучшения их качество заметно улучшилось.
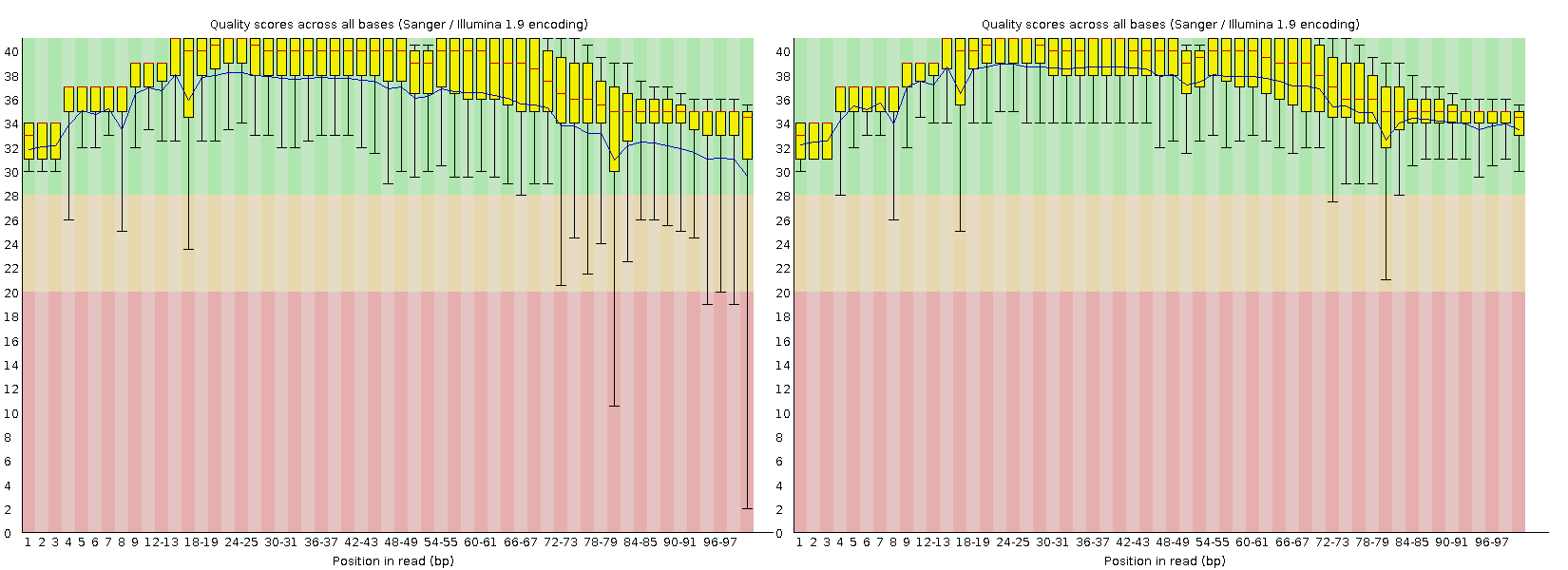
Рис. 1. Графики, отражающие качество каждого нуклеотида в чтениях (Per base sequence quality). Слева приведён график для исходного файла с чтениями, справа - график после улучшения с помощью программы Timmomatic. Графики получены с помощью программы FastQC.
График, отражающий частоту встречаемости каждого из четырёх нуклеотидов (Per base sequence content) совсем не изменился.
Распределение длин чтений изменилось. На графиках Sequence length distribution, приведённых на рис. 2, можно увидеть, что до улучшения все чтения имели длину 101, а после улучшения - от 50 до 101. Однако, всё равно почти все чтения имеют длину 100-101 нуктеотид, количество более коротких чтений незначительно.
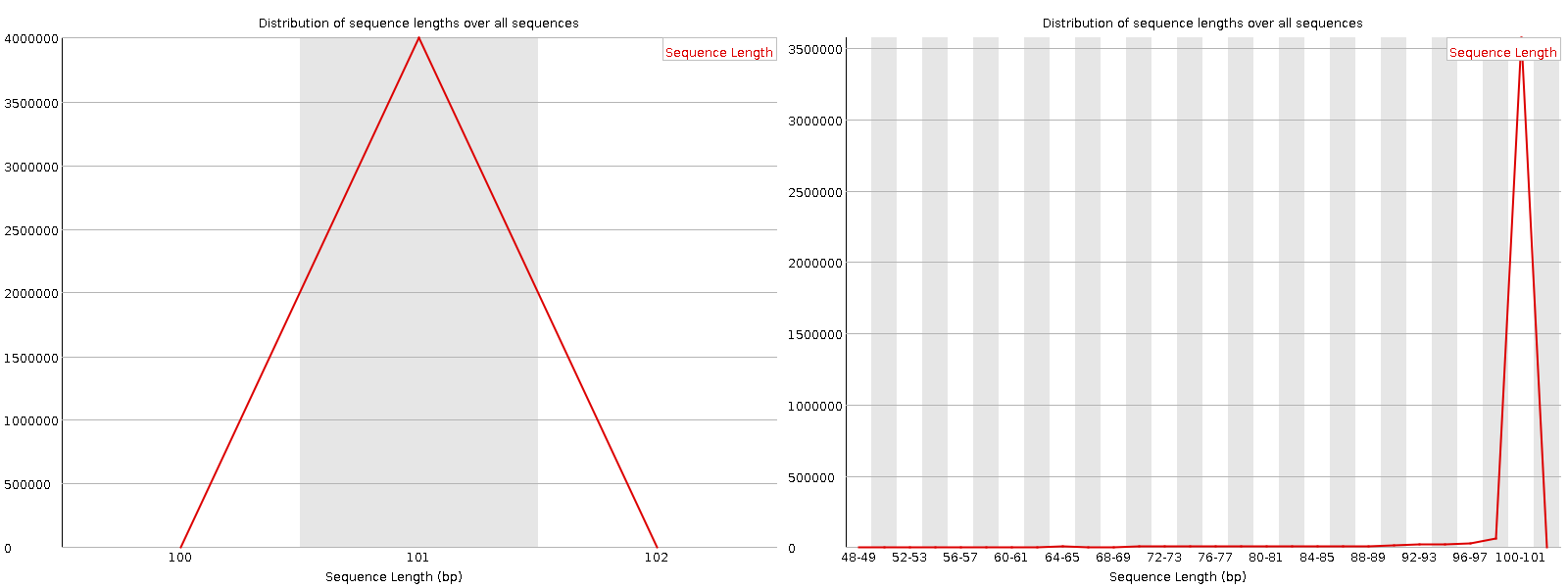
Рис. 2. Графики, отражающие распределение длин чтений (Sequence length distribution). Слева приведён график для исходного файла с чтениями, справа - график после улучшения с помощью программы Timmomatic. Графики получены с помощью программы FastQC.
Т. к. графики, отражающие качество нуклеотидов и частоту встречаемости каждого из четырёх нуктеотидов почти не изменились или не изменились вообще, и, т. к. количество и длина чтений уменьшились незначительно, можно предположить, что исходные чтения были достаточно качетвенными.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016