| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Standalone BLAST
Поиск некодирующих РНК (misc_RNA), аннотированных в одном штамме, в геноме другого штамма
С FTP-сервера NCBI скачан файл NC_000964.frn, содержащий последовательности всех аннотированных РНК штамма Bacillus_subtilis_168_uid57675, из этого файла с помощью команды "infoseq NC_000964.frn | grep 'misc_RNA' | awk '{ print $1 }' > usa.txt" получен файл-список usa.txt, содержащий универсальные адреса (USA) всех некодирующих РНК. Наконец, с помощью команды "seqret @usa.txt misc_rna.fasta" получен файл misc_rna.fasta, содержащий все некодирующие РНК штамма Bacillus_subtilis_168_uid57675.
Затем с того же FTP-сервера NCBI скачаны файлы NC_016047.fna и NC_016047.gbk, содержащие геном Bacillus_subtilis_spizizenii_TU_B_10_uid73967 (другого штамма этой же бактерии). На основе генома этого штамма с помощью команды "makeblastdb -in NC_016047.fna -dbtype nucl" создана нуклеотидная база данных локального BLAST-а в виде трёх файлов: NC_016047.fna.nhr, NC_016047.fna.nin и NC_016047.fna.nsq.
Далее с помощью алгоритма megablast произведён поиск misc_RNA штамма Bacillus_subtilis_168_uid57675 в геноме Bacillus_subtilis_spizizenii_TU_B_10_uid73967 с помощью команды: "blastn -task megablast -query misc_rna.fasta -db NC_016047.fna -out blast1.out -outfmt 7 -num_alignments 1".
В результате получен файл blast1.out, содержащий информацию о находках и на его основе составлена таблица, в которой для каждой misc_RNA указан номер, цепь (прямая или обратная на основе записи RefSeq; информация о цепи взята из соответствующего Bacillus_subtilis_168_uid57675 *.gbk файла), а также координаты начала и конца лучшего найденного гомолога. Полученную таблицу можно скачать здесь.
Поиск гомологов РНК Bacillus subtilis в геноме другой бактерии
Произведен поиск гомологов некодирующих РНК Bacillus_subtilis_168_uid57675 в геноме Bacillus_cereus_biovar_anthracis_CI_uid50615.
На FTP-сервере NCBI штамму Bacillus_cereus_biovar_anthracis_CI_uid50615 соответствует 4 файла в формате *.fna. Один из них (NC_014335.fna) содержит последовательность кольцевой хромосомы, а файлы NC_014331.fna, NC_014332.fna и NC_014333.fna соответствуют плазмидам. Т.к. полный геном бактерии включает в себя и плазмиды в том числе, для дальнейшего запуска BLAST создан общий файл B_cereus_biovar.fna, содержащий все 4 последовательности.
Далее на основе файла B_cereus_biovar.fna создана нуклеотидная база данных тремя способами и произведён поиск гомологов:
- с помощью алгоритма megablast (команда "blastn -task blastn -query misc_rna.fasta -db B_cereus_biovar.fna -out blast2_bn0.out -outfmt 7 -num_alignments 1 -evalue 0.001")
- с помощью алгоритма blastn с параметрами по умолчанию (команда "blastn -task megablast -query misc_rna.fasta -db B_cereus_biovar.fna -out blast2_m0.out -outfmt 7 -num_alignments 1 -evalue 0.001")
- с помощью blastn c измененными параметрами: длина слова = 4, награда за совпадение = 1, штраф за несовпадение = –1 (команда "blastn -task blastn -query misc_rna.fasta -db B_cereus_biovar.fna -out blast2_bn1.out -outfmt 7 -num_alignments 1 -word_size 4 -reward 1 -penalty -1 -evalue 0.001")
Затем произведён подсчёт находок с e-value меньше 0.001 для каждого запуска blast, результаты оформлены в форме таблицы, которую можно скачать здесь.
Поиск неправильно аннотированных генов программой blastx
С FTP-сервера NCBI скачаны последовательности предсказанных белков штамма Bacillus_subtilis_spizizenii_TU_B_10_uid73967 (файл NC_016047.faa). На основе этого файла создана белковая база данный локального BLAST-а (команда "makeblastdb -in NC_016047.faa -dbtype prot").
С помощью программы blastx, которая в качестве query принимает файл с нуклеотидными последовательностями, но производит поиск по белковой базе данных, был произведён поиск гомологов некодирующих РНК Bacillus_subtilis_168_uid57675 среди предсказанных белков Bacillus_subtilis_spizizenii_TU_B_10_uid73967. Выставлен порог e-value (меньше 0.001). Использовалась команда "blastx -query misc_rna.fasta -db NC_016047.faa -out blastx.out -outfmt 7 -num_alignments 1 -evalue 0.001". Полученный файл с находками - blastx.out.
Гомологи нашлись для misc_RNA под номерами 1, 25, 41, 46, 53, 57, 58, 61, 62. Почти все найденные гомологи оказались гипотетическими ("предполагаемыми") белками, кроме гомолога 1-ой misc_RNA (гомологичный белок - серил-тРНК синтаза) и 46-ой (гомолог - аспартокиназа).
Cхема генома в интересном месте
Составлены карты генов Bacillus_subtilis_168_uid57675 и Bacillus_subtilis_spizizenii_TU_B_10_uid73967 в окрестности участка с координатами 20042-23842. Выровненные карты генов можно увидеть здесь.
На последовательность BSU_misc_RNA_1 Bacillus_subtilis_168_uid57675 накладываются последовательности гена из локуса GYO_0016 и гена serS штамма Bacillus_subtilis_spizizenii_TU_B_10_uid73967. В прошлом задании blastx определил последовательность гена serS (кодирующего серил-тРНК синтазу) как гомологичную первой некодирующей РНК. Видимо так произошло, потому что последовательность из генома Bacillus_subtilis_spizizenii_TU_B_10_uid73967, гомологичная первой некодирующей РНК Bacillus_subtilis_168_uid57675, находилась рядом с последовательностью гена serS и её по ошибке аннотировали как кодирующую реальный белок.
Зависимость времени работы blastn от длины затравки
Проанализирована зависимость времени работы blastn от длины затравки на примере поиска гомологов некодирующих РНК Bacillus_subtilis_168_uid57675 в геноме Bacillus_cereus_biovar_anthracis_CI_uid50615. Использовалась команда "time blastn -task blastn -query misc_rna.fasta -db B_cereus_biovar.fna -out test.blastn -word_size x", где x - целое число в диапазоне от 4 до 16. Информация о времени работы blastn в зависимости от длины затравки представлена на таблице 1. Графическое представление зависимости можно увидеть на рис. 1.
Таблица 1. Зависимость времени работы blastn от длины затравки
| Длина затравки | Время работы blastn (в секундах) |
| 4 | 82,909 |
| 5 | 21,596 |
| 6 | 6,13 |
| 7 | 2,277 |
| 8 | 1,277 |
| 9 | 1,121 |
| 10 | 0,894 |
| 11 | 0,827 |
| 12 | 0,767 |
| 13 | 0,751 |
| 14 | 0,732 |
| 15 | 0,707 |
| 16 | 0,656 |
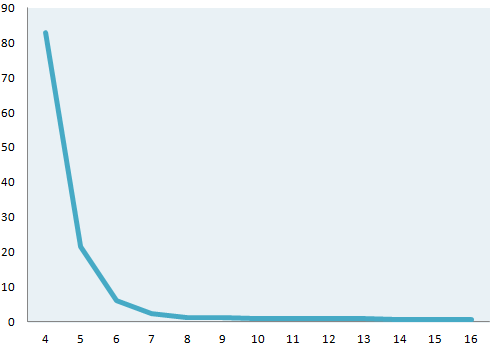
Рис. 1. Графическое представление зависимости времени работы blastn от длины затравки. По оси абсцисс отложена длина затравки, по оси ординат - время в секундах.
Видно, что при увеличении длины затравки время, затрачиваемое на поиск гомологов, уменьшается.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016