| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Ферменты. База KEGG. Работа с KEGG ORTHOLOGY
Цель данного задания было проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками.
Для работы выбрана реакция с ЕС-номером 1.2.99.8 пентозофосфатного пути (pentose phosphate pathway). Описание данного метаболического пути было найдено в базе данных KEGG, его графическое изображение можно увидеть на рис. 1.
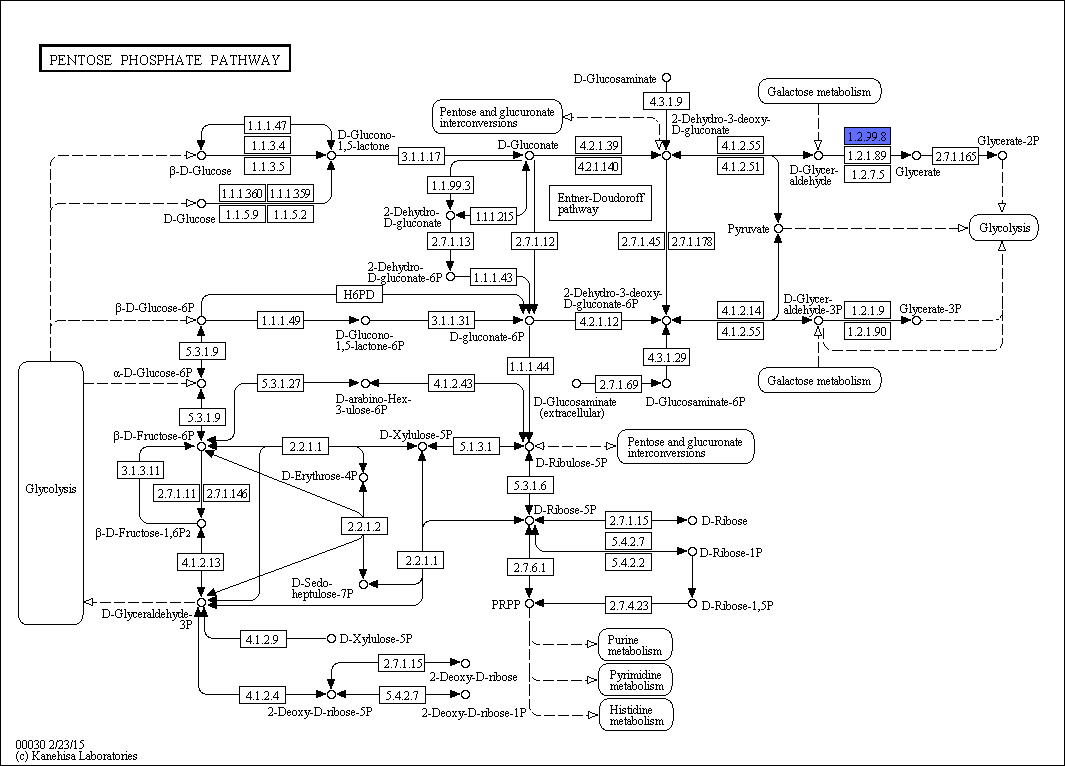
Рис. 1. Изображение пентозофосфатного пути. Синим выделена рассматриваемая реакция 1.2.99.8. Изображение получено с помощью базы данных KEGG.
Реакцию 1.2.99.8 катализирует три ортологических ряда белков: K18020 (большая субъединица глицеральдегид дегидрогеназы; 20 белков Uniprot), K18021 (средняя субъединица глицеральдегид дегидрогеназы; 19 белков) и K18022 (малая субъединица глицеральдегид дегидрогеназы; 18 белков).
Далее были получены идентификаторы белков каждого ряда из таблицы идентификаторов (чтобы открыть эту таблицу надо нажать на кнопку "Uniprot" на странице с описанием нужного ряда). Список идентификаторов был подан на вход сервису "Retrieve/ID mapping" базы данных Uniprot. В результате были получены файлы с fasta-последовательностями нужных белков. Названия последовательностей были отредактированы с помощью скрипта скрипта, все последовательности были объединены в общий файл и выравнены с помощью сервера Muscle.
Затем полученное выравнивание было открыто в программе Jalview, покрашено ClustalX. Проект Jalview можно скачать здесь. Изображение выравнивания можно увидеть на рис. 2.
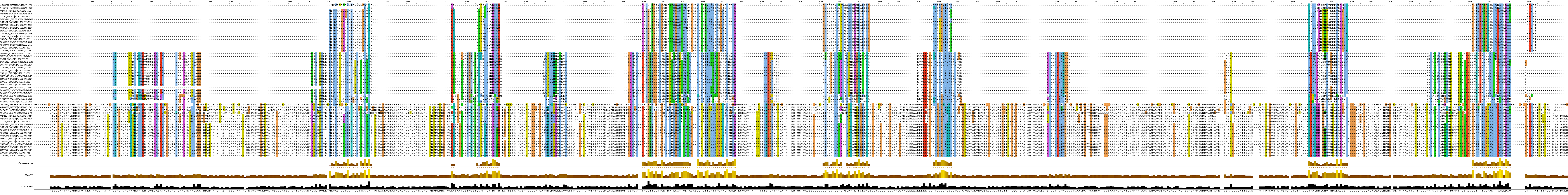
Рис. 2. Выравнивание последовательностей белков, катализирующих реакцию 1.2.99.8. Построено с помощью программы Muscle. Изображение получено с помощью программы Jalview. Окраска ClustalX.
Далее с помощью программы MEGA было построено филогенетическое дерево выбранных белков (использовался метод Neighbor-Joining с 100 бутстреп-репликами). Изображение получившегося дерева можно увидеть на рис. 3. PDF-файл с деревом можно скачать здесь.
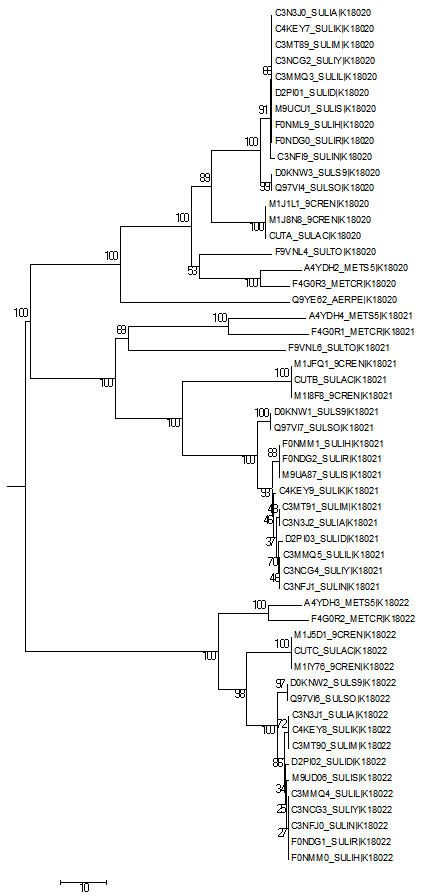
Рис. 3. Филогенетическое дерево белков, катализирующих реакцию 1.2.99.8. Получено с помощью программы MEGA (метод Neighbor-Joining с 100 бутстреп-репликами) на основе Muscle-выравнивания.
Заметно, что дерево разделяется на три большие ветви, имеющие большую поддержку по результатам анализа bootstrap. Эти ветви соответствуют ортологическим рядам. На дереве нет ветвей, длины которых бы существенно отличались от длин других тривиальных ветвей. Однако, на основе этих характеристик построенного дерева нельзя сделать вывод о гомологичности всех исследуемых белков.
Если внимательно посмотреть на выравнивание их последовательностей (рис. 2), то можно заметить, что, хотя в выравнивании есть некоторое количество участков, сходных между группами, в основном хорошо выравнены только белки в пределах каждого ортологического ряда, но выравнивания белков из разных групп очень плохие, к тому же, длина белков, входящих в разные ортологические ряды, сильно различается (есть группа длинных белков, средних и коротких). Ещё один факт, заставляющий усомниться в предположении о гомологичности этих белков: судя по названиям, ортологические ряды отвечают трём субъединицам одного комплекса, поэтому, вероятнее всего, они должны достаточно сильно различаться.
Таким образом, эти три ортологических ряда представляют собой группы белков, отвечающих трём разным субъединицам одного фермента (глицеральдегид дегидрогеназы), катализирующего реакцию 1.2.99.8. Белки этих ортологических рядов вероятно не являются гомологами. Именно тот факт, что три исследуемых группы отвечают разным субъединицам комплекса, вероятно, и явился причиной того, что дерево распалось на три ветви с такой высокой поддержкой (чёткие различия в последовательностях этих групп белков привели к очевидному разделению дерева на три ветви).
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016