| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Геномное окружение. База данных STRING
С помощью базы данных STRING можно определить белки, с которыми связан (взаимодействует) исследуемый белок. Белок Rhodanese бактерии Azotobacter vinelandii DJ имеется в базе данных STRING (ген rhdA), поиск проводился на основе названия белка и организма, которому он принадлежит. На рис. 1 можно увидеть граф взаимодействий этого белка с белками, связанными с ним на первом уровне близости.
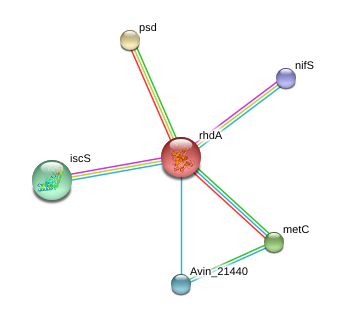
Рис. 1. Изображение "графа взаимодействий" белка Rhodanese бактерии Azotobacter vinelandii DJ и других белков (связанных с Rhodanese на первом уровне близости) в режиме evidence. Зелёным цветом покрашены рёбра графа, соединяющие белки, взаимодействие между которых определено на основе соседства их генов, красным - на основании сшивки генов, сиреневым - на основе экспериментальных данных, бирюзовым - на основе информации из базы данных, желтым - на основе литературных данных. Изображение получено с помощью базы данных STRING.
Белок Rhodanese взаимодействует с пятью белками, информацию о которых можно увидеть в таблице 1.
Таблица 1. Информация о белке Rhodanese бактерии Azotobacter vinelandii DJ и его окружении.
| Ген/локус | Название на английском | Название на русском | Длина белка (аа) | Функция (по данным KEGG и STRING) | ЕС номер катализируемой реакции | Метаболический путь (по данным KEGG) |
| rhdA | Rhodanese | Роданеза | 271 | Тиосульфат/3-меркаптопируват сульфуртрансфераза | 2.8.1.1 | Метаболизм серы, Бактериальный метаболизм в разных условиях, Система передачи серы |
| psd | Phosphatidylserine decarboxylase | Фосфатидилсерин декарбоксилаза | 286 | - | 4.1.1.65 | Метаболизм глицерофосфолипидов |
| metC | Cystathionine beta-lyase | Цистатионин бета-лиаза | 388 | - | 4.4.1.8 | Метаболизм цистеина и метионина, Метаболизм селеносодержащих соединений, Биосинтез вторичных метаболитов, Биосинтез аминокислот |
| iscS | Cysteine desulfurase | Цистеин десульфураза | 404 | Катализирует удаление серы из цистеина, чтобы получить аланин. | 2.8.1.7 | Метаболизм тиамина, Система передачи серы |
| Avin_21440 | Aspartate aminotransferase | Аспартат аминотрансфераза | 405 | - | 2.6.1.1 | Метаболизмы биосинтеза разных аминокислот, Биосинтез новобиоцина, Биосинтез вторичных метаболитов, Биосинтез антибиотиков, Метаболизм 2-оксокарбоновой кислоны, Биосинтез аминокислот |
| nifS | Nitrogen fixation cysteine desulfurase | Цистеин десульфураза фиксации азота | 402 | Катализирует удаление атомов серы из цистеина для получения аланина. | 2.8.1.7 | Метаболизм тиамина, Система передачи серы |
Данные белки не входят в один белковый комплекс и не катализируют последовательные реакции одного метаболического пути.
Белки, кодируемые генами rhdA, nifS и iscS участвуют в метаболизме серы, причём nifS и iscS катализируют одну и ту же реакцию (2.8.1.7) по данным KEGG, имеют практически одинаковые названия и имеют одну и ту же функцию. Белок, кодируемый в локусе Avin_21440 и белок, кодируемый геном metC участвуют в метаболизме аминокислот.
Получена информация о геномном окружении гена белка Rhodanese бактерии Azotobacter vinelandii DJ. Дерево с информацией о геномном окружении с точностью до филума можно увидеть на рис. 2. Полное дерево можно скачать здесь.

Рис. 2. "Геномное окружение" (genome neighborhood) для белка Rhodanese бактерии Azotobacter vinelandii DJ с точностью до филума. "Полоски" красного цвета соответствуют гену rhdA (Роданеза), зелёные - metC (Цистатионин бета-лиаза), жёлтые - psd (Фосфатидилсерин декарбоксилаза), бирюзовые - iscS (Цистеин десульфураза), синие - Avin_21440 (Аспартат аминотрансфераза), сине-сиреневые - nifS (Цистеин десульфураза фиксации азота). Изображение получено с помощью базы данных STRING.
Найденные белки встречаются рядом только у бактерий (для архей и эукариот совместная встречаемость не выявлена).
На общем дереве можно видеть, что гены rhdA и psd находятся рядом у некоторого количества гамма-протеобактерий (особенно у родов Pseudomonas и Actinobacter), из чего можно предположить, что они входят в один оперон. Также, гены rhdA и metC расположены рядом в геномах некоторых альфа-протеобактерий, что аналогично даёт возможность предполагать, что у этих организмов они включены в один оперон. Также, для этих двух пар есть примеры сшивок, это является ещё одним доводом к выводу об их включении в опероны, а также из этого можно предполагать, что у некоторых организмов эти белки могут быть ассоциированы.
Получен график "совместной встречаемости" (co-occurrence) для белка Rhodanese бактерии Azotobacter vinelandii DJ, который с точностью до филума предствален на рис. 3.

Рис. 3. График "совместной встречаемости" (co-occurrence) для белка Rhodanese бактерии Azotobacter vinelandii DJ с точностью до филума. Изображение получено с помощью базы данных STRING.
Чем темнее квадратик на графике совместной встречаемости, тем белок таксона более гомологичен одному из наших белков. Полнота квадратика отражает встречаемость соответствующего белка (или его гомолога) в таксоне. Поэтому, неполные тёмные квадратики в строке, соответствующей протеобактериям (к которым и относится наша бактерия Azotobacter vinelandii DJ) означают, что найденные у протеобактерий белки гомологичны нашим (что логично, т. к. найденные белки как раз и являются белками протеобактерии Azotobacter vinelandii DJ), но эти белки присутствуют только у части протеобактерий, а у остальных эти белки отсутствуют.
Исходя из рис. 3 гомологи (хоть и разной достоверности) всех шести белков присутствуют во многих таксонах. Это бактерии классов Proteobacteria, Firmicutes, Actinobacteria, Cyanobacteria, Spirochaetales, Fusobacteriaceae и др., эукариотические организмы классов FungiMetazoa group, Viridiplantae, Stramenopilus, Amoebozoa, Trypanosomatidae. Причём гомологи белков, кодируемые генами iscS, Avin_21440, nifS распространены во всех таксонах, поэтому говорить о существовании паттерна совместной встречаемости этих белков с rhdA нельзя. Для psd и metC также не прослеживается наличие совместной встречаемости с rhdA. Т. к. ранее было выяснено, что функции белков различаются (rhdA, iscS, nifS задействованы в метаболизме серы, metC, Avin_21440 - в метаболизме аминокислот, а psd - в метаболизме глицерофосфолипидов), данные белки не являются частью одного процесса. Однако, интересно, что совместная встречаемость iscS и nifS коррелирует с наличием сходной функции у этих белков (тоесть можно считать, что эти два белка вовлечены в один процесс).
То, что во многих таксонах эти шесть белков присутствуют одновременно скорее связано с тем, что они участвуют в достаточно важных процессах в организме.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 07.12.2016