| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Расчет вторичной структуры
В рамках данного практикума производился расчёт вторичной структуры для пептидогликан связывающего белка Enterococcus faecium (pdb-код 1zat). Данный белок содержит 6 спиралей, и 6 бета листов, состоящих из нескольких бета-тяжей (в общей сложности структура содержит 18 бета-тяжей). Предсказание границ вторичных структур белка производили с помощью Stride.
Текстовую выдачу Stride можно скачать здесь, визуальная выдача представлена на рис. 1. Stride обнаружил в белке 5 альфа-спиралей и 2 310-спирали, одна из них (координаты 349-351) очень короткая и вообще никак не отражена в аннотации PDB. Помимо этого Stride определил 18 бета-тяжей. В таблице 1 представлено сравнение границ 2-ух альфа-спиралей и 2-ух бета-тяжей по данным PDB и Stride.
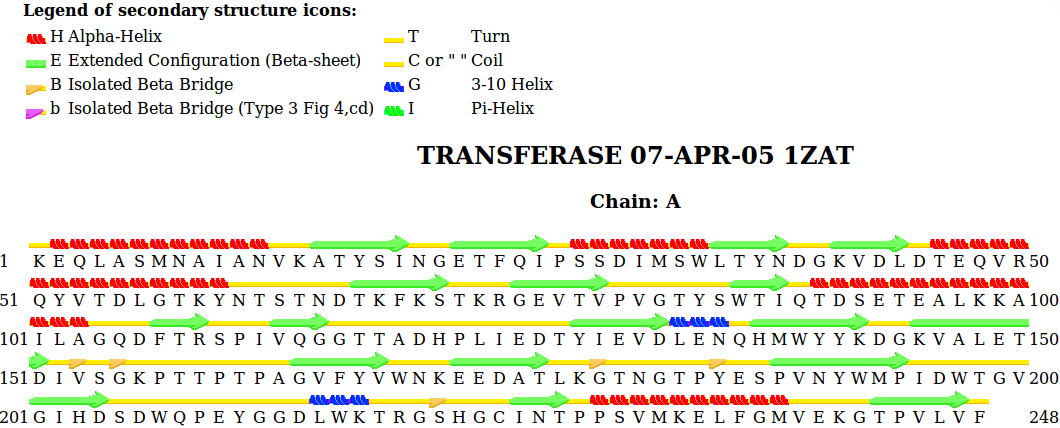
Рис. 1. Визуальная выдача Stride для пептидогликан связывающего белка Enterococcus faecium (pdb-код 1zat)
Таблица 1. Сравнение границ двух ɑ-спиралей и двух β-тяжей пептидогликан связывающего белка Enterococcus faecium по данным PDB и Stride
| Вторичная структура | Границы в PDB | Границы по Stride |
| ɑ-спираль | 261-277 | 262-276 |
| ɑ-спираль | 446-457 | 447-456 |
| β-тяж | 231-235 | 231-235 |
| β-тяж | 406-412 | 406-412 |
Stride правильно рассчитал границы двух рассмотренных бета-тяжей, границы альфа-спиралей также отличаются от границ, указанных в PDB незначительно (на 1 аминокислоту).
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 29.05.2015