| Учебный сайт Екатерины Швецовой | |||
| Главная | Обо мне | Семестры | Ссылки |
| 1 семестр | 2 семестр | 3 семестр | 4 семестр | 7 семестр | 8 семестр |
Совмещение структур
Совмещение структур белка 1SRN и его гомологов
С помощью сервера PDBeFold были найдены структурные гомологи для белка рибонуклеазы (идентификатор PDB: 1SRN). Было отобрано 4 гомолога, их характеристика представлена в таблице 1.
Таблица 1. Выбранные структурные гомологи белка 1SRN
| Идентификатор PDB | Описание | RMSD | Длина выравнивания от белка 1SRN, (%) |
| 1srn:A | THE REFINED CRYSTAL STRUCTURE OF A FULLY ACTIVE SEMISYNTHETIC RIBONUCLEASE AT 1.8 ANGSTROMS RESOLUTION | 0 | 100 |
| 3lje:A | The X-ray structure of zebrafish RNase5 | 1.36 | 82,2 |
| 2vq8:A | RNASE ZF-1A | 1.35 | 80,5 |
| 2p6z:A | ENZYMATIC AND STRUCTURAL CHARACTERISATION OF AMPHINASE, A NOVEL CYTOTOXIC RIBONUCLEASE FROM RANA PIPIENS OOCYTES | 1.67 | 72,9 |
| 1e21:A | RIBONUCLEASE 1 DES1-7 CRYSTAL STRUCTURE AT 1.9A | 0.88 | 84,7 |
Далее пять структур (4 гомолога + наш белок) были поданы на вход PDBeFold для построения структурного выравнивания. Визуализацию получившегося выравнивания можно увидеть на рис. 1, соответствующее ему выравнивание последовательностей можно скачать здесь.
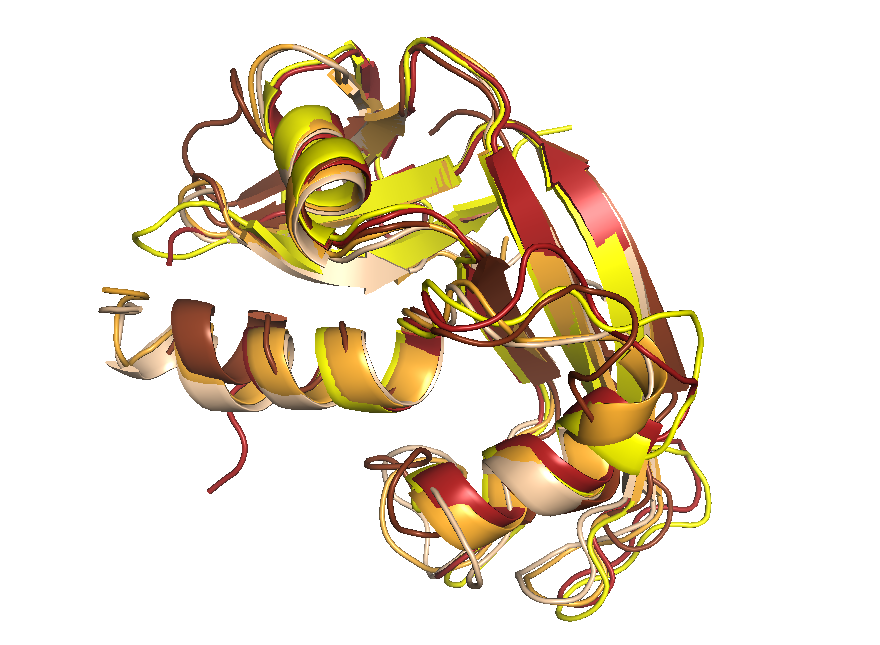
Рис. 1. Совмещение структур гомологов белка 1SRN. Рисунок получен с помощью программы PyMOL.
Затем были скачаны последовательности 5-ти рассматриваемых белков и выравнены с помощью MUSCLE. Получившееся выравнивание можно скачать здесь.
Оба выравнивания были открыты в программе Jalview, соответствующие изображения можно увидеть на рис. 2 и 3.

Рис. 2 Множественное выравнивание гомологов белка 1SRN полученное по структурному совмещению. Раскраска BLOSUM62 (By Conservation 30%)

Рис. 3 Множественное выравнивание гомологов белка 1SRN полученное с помощью MUSCLE. Раскраска BLOSUM62 (By Conservation 30%)
Видно, что большинство остатков, выравненных в структурном выравнивании, выравнены и в выравнивании MUSCLE, однако, есть участки, в которых это не так. Например участок в районе позиции 90 выравниваний (см. рис. 4). Видно, что выравнивание структуры 1p6z с остальными выглядит по-разному. Например, в структурном выравнивании валин структуры 2p6z выравнен со столбиком аспарагинов в остальных структурах, чего не наблюдается в выравнивании MUSCLE.

Рис. 4. Участок множественных выравниваний гомологов белка 1SRN полученных с помощью MUSCLE (сверху) и по структурному совмещению (снизу). Раскраска BLOSUM62 (By Conservation 30%)
Пара белков, для которых гибкое структурное выравнивание включает существенно больше остатков, чем жесткое
В примерах сервиса POSA было приведено сравнение структур доменов 2-RRM (узнающих РНК), для которых гибкое выравнивание лучше, чем жесткое. Было взято 2 белка из примера (1cvj:A и 1fnx:H) и для них были построены гибкое (см. рис. 5) и жесткое (см. рис. 6) выравнивания с помощью сервиса FATCAT.
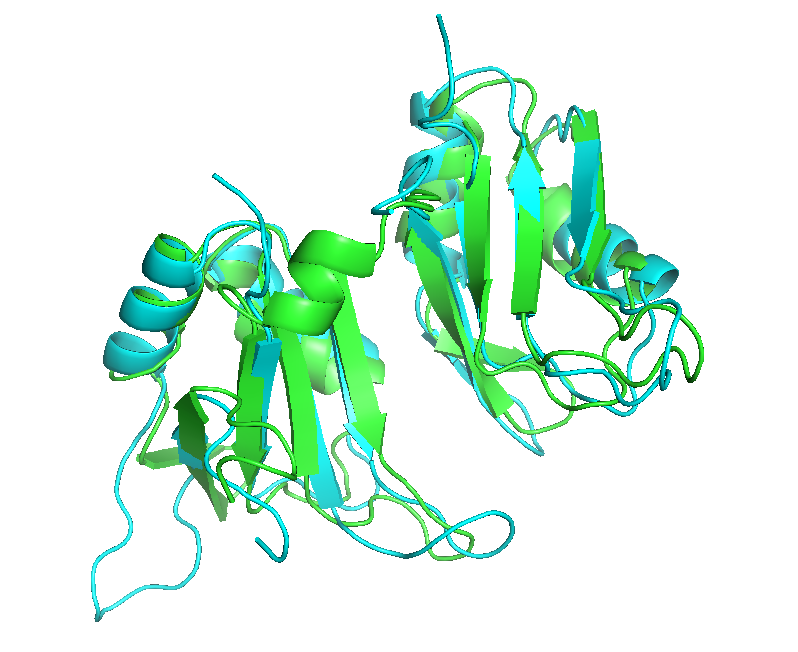
Рис. 5. Гибкое структурное выравнивание 1cvj:A и 1fnx:H. Рисунок получен с помощью программы PyMOL.
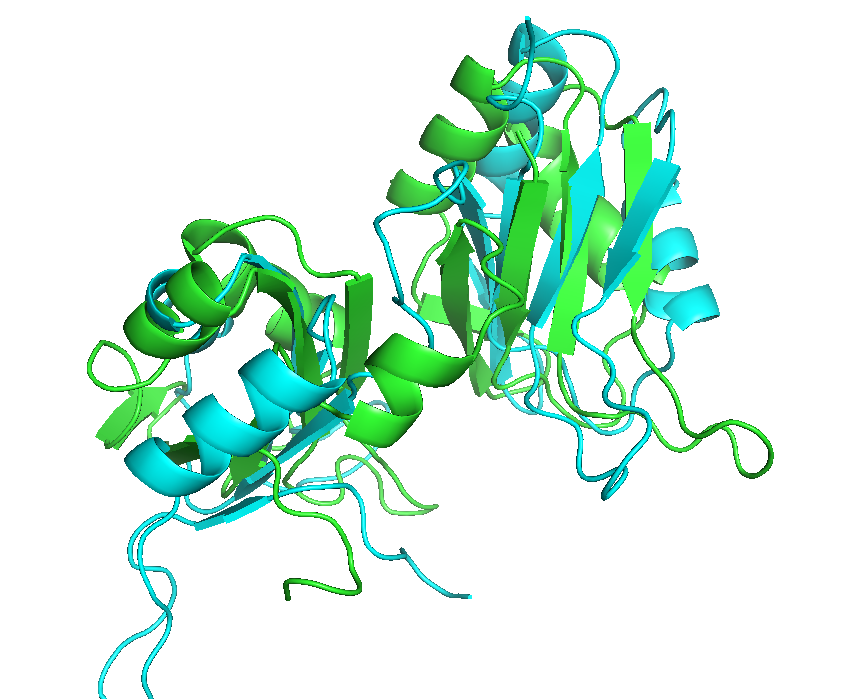
Рис. 6. Жесткое структурное выравнивание 1cvj:A и 1fnx:H. Рисунок получен с помощью программы PyMOL.
На рисунках 4 и 5 видно, что гибкое выравнивание даёт хорошее совмещение доменов белка, а жесткое не даёт нормального совмещения.
© Shvetsova Ekaterina, FBB MSU, 2013
Дата последнего изменения: 29.05.2015