Различные выравнивания и их анализ
1. Отличия между двумя разными выравниваниями
В данной части практикума были сравнены два выравнивания: выравнивание с помощью BLAST и оптимальное локальное выравнивание с помощью программы water. В качестве белков для сравнения были выбраны ферменты двух ретровирусов: интеграза ВИЧ-1 (Human immunodeficiency virus 1 - HIV-1) и интеграза Пенообразующего вируса человека (Human foamy virus - HFV).
Интеграза - фермент, катализирующий включение вирусного ДНК в хромосому клетки-хозяина. Ретровирусные интегразы входят в сложные прединтеграционные комплексы, в составе которых они и проявляют свою ферментативную активность [1]. Интегразы ВИЧ-1 и HFV транслируются в составах полипротеинов Gag-Pol и Pro-Pol соответствующих вирусов (AC: P04585; AC: P14350).
С помощью команды seqret 'sw:P14350[752:1143]' 'foam_in.fasta' и аналогичной для полипротеина из ВИЧ-1 (интеграза находится на участке [1148:1435]) были получены последовательности ферментов. Ссылки на полученные файлы: hiv_in.fasta, foam_in.fasta. Далее было выполнено два выравнивания при стандартных настройках: ссылка на выравнивание BLAST, ссылка на выравнивание water. Выравнивания были импортированы в Jalview для более удобного поиска различий. Результаты представлены на Fig.1. и Fig.2. Из BLAST было выбрано лучшее выравнивание, оно же представлено на Fig.1.
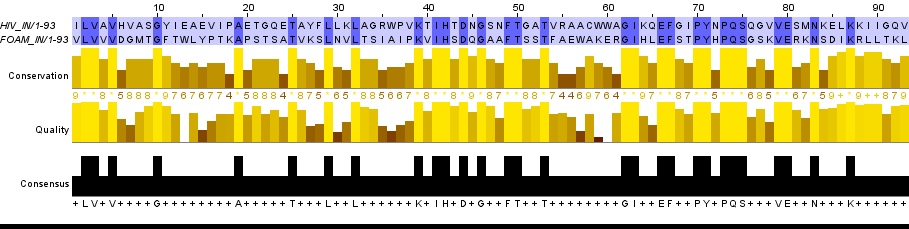
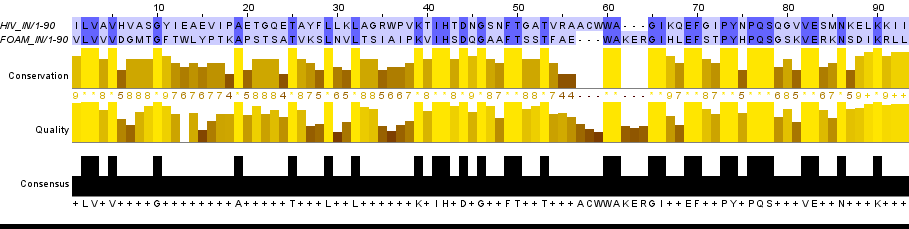
Оба выравнивания начинаются с 73 аминокислотного остатка интегразы ВИЧа и 142 остатка интегразы HFV. В целом выравнивания очень схожи, но есть некоторые различия:
- Выравнивания немного отличаются по длине: в выравнивании BLAST сопоставлены друг другу 93 позиции последовательности ферментов, в water всего 90.
- До 56-ого столбца выравнивания полностью совпадают. Различаются отрезки 57-61 в BLAST и 57-64 в water. В water появляются два инделя длиной 3, за счет этого достигается совпадение 60 и 61 столбцов. В случае BLAST выравнивание идет без гэпов вообще.
- В выравнивание BLAST входит 3 его последних столбца, которых нет в выравнивании water: G-T, Q-K, V-L. В матрице BLOSUM 62, которая использовалась в выравниваниях, эти сопоставления имеют веса -2, 1, 1 соответственно [2].
Конкретные различия в выравниваниях представлены в таблице 1.
| № столбца в BLAST | № столбца в water | Пара в выравнивании BLAST | Пара в выравнивании water | Коэффициент для пары из BLAST | Коэффициент для пары из water |
|---|---|---|---|---|---|
| 57 | 57 | Ala128 - Glu197 | Ala128 - Gap | -1 | Штраф за гэп |
| 60 | 60 | Trp132 - Glu201 | Trp132 - Trp198 | -3 | 11 |
| 59 | 62 | Trp131 - Lys200 | Gap - Lys200 | -3 | Штраф за гэп |
2. Карта локального сходства. Сравнение с оптимальным локальным выравниванием
Для данной части работы были выбраны уже упомянутые выше полипротеины тех же вирусов: Gag-Pol из ВИЧ-1 и Pro-Pol из HFV. Информация об этих белках представлена в таблице 2.
| ID | AC | Рекомендуемое имя | Длина, а.о. |
|---|---|---|---|
| POL_HV1H2 | P04585 | Gag-Pol polyprotein | 1435 |
| POL_FOAMV | P14350 | Pro-Pol polyprotein | 1143 |
Теперь построим также парное выравнивание в BLAST, но сравнивать будем полипротеины полностью. В параметрах BLAST был изменем "Word size" на 2, остальные настройки использовались по умолчанию. Ссылка на полученные выравнивания; на картинке 3 представлена карта выданных выравниваний.
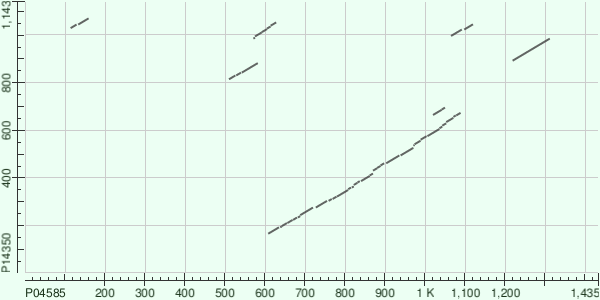
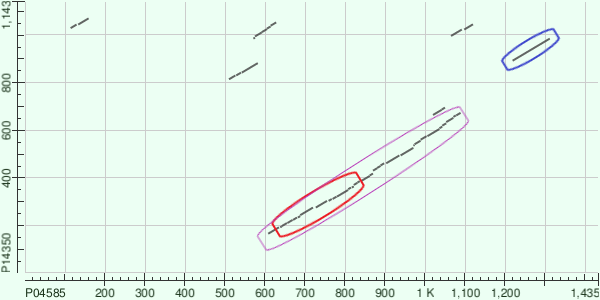
Лучшее по весу выравнивание, обозначенное фиолетовым цветом на картинке 4, сопоставляет 609-1089 остатки из Gag-Pol и 166-673 остатки из Pro-Pol. Посмотрев на строки FT записей полипротеинов, можно заметить, что в рассматриваемое выравнивание в обоих случаях попадает участок будущей обратной транскриптазы: 198-363 а.о. для HFV и 631-821 а.о. из ВИЧа. Область этого фермента обозначена на картинке 4 красным цветом. Данная часть графика близка к диагональному виду, что говорит о вероятной гомологии этих двух ферментов. Также со строками FT согласуется полностью диагональный участок обозначенный синим цветом. Этот отрезок находится в области интегразы в обоих политротеинах и свидительствует о гомологии ферментов.
Ниже представлено лучшее по весу выравнивание. Query == полипротеин Gag-Pol из HIV-1; Sbjct == полипротеин из HFV. Характеристики данного выравнивания приведены в таблице 3.
Query 609 KQWPLTEEKIKALVEICTEMEKEGKISKIGPENP-YNTPVFAIKKKDSTKWRKLVDFREL 667
KQ+P+ + ++ + ++ K+G ++ P+N NTPV+ + K D +WR ++D+RE+
Sbjct 166 KQYPINPKAKPSIQIVIDDLLKQGVLT---PQNSTMNTPVYPVPKPDG-RWRMVLDYREV 221
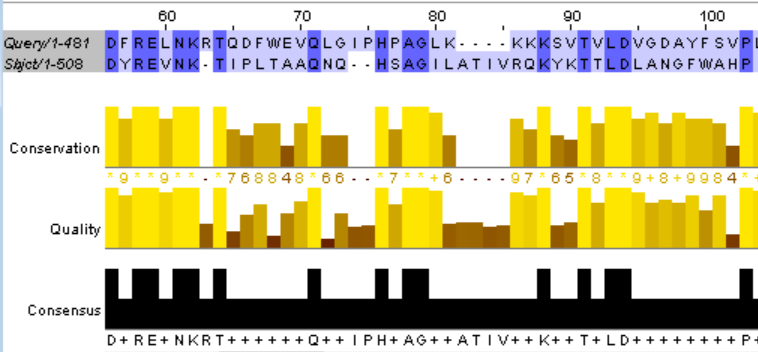
Query 668 NKRTQDFWEVQLGIPHPAGLK----KKKSVTVLDVGDAYFSVPLDEDFRKYTAFTIPSIN 723
NK T Q H AG+ ++K T LD+ + +++ P+ + TAFT
Sbjct 222 NK-TIPLTAAQNQ--HSAGILATIVRQKYKTTLDLANGFWAHPITPESYWLTAFTWQ--- 275
Query 724 NETPGIRYQYNVLPQGWKGSPAIFQSSMTKILEPFRKQNPDIVIYQYMDDLYVGSDLEIG 783
G +Y + LPQG+ SPA+F + + +L K+ P++ + Y+DD+Y+ D +
Sbjct 276 ----GKQYCWTRLPQGFLNSPALFTADVVDLL----KEIPNVQV--YVDDIYLSHD-DPK 324
Query 784 QHRTKIEELRQHLLRWGLTTPDKKH---QKEPPFLWMGYELH------PDKWTVQPI-VL 833
+H ++E++ Q LL+ G KK QK FL G+ + D + + + +
Sbjct 325 EHVQQLEKVFQILLQAGYVVSLKKSEIGQKTVEFL--GFNITKEGRGLTDTFKTKLLNIT 382
Query 834 PEKDSWTVNDIQKLVGKLNWASQIYPGIK--VRQLCKLL------------RGTKALTEV 879
P KD + +Q ++G LN+A P V+ L L+ TK L V
Sbjct 383 PPKD---LKQLQSILGLLNFARNFIPNFAELVQPLYNLIASAKGKYIEWSEENTKQLNMV 439
Query 880 I-PLTEEAELE--LAENREILKEPVHGVYYDPSKDLIAEIQKQGQGQWTYQIYQEPFKNL 936
I L + LE L E R ++K V PS + + G+ Y Y L
Sbjct 440 IEALNTASNLEERLPEQRLVIK-----VNTSPSAGYVRYYNETGKKPIMYLNYVFSKAEL 494
Query 937 KTGKYARMRGAHTNDVKQLTEAVQKITTESIVIWG---------KTP---KFKLPIQKET 984
K++ + T K L +A+ + I+++ KTP + LPI+ T
Sbjct 495 ---KFSMLEKLLTTMHKALIKAMDLAMGQEILVYSPIVSMTKIQKTPLPERKALPIRWIT 551
Query 985 WETWW----TEYWQATWIPEWEFVNTPPLVKLWYQLEKEPIVGAETFYVDGAANRE---T 1037
W T+ ++ +PE + + P + K P FY DG+A + T
Sbjct 552 WMTYLEDPRIQFHYDKTLPELKHI--PDVYTSSQSPVKHPSQYEGVFYTDGSAIKSPDPT 609
Query 1038 KLGKAG----YVTNRGRQKV-----VTLTDTTNQKTELQAIYL----ALQDSGLEVNIVT 1084
K AG + T + +V + L + T Q E+ A+ AL+ G V ++T
Sbjct 610 KSNNAGMGIVHATYKPEYQVLNQWSIPLGNHTAQMAEIAAVEFACKKALKIPG-PVLVIT 668
Query 1085 DSQYA 1089
DS Y
Sbjct 669 DSFYV 673
| Score | Expect value | Identities | Совпадений с положительнми коэффициентами | Gaps |
|---|---|---|---|---|
| 83.6 bits(205) | 6e-20 | 141/545(26%) | 240/545(44%) | 101/545(18%) |
Далее попробуем выровнять эти полипротеины программой water с параметрами по умолчанию. Часть получившегося выравнивание, которая пересекается с выравниванием BLAST, представлена ниже; ссылку на полный файл выдачи можно найти тут.
POL_HV1H2 609 KQWPLTEEKIKALVEICTEMEKEGKISKIGP 639
||:|:..:...::..:..::.|:|.:: |
POL_FOAMV 166 KQYPINPKAKPSIQIVIDDLLKQGVLT---P 193
POL_HV1H2 640 EN-PYNTPVFAIKKKDSTKWRKLVDFRELNKRTQDFWEVQLGIP------ 682
:| ..||||:.:.|.|. :||.::|:||:||. ||
POL_FOAMV 194 QNSTMNTPVYPVPKPDG-RWRMVLDYREVNKT----------IPLTAAQN 232
POL_HV1H2 683 -HPAG----LKKKKSVTVLDVGDAYFSVPLDEDFRKYTAFTIPSINNETP 727
|.|| :.::|..|.||:.:.:::.|:..:....|||| ..
POL_FOAMV 233 QHSAGILATIVRQKYKTTLDLANGFWAHPITPESYWLTAFT-------WQ 275
POL_HV1H2 728 GIRYQYNVLPQGWKGSPAIFQSSMTKILEPFRKQNPDIVIYQYMDDLYVG 777
|.:|.:..||||:..|||:|.:.:..:| |:.|::.: |:||:|:.
POL_FOAMV 276 GKQYCWTRLPQGFLNSPALFTADVVDLL----KEIPNVQV--YVDDIYLS 319
POL_HV1H2 778 SDLEIGQHRTKIEELRQHLLRWGLTTPDKKH---QKEPPFLWMGYELH-- 822
.| :..:|..::|::.|.||:.|.....||. ||...|| |:.:.
POL_FOAMV 320 HD-DPKEHVQQLEKVFQILLQAGYVVSLKKSEIGQKTVEFL--GFNITKE 366
POL_HV1H2 823 ----PDKWTVQPI-VLPEKDSWTVNDIQKLVGKLNWASQIYPGIK--VRQ 865
.|.:..:.: :.|.|| :..:|.::|.||:|....|... |:.
POL_FOAMV 367 GRGLTDTFKTKLLNITPPKD---LKQLQSILGLLNFARNFIPNFAELVQP 413
POL_HV1H2 866 LCKLL------------RGTKALTEVIPLTEEA---ELELAENREILKEP 900
|..|: ..||.|..||.....| |..|.|.|.::|
POL_FOAMV 414 LYNLIASAKGKYIEWSEENTKQLNMVIEALNTASNLEERLPEQRLVIK-- 461
POL_HV1H2 901 VHGVYYDPSKDLIAEIQKQGQGQWTYQIYQEPFKNLKTGKYARMRGAHTN 950
|...||...:....:.|:....|..|......| |::.:....|.
POL_FOAMV 462 ---VNTSPSAGYVRYYNETGKKPIMYLNYVFSKAEL---KFSMLEKLLTT 505
POL_HV1H2 951 DVKQLTEAVQKITTESIVIWG---------KTP---KFKLPIQKETWETW 988
..|.|.:|:.....:.|:::. ||| :..|||: |.||
POL_FOAMV 506 MHKALIKAMDLAMGQEILVYSPIVSMTKIQKTPLPERKALPIR---WITW 552
POL_HV1H2 989 WT-------EYWQATWIPEWEFVNTPPLVKLWYQLEKEPIVGAETFYVDG 1031
.| ::.....:||.:.: |.:........|.|......||.||
POL_FOAMV 553 MTYLEDPRIQFHYDKTLPELKHI--PDVYTSSQSPVKHPSQYEGVFYTDG 600
POL_HV1H2 1032 AANRE---TKLGKAG----YVTNRGRQKV-----VTLTDTTNQKTELQAI 1069
:|.:. ||...|| :.|.:...:| :.|.:.|.|..|:.|:
POL_FOAMV 601 SAIKSPDPTKSNNAGMGIVHATYKPEYQVLNQWSIPLGNHTAQMAEIAAV 650
POL_HV1H2 1070 YL----ALQDSGLEVNIVTDSQYA 1089
.. ||:..| .|.::|||.|.
POL_FOAMV 651 EFACKKALKIPG-PVLVITDSFYV 673
Для поиска отличий в выравниваниях был использован Jalview: тут ссылка на проект для выравнивания water; тут ссылка на проект выравнивания BLAST. Некоторые отличия приведены в таблице 4, на картинках 5 и 6 приведены эти же отличия в Jalview для наглядности.

| № столбца в BLAST | № столбца в water | Пара в выравнивании BLAST | Пара в выравнивании water | Коэффициент для пары из BLAST | Коэффициент для пары из water |
|---|---|---|---|---|---|
| 75 | 75 | Pro682 - Gap | Pro682 - Pro226 | Штраф за гэп | 7 |
| 70 | 70 | Val677 - Ala230 | Vla677 - Gap | 0 | Штраф за гэп |
Литература
1. Interactions between Retroviruses and the Host Cell Genome. Valentina Poletti and Fulvio Mavilio. Ссылка на источник.
2. Мартица BLOSUM 62. Ссылка.