Секвенирование по Сэнгеру
1. Редактирование хроматограмм.
В данном практикуме были обработанны две хроматограммы, полученные в результате секвенирования прямой и обратной последовательностей по Сэнгеру.
Сначала редактирование хроматограмм проводилось с помощью веб-платформы Pearl. Программа позволяет загрузить одновременно два файла с последовательностями, переворачивает и комплементарит одну из них и выдает результат их выравнивания. К сожалению, Pearl распознавал прямую последовательность за обратную, а обратную за прямую соответственно. Качество выравнивания от этого, очевидно, не изменилось, но учтем, что в итоговом файле из Pearl будет лежать обратная комплементарная последовательность.
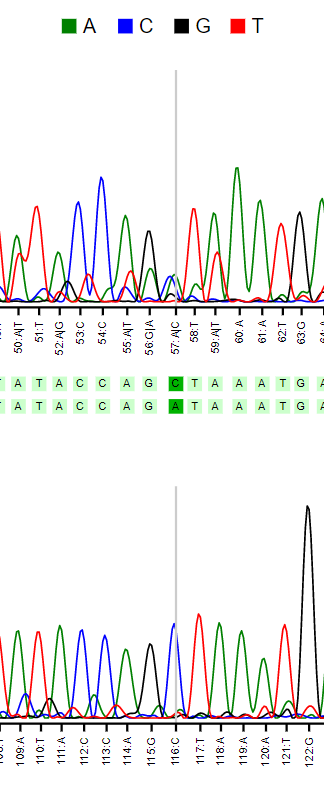
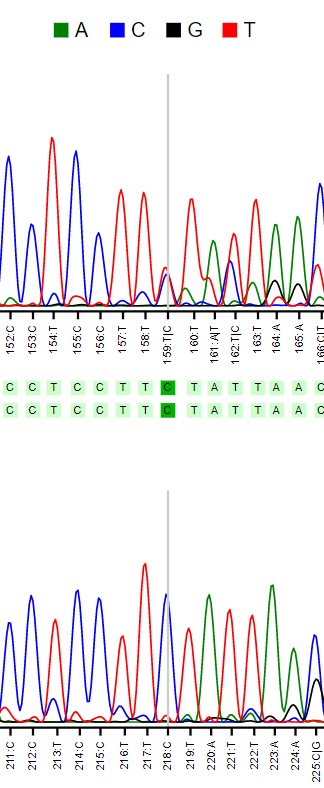
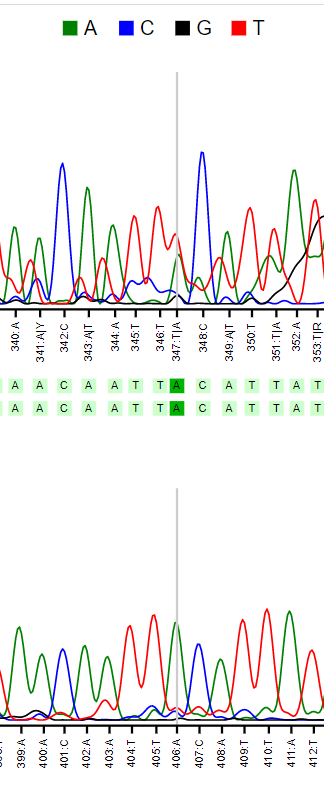
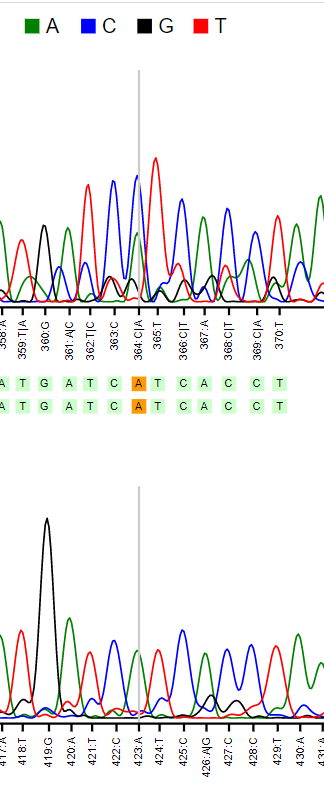
Pearl обнаружил пять проблемных нуклеотидов (они представлены на картинках 1-5). Каждый из них однозначно определялся за счет второй хроматограммы. Во всех случаях кроме одного (второго), решение моё и программы совпадали. Все спорные случаи возникали из-за сильных шумовых сигналов на какой-то из хроматограмм. Например, на картинке 5 видно, что проблемный нуклеотид находится на концевом участке верхней последовательности, где точность определения нукледтидов меньше за счет сложности разделения больших последовательностей в геле.
Файл с итоговой последовательностью из Pearl в fasta-формате находится тут. В данном файле проблемные нуклеотиды обозначены строчными буквами. Файл с выравниванием из Pearl прямой и обратной последовательностей находится тут.
Далее я проанализировала те же последовательности в программе Chromas. В отличие от Pearl, Chromas показывает какое количество нуклеотидов он отбрасывает с 3' и 5' концов (данные представлены в таблице 1).
В целом по по хроматограммам шума мало и он распределен равномерно. Но бывают выделяющиеся шумовые пики (например, такой пик показан на рисунке 3). Длины 3' и 5' нечитаемых концов у двух хроматограм примерно одинаковы.
| 14_F 5'-конец (автоматическое определение) | 1 - 148 нуклеотиды |
|---|---|
| 14_F 3'-конец (автоматическое определение) | 677 - 716 нуклеотиды |
| 14_F 5'-конец (моё определение) | 1 - 148 нуклеотиды |
| 14_F 3'-конец (моё определение) | 677 - 716 нуклеотиды |
| 14_R 5'-конец (автоматическое определение) | 1 - 138 нуклеотиды |
| 14_R 3'-конец (автоматическое определение) | 688 - 724 нуклеотиды |
| 14_R 5'-конец (моё определение) | 1 - 137 нуклеотиды |
| 14_R 3'-конец (моё определение) | 688 - 724 нуклеотиды |
После редактирования и удаления концов я выровняла последовательности с помощью water. Результаты выравнивания представлены ниже.
#=======================================
#
# Aligned_sequences: 2
# 1: L47
# 2: L47
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 406
# Identity: 376/406 (92.6%)
# Similarity: 376/406 (92.6%)
# Gaps: 0/406 ( 0.0%)
# Score: 1820.0
#
#
#=======================================
L47 144 CACGCTTTCGTGATAATTTTTTTTATAGTTATACCGTTAATAATTGGTGG 193
||||||||||.||||||||||||||||.||||||||||||.|||||||||
L47 1 CACGCTTTCGNGATAATTTTTTTTATANTTATACCGTTAANAATTGGTGG 50
L47 194 TNTTGGTAACTGAATAGTTCCTTTATTGATTGGGGCCCCAGATATAAGGT 243
| ||||||||||||||||||||||||||||||||||||||.|||||||||
L47 51 TTTTGGTAACTGAATAGTTCCTTTATTGATTGGGGCCCCANATATAAGGT 100
L47 244 TTCCTCGNATAAATAATATGAGTTTTTGATTATTACCACCTTCATTTATT 293
||||||. ||||.||||||||||||||||||||||||||||||.||||||
L47 101 TTCCTCNTATAANTAATATGAGTTTTTGATTATTACCACCTTCNTTTATT 150
L47 294 TTATTATTATGTTCTAGNTTAATAGAAGGAGGTGCTGGTACAGGNTGAAC 343
||||||||||||||||| |||.||.||||||||||||||||||| |||||
L47 151 TTATTATTATGTTCTAGGTTANTANAAGGAGGTGCTGGTACAGGTTGAAC 200
L47 344 TGTATATCCTCCTCTATCAGGTCCTGTAGCTCACGGGGGAAGTTCAGTAG 393
||||||||||||||||||||||||||||.|||||||||||||||||.|||
L47 201 TGTATATCCTCCTCTATCAGGTCCTGTANCTCACGGGGGAAGTTCANTAG 250
L47 394 ATTTAGCTATTTTTTCCCTTCATTTAGCTGGTATATCTTCTCTATTAGGA 443
||||||||||||||||||||||||||.||||||.|||||||||||.||||
L47 251 ATTTAGCTATTTTTTCCCTTCATTTANCTGGTANATCTTCTCTATNAGGA 300
L47 444 GCTATTANTTTNATTANTACTATTTTTAATATACGGTCTAGAGCTATAAC 493
||||||| ||| |||| |||||||||||||||||||||||.|||||||||
L47 301 GCTATTAATTTTATTACTACTATTTTTAATATACGGTCTANAGCTATAAC 350
L47 494 AATAGAACGATTGAGTTTGTTCGTATGATCCATTTTAGTGACAGTATTCT 543
||||.|...|||||.|||||||.||||||||||||||.||||||||||||
L47 351 AATANANNNATTGANTTTGTTCNTATGATCCATTTTANTGACAGTATTCT 400
L47 544 TACTNT 549
|||| |
L47 401 TACTAT 406
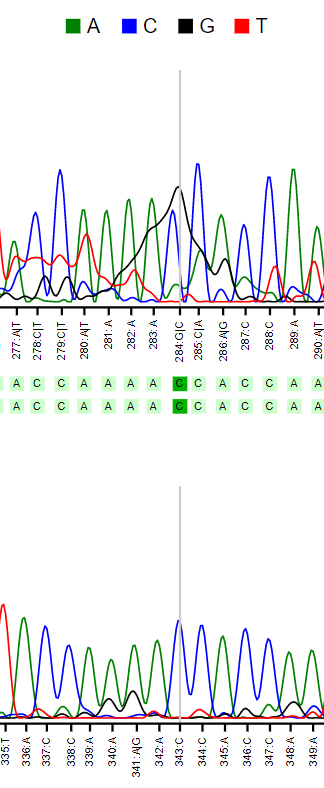
Далее в Chromas я проанализировала позиции, которые хотя бы в одной из хроматограмм не определись. В данном случае из выравнивания видно, что все проблемные нуклеотиды хорошо распознаются хотя бы в одной из последовательностей. На рисунках 7, 8 представлены примеры таких проблемных нуклеотидов.
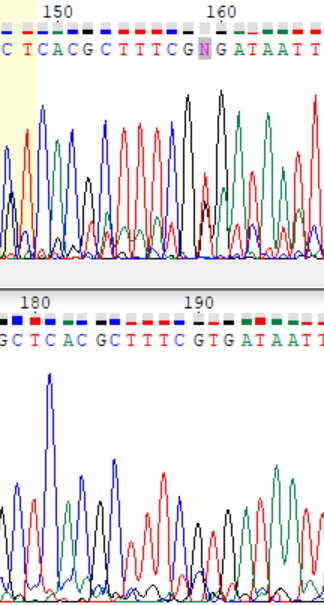
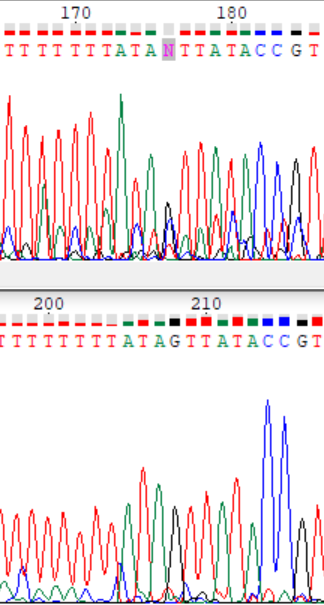
Итоговый файл с последовательностью из Chromas в можно найти тут. Тут проблемные нуклеотиды так же обозначены строчными буквами.
Далее я выровняла последовательность из Pearl и последовательность из Chromas с помощью water. Перед этим перевернула и откомплементарила последовательность из Pearl (forward последовательность лежит тут). Само выравнивание можно найти тут.
Итог: консенсусная последовательность с проблемными нуклеотидами в нижнем регистре, консенсусная последовательность в fasta-формате.
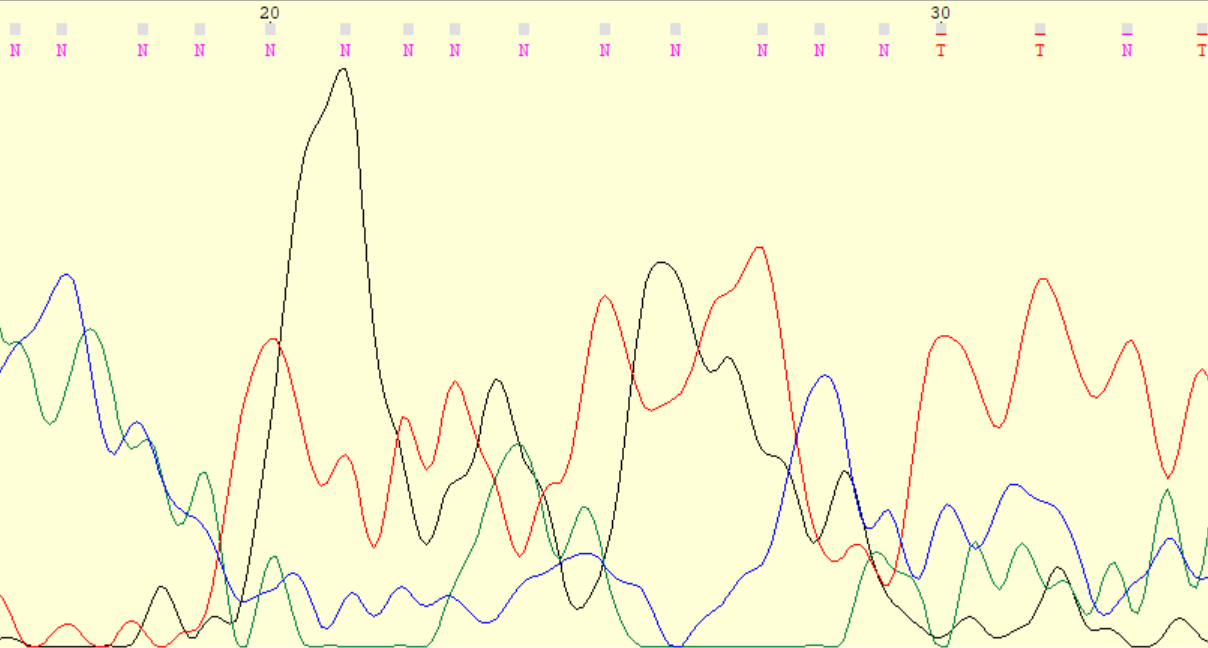
2. Пример нечитаемого фрагмента хроматограммы.
В качестве нечитаемого участка возьмем начало 5'-конца изучаемой выше хроматограммы 14_F. Видно, что сигналы накладываются друг на друга. Пики растянуты на нескольно нуклеотидов. Из-за этого невозможно понять какой из сигналов преобладает и соответственно нельзя определить нуклеотид.