Банки нуклеотидных последовательностей
Задание 1.
В первом задании была выбрана нуклеотидная последовательность с идентификатором KF986566. Она была найдена в базе данных GeneBank с помощью запроса: "WSBS" "White Sea".
Из записи можно получить следующую информацию:
- Идентификатор записи: KF986566
- Это линейная последовательность ДНК длиной 268 пар оснований
- Дата депонирования в банк: 20 февраля 2014 года
- Авторы записи: Neretina,T., Stupnikova,A., Kolbasova,G., Konovalova,O., Schepetov,D. and Mugue,N.
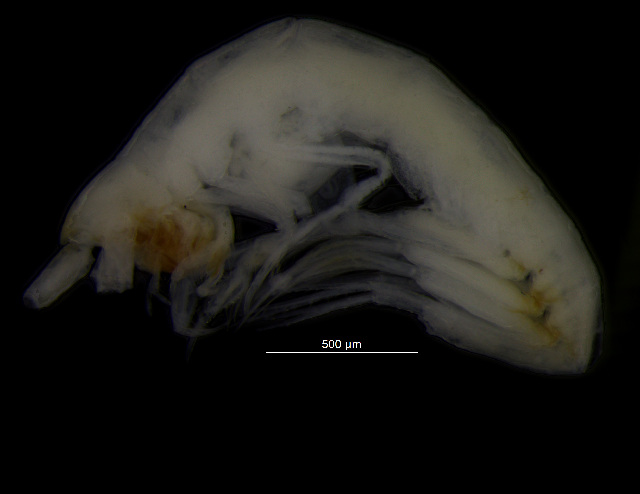
- Организм, из которого был получен образец: Dyopedos porrectus. Это мелкий рачок с тонким панцирем, фотография представлена на рис. 1.
Понять, что этот образец был получен на ББС (White Sea Biologocal Station - WSBS) можно из следующих строк:
FEATURES Location/Qualifiers
source 1..268
/specimen_voucher="WS1396 WSBS Malyi Krestovyi Island"
/lat_lon="66.52 N 33.19 E"
/collection_date="21-Aug-2011"
/collected_by="N. Neretin"
Также видно, что образец собрал N. Neretin 21 августа 2011 года в месте с координатами 66.52 N 33.19 E. Ниже на карте я отметила место сбора и расположение биологической станции.
Интересно, что в том же разделе FEATURES указали последовательности и названия праймеров, используемых для ПЦРа.
FEATURES Location/Qualifiers
source 1..268
/PCR_primers="fwd_name: h3af, fwd_seq:
atggctcgtaccaagcagacvgc, rev_name: h3ar, rev_seq:
atatccttrggcatratrgtgac"
Последовательность в fasta-формате можно найти тут.