Nucleotide BLAST
1. Определение функции и таксономии нуклеотидной последовательности.
Цель задания - научиться по нуклеотидной последовательности делать какие-то выводы о ее таксономической принадлежности и предсказывать функцию возможно кодируемого белка.
В данном разделе практикума использовалась консенсусная последовательность, полученная в результате обработки двух хроматограмм неизвестной нуклеотидной последовательности (анализ хроматограмм подробнее описан в практикуме 6).
Чтобы найти сколько-нибудь похожие последовательности использовался алгоритм blastn. Измененные параметры поиска представлены ниже (остальные использовались по умолчанию):
- Database: Nucleotide collection (nr/nt)
- Exclude: Uncultured/environmental sample sequences
- Optimize for: blastn
- Word size: 7
Выдача бласта лежит тут.
Пять лучших находок относятся к митохондриальному гену цитохром с-оксидазы Ⅰ (COⅠ) одного вида - Ancula gibbosa. Все эти выравнивания имеют 100% покрытие, E-value каждого очень мал (E-value первых трех находок вообще приравнивается к 0.0). Отсюда можно с большой точностью утверждать, что изначальная последовательность также относится к данному виду и кодирует такой же белок.
Ancula gibbosa - это маленький морской брюхоногий моллюск из семейства Goniodorididae (представлен на картинке 1).

2. Поиск генов белков в неаннотированной нуклеотидной последовательности
В данном задании нужно было исследовать неаннотированную нуклеотидную последовательность на наличие генов и предсказать функцию кодируемого белка.
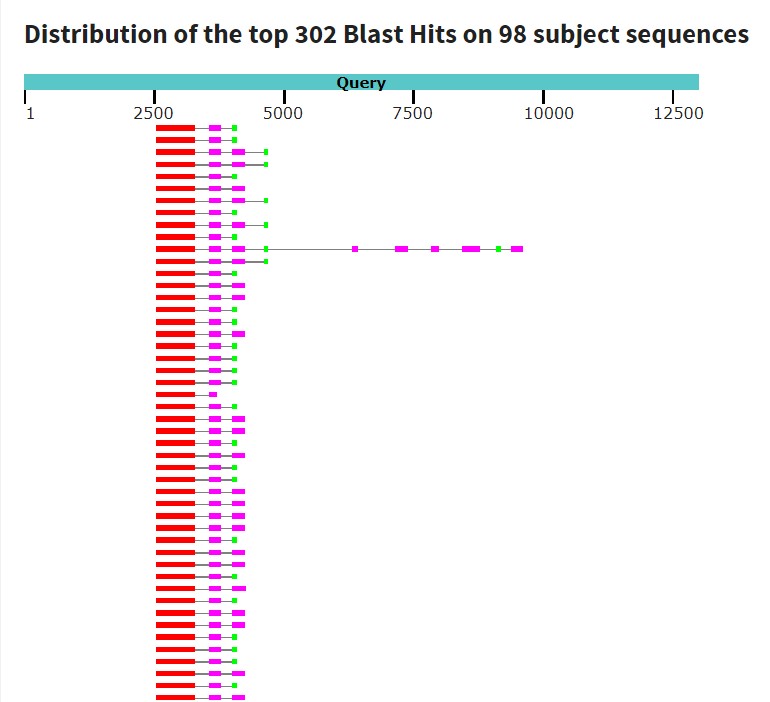
В качестве последовательности был взят контиг из сборки Cucurbita pepo subsp. pepo (AC сборки из RefSeq: GCF_002806865.1), которая исследовалась в предыдущем практикуме. Для того чтобы понять, кодирует ли эта последовательность какие-нибудь белки, и если кодирует, то какие, воспользуемся алгоритмом blastx. Из параметров была изменена только база данных - из поиска были исключены белки, принадлежащие самому виду Cucurbita pepo (taxid:3663). Выдачу бласта можно посмотреть по ссылке. Визуальное представление нескольких первых выравниваний представлено на картинке 2.
Из картинки видно, что бласт строит выравнивания в одном положении контига. При этом в выравнивание разрывается на несколько консервативных достаточно больших кусков. Характеристики лучших выравниваний позволяют предположить, что в данной последовательности кодируется белок под названием O-fucosyltransferase 13-like.
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
Для исследования карты локального сходства были выбраны полные геномы двух родственных бактерий - Mycobacterium tuberculosis и Mycobacterium kansasii. Карта была получена с помощью алгоритма megablast (представлена на рисунке 3). Параметры были оставлены по умолчанию.
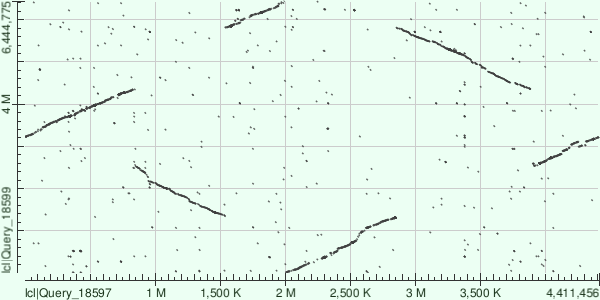
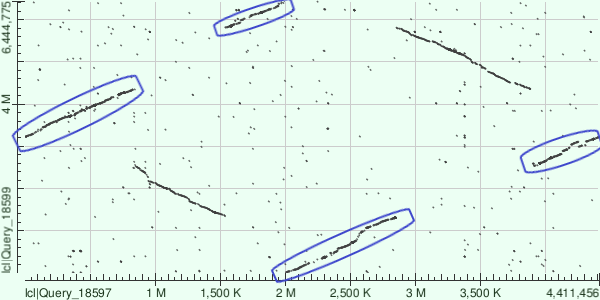
В целом из карты видно, что бактерии являются родственными - есть протяженные гомологичные участки в разных частях хромосомы. Также на карте видны два участка крупных геномных перестроек - инверсий (см. на рис. 4.). Геном бактерий кольцевой, поэтому из-за особенностей нумерации нуклеотидов видны разрывы в карте.