2. Реконструкция и визуализация.
(a) Множественное выавнивание было импортированно в MEGA методом Analyze. Далее было построено дерево с помощью алгоритма наибольшего правдоподобия. Полученное дерево в Newick-формате представлено ниже.
(((((((CLPX_ECOLI,CLPX_SERP5),CLPX_SHEDO),CLPX_HAEIN),(CLPX_BARHE,(CLPX_ROSDO,CLPX_PARDP))),CLPX_POLAQ),(((HSLU_PARDP,HSLU_ROSDO),HSLU_BARHE),(HSLU_SHEDO,(HSLU_HAEIN,(HSLU_SERP5,HSLU_ECOLI))))),(A1B8N4_PARDP,(A8GCD8_SERP5,CLPA_ECOLI)),((RUVB_ROSDO,RUVB_BARHE),((((A1AZV8_PARDP,Q167Z2_ROSDO),A1BBJ2_PARDP),A0A0H3LXZ4_BARHE),(A4SXL5_POLAQ,(FTSH_HAEIN,(Q12QI8_SHEDO,(FTSH_ECOLI,A8G901_SERP5)))))));
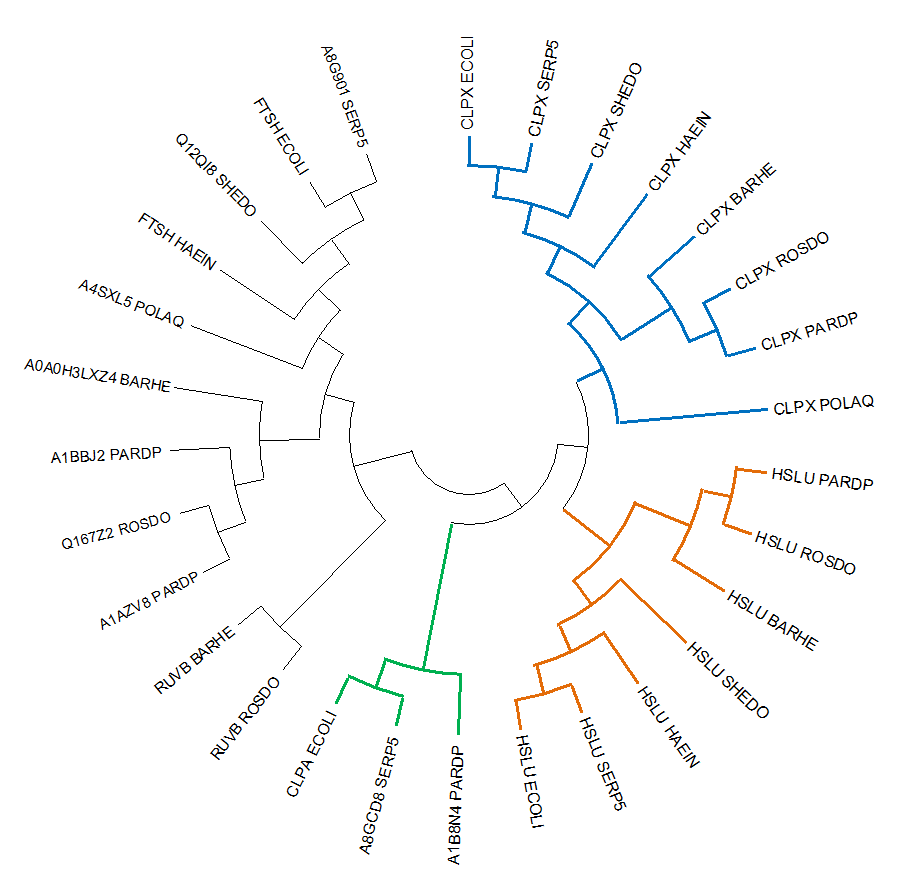
(b) Пары ортологов: CLPX_ROSDO и CLPX_PARDP; HSLU_PARDP и HSLU_ROSDO; RUVB_BARHE и
RUVB_ROSDO.
Пары паралогов: A1BBJ2_PARDP и A1AZV8_PARDP; RUVB_BARHE и A0A0H3LXZ4_BARHE; A1B8N4_PARDP
и A1AZV8_PARDP.
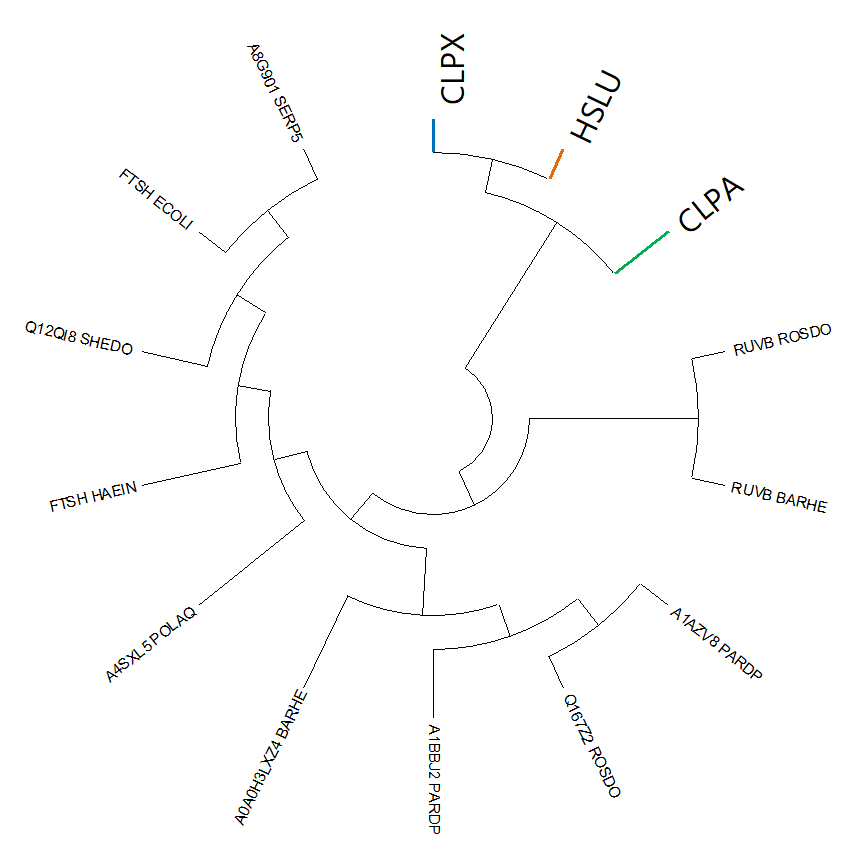
Внутри оранжевой (HSLU) и зеленой (CLPA) групп филогения белков совпадает с филогенией бактерий, полученных в первом практикуме. В синей группе (CLPX) филогения белков отличается от филогении бактерий.