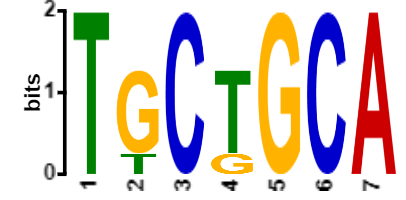2. Поиск мотива сайта разрывной транскрипции sgRNA в геноме одного из коронавирусов (Coronaviridae) с помощью программы MEME
sgmRNA - субгеномная матричная РНК коронавирусов (Coronaviridae) и некоторых других нидовирусов (Nidovirales). Она образуется путем объединения гена позднего белка с лидерной последовательностью при образовании -РНК, с последующей репликацией полученной -sgRNA в +sgRNA. Сигналы разрывной транскрипции направляют перескок вирусной РНК-зависимой РНК-полимеразы (RdRP) при синтезе -RNA.
В лидерной последовательности находится сигнал называемый TRS-L (TRS от Transcription-Regulating Sequence). Перед кодирующей последовательностью каждого позднего гена находятся последовательности TRS-B. Все сигналы TRS-B и TRS-L включают одинаковую последовательность из шести нуклеотидов. Последовательность называется CS (от Core Sequence). В статье Sonia Zúñiga 2004 года [1] она приводится в явном виде: 5′-CTAAAC-3′. Сигналы TRS шире CS на несколько (неизвестно сколько) нуклеотидов с 5' и 3' стороны.
Целью данного практикума заключалась в поиске данных сигналов с помощью программы MEME.
Подготовка входных последовательностей
Для поиска мотивов был выбран геном Severe acute respiratory syndrome coronavirus 2. Файл с полным геномом вируса можно найти по ссылке. Сначала был составлен файл с координатами upstream областей перед геном полипротеина (orf1ab) и перед каждым поздним геном. Далее с помощью утилит из EMBOSS был получен отдельный файл с upstream областями - upstream.fasta. Историю команд можно найти тут.
Нахождение мотива CS с помощью MEME
Итоговый файл анализировался с помощью программы MEME. Параметры поиска мотивов указаны ниже:
- Select the site distribution - 0 или 1 мотив в последовательности
- Select the number of motifs - 3
- How wide can motifs be? - минимум 2, максимум 600
- Can motif sites be on both strands? - искать только в одной цепи
- What should be used as the background model? - 0-order model of sequences
Выдачу программы в текстовом виде можно найти по ссылке.
Расположение найденных мотивов представлено ниже.
Наиболее распространенный мотив (красный цвет на рис. 1.) найден в 7 последовательностях из 11. Но данная
последовательность
не обружена перед полипротеином. При этом в мотиве найдена последовательность CS. На рисунке
2 представлен данный мотив с выделенным CS участком.
Еще один
достаточно консервативный участок (зеленый цвет на рис. 1.) найден в пяти последовательностях, включая
upstream область orf1ab. Он представлен на рисунке 3.