2. Поиск сайтов разрывной транскрипции sgmRNA в геноме коронавируса, используя PWM из задания 6.2
Из прошлого практикума была взята находка с наименьшим p-value (7.6e-006). Она была передана программе FIMO. Поиск мотивов производился в том же коронавирусе Severe acute respiratory syndrome coronavirus 2. Выдача программы представлена на рисунке 1.
Сравнивая данные о расположении полипротеина и поздних генов с полученными мотивами, получаем следующее расположение: 1-ый мотив находится между 8 и 9 поздним геном, 2-ая между полипротеином и 1 поздним геном, 3-я между 1 и 2, 4-ая между 7 и 8, 5-ая между 3 и 4, 6-ая между 9 и 10, 7-ая между 6 и 7, а последние пять находок лежал внутри гена полипротеина. Первые восемь находок вероятно являются нужными TRS-L(вторая находка) и TRS-B (остальные) мотивами. Таблица с координатами гена полипротеина и поздних генов лежит тут.
Далее нужно было построить LOGO для последовательностей Козак поздних генов выбранного коронавируса. Для этого был создан файл с последовательностями соответствующих последовательности Козак областей (координаты -6...+4 вокруг нужных генов). Файл можно найти тут. Данный файл был загружен в сервис для LOGO. Выдача представлена на рисунке 2.
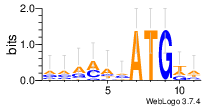
На рисунке 3 изображен консенсус последовательноси Козак для человеческих генов. Видно, что выдача для коронавируса сильно отличается от человеческой.
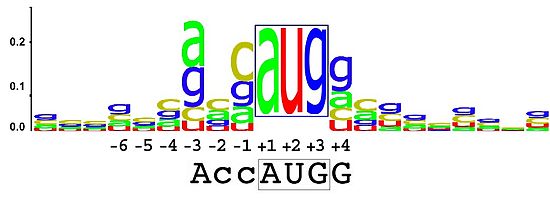
Для определения специфичности сигнала из первой части практикума, был произведен его поиск с помощью программы FIMO в геномах двух близкородственных коронавирусов - Bat coronavirus 1A и Night heron coronavirus HKU19. Выдача представлена на рисунках 4 и 5.
Видно, что для Bat coronavirus 1A находится достаточно много сходных нашему мотивов. А для Night heron coronavirus HKU19 находок мало. Вероятно, это говорит о специфичности нашего сигнала.