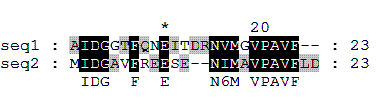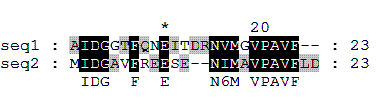Программа BLASTP
Страница запроса BLASTP
1.
| |
Поиск по БД Swiss-Prot |
Поиск по БД PDB |
Поиск по БД "nr" |
| 1. Лучшая находка |
| Идентификатор БД |
P35340 |
1HYU |
NP_286333.1 |
| E-value |
0.0 |
0.0 |
0.0 |
| Вес (в битах) |
1068 |
1011 |
1069 |
| % идентичности |
100% |
95% |
|
| Найдены ли другие белки с теми же значениями E-value и веса в битах? |
Белков с таким же весом не обнаружено, зато есть ещё 5 с нулевым E-value. ID самого близкого по весу (1011) - P19480 (AHPF_Salty) |
2 белка с нулевым E-value, из которых вес второго - 630 (1FL2 - AHPF_Ecoli) |
148 белков с нулевым E-value, из них два с весом 1068. Пример - YP_001461770.1 (AHPF_Ecoli) |
| 2. Сколько хороших кандидатов в гомологи найдено?
(число находок в списке описаний, Descriptions,
с E-value < 1E-10)
| 163 |
18 |
4999 |
| 2.
"Худшая" находка (последняя в выдаче с E-value < 1)
|
| Номер находки в списке описаний (Descriptions) |
433 |
91 |
9386 |
| Идентификатор БД |
A5E8G8 |
1ZMC (P09622) |
YP_151868.1 (Q5PF36) |
| E-value |
0.98 |
0.64 |
0.99 |
| Вес (в битах) |
35 |
31.6 |
39.3 |
| % идентичности |
50% |
42% |
25% |
| % сходства |
64% |
60% |
44% |
| Длина выравнивания |
34 |
40 |
160 |
| Координаты выравнивания (номера первых и последних а.о.) |
212-245 AHPF_Ecoli, 7-40 MNMG_Brasb |
214-251 AHPF_Ecoli, 8-47 DLDH_Human |
348-498 AHPF_Ecoli, 134-288 NORW_Salpa |
| % гэпов
| 0% |
5% |
8% |
Мы видим, что, чем объёмней банк (в порядке убывания числа последовательностей - "nr">SwissProt>PDB), тем больше будет номер "худшего" выравнивания,
а E-value его ближе к, собственно, единице. Относительно низкое E-value для PDB-банка объясняется тем, что результатов в принципе немного и разброс величин E-value
для них велик. Так что что следующий, не вошедший в список результат просто больше единицы.
2.Гомолог моего белка у человека: Q96NN9 (AIFM3_Human)
E-value 3e-06
Вес 50.4
Идентичность 23%
Сходство 42%
Длина выравнивания 297
Координаты 214-493 AHPF_Ecoli, 196_472 AIFM3_Human
Гэпы 12%
3.Результаты поиска белка в Swiss-Prot по фрагменту последовательности
| |
Поиск по фрагменту |
Поиск по полной
последовательности |
| АС лучшей находки |
Q5HRY2 (AHPF_STAEQ) |
Q5HRY2 (AHPF_STAEQ) |
| E-value |
3e-15 |
0.0 |
| Вес (в битах) |
78.7 |
1035 |
Найдены ли другие белки с теми же значениями E-value и веса в битах?
|
Да, белок AHPF_STAES, тот же самый, но из другого штамма Staphylococcus epidermidis |
7 белков с нулевым E-value. Вес выравнивания с AHPF_Staes - 1034 |
Разница между значениями весов и E-value для выравниваний участков и полных последовательностей объясняется разницей длин.
Маленький участок имеет меньшую длину (что уменьшает E), но и меньший вес (что значитительно увеличивает E, т.к. вес стоит в показателе степени e со знаком "-")
Соответственно, выравнивание полных последовательностей со значительно большим весом будет иметь гораздо меньшее E-value.
Выравнивание AHPF_Staeq (query 1) с моим белком (sbjct 1)
Query 1 MLNADLKQQLQQLLELMEGDVEFVASLGSDDKSNELKELLNEMAEMSAHITITE-KSLK- 58
ML+ ++K QL+ LE + VE +A+L KS E+KELL E+AE+S +T E SL
Sbjct 1 MLDTNMKTQLKAYLEKLTKPVELIATLDDSAKSAEIKELLAEIAELSDKVTFKEDNSLPV 60
Query 59 RTPSFSVNRPGEETGITFAGIPLGHEFNSLVLAILQVSGRAPKEKQSIIDQIKGLEGPFH 118
R PSF + PG G FAG PLGHEF SLVLA+L G KE QS+++QI+ ++G F
Sbjct 61 RKPSFLITNPGSNQGPRFAGSPLGHEFTSLVLALLWTGGHPSKEAQSLLEQIRHIDGDFE 120
Query 119 FETFVSLTCQKCPDVVQALNLMSVINPNITHTMIDGAVFREE--SENIMAVPAVFLDGQE 176
FET+ SL+C CPDVVQALNLMSV+NP I HT IDG F+ E N+M VPAVF++G+E
Sbjct 121 FETYYSLSCHNCPDVVQALNLMSVLNPRIKHTAIDGGTFQNEITDRNVMGVPAVFVNGKE 180
Query 177 FGNGRMTVQDILTKL---GSTQDASEFNDKDPYDVLIVGGGPASGSAAIYTARKGLRTGI 233
FG GRMT+ +I+ K+ + A E N +D YDVLIVG GPA +AAIY+ARKG+RTG+
Sbjct 181 FGQGRMTLTEIVAKIDTGAEKRAAEELNKRDAYDVLIVGSGPAGAAAAIYSARKGIRTGL 240
Query 234 VADRIGGQVNDTAGIENFITVKETTGSEFSSNLAEHIAQYDIDTMTGIRATNIEKTDSAI 293
+ +R GGQ+ DT IEN+I+V +T G + + L H+ +YD+D + A+ + +A+
Sbjct 241 MGERFGGQILDTVDIENYISVPKTEGQKLAGALKVHVDEYDVDVIDSQSASKL--IPAAV 298
Query 294 -----RVTLENDAVLESKTVIISTGASWRKLNIPGEDRLINKGVAFCPHCDGPLFENKDV 348
++ + AVL+++++I++TGA WR +N+PGED+ KGV +CPHCDGPLF+ K V
Sbjct 299 EGGLHQIETASGAVLKARSIIVATGAKWRNMNVPGEDQYRTKGVTYCPHCDGPLFKGKRV 358
Query 349 AVIGGGNSGVEAAIDLAGIVKHVTLFEYASELKADSVLQERLRSLPNVDIKTSAKTTEVI 408
AVIGGGNSGVEAAIDLAGIV+HVTL E+A E+KAD VLQ++LRSL NVDI +A+TTEV
Sbjct 359 AVIGGGNSGVEAAIDLAGIVEHVTLLEFAPEMKADQVLQDKLRSLKNVDIILNAQTTEVK 418
Query 409 GD-DYVTGISYEDMTTGESQVVNLDGIFVQIGLVPNTSWLQNAVELNERGEVMINRDNAT 467
GD V G+ Y D +G+ + L GIFVQIGL+PNT+WL+ AVE N GE++I+ T
Sbjct 419 GDGSKVVGLEYRDRVSGDIHNIELAGIFVQIGLLPNTNWLEGAVERNRMGEIIIDAKCET 478
Query 468 NVPGIFAAGDVTDQKNKQIIISMGAGANAALNAFDYIIR 506
NV G+FAAGD T KQIII+ G GA A+L+AFDY+IR
Sbjct 479 NVKGVFAAGDCTTVPYKQIIIATGEGAKASLSAFDYLIR 517
Выравнивание, сделанное мной прежде в домашней работе (153-175 AHPF_Ecoli, 151-173 AHPF_Staeq)
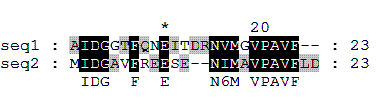
Видим, что выравнивания различаются положением гэпа.
А вот в случае с выравниванием участка наблюдаем следующее
Query 2 IDGAVFREE-SE-NIMAVPAVF 21
IDG F+ E ++ N+M VPAVF
Sbjct 154 IDGGTFQNEITDRNVMGVPAVF 175
Здесь сразу два гэпа. С другой стороны, замены серина на треонин и глутаминовой кислоты на аспарагиновую имеют положительный вес,
а вот серина на изолейцин и глутаминовой к-ты на треонин - отрицательный
(замены S и E на D и R имеют нулевой вес, так что разница в положении, указанная выше, имеет смысл).
Таким образом, видим, что вес выравнивания, сделанного мной, - наименьший из трёх
(причина в том, что вес любой близкородственной замены мы принимали равным за единицу - разумеется, BLASTP использовал более адекватные числа).
3.Выравнивания AHPF_Ecoli и TRXB_Bacsu (штраф за открытие гэпа - 11, за протяжённость - 1)
- BLASTP (207-515 AHPF_Ecoli, 1-305 TRXB_Bacsu, вес - 466)
Query 207 LNKRDAYDVLIVGSGPAGAAAAIYSARKGIRTGLMGERF--GGQILDTVDIENYISVPKT 264
+++ YDV+I+G+GPAG AA+Y++R + T LM ER GGQ+ +T D+ENY
Sbjct 1 MSEEKIYDVIIIGAGPAGMTAAVYTSRANLST-LMIERGIPGGQMANTEDVENYPGFESI 59
Query 265 EGQKLAGALKVHVDEYDVDVIDSQSASKLIPAAVEGGLHQIETASGAVLKARSIIVATGA 324
G +L+ + H ++ ++ A I ++G +++ A KAR++I+A GA
Sbjct 60 LGPELSNKMFEHAKKFG-----AEYAYGDIKEVIDGKEYKVVKAGSKEYKARAVIIAAGA 114
Query 325 KWRNMNVPGEDQYRTKGVTYCPHCDGPLFKGKRVAVIGGGNSGVEAAIDLAGIVEHVTLL 384
+++ + VPGE + +GV+YC CDG FKGK + V+GGG+S VE + L VT++
Sbjct 115 EYKKIGVPGEKELGGRGVSYCAVCDGAFFKGKELVVVGGGDSAVEEGVYLTRFASKVTIV 174
Query 385 EFAPEMKADQVLQDKLRSLKNVDIILNAQTTEVKGDGSKVVGLEYRDRVSGDIHNIELAG 444
+++A +LQ + + VD + N E+ + KV + D V+G+ + G
Sbjct 175 HRRDKLRAQSILQARAFDNEKVDFLWNKTVKEIHEENGKVGNVTLVDTVTGEESEFKTDG 234
Query 445 IFVQIGLLPNTNWLEGAVERNRMGEIIIDAKCETNVKGVFAAGDCTTVPYKQIIIATGEG 504
+F+ IG+LP + E N G I + + ET V+G+FAAGD +QI+ ATG+G
Sbjct 235 VFIYIGMLPLSKPFENLGITNEEGYIETNDRMETKVEGIFAAGDIREKSLRQIVTATGDG 294
Query 505 AKASLSAFDYL 515
+ A+ S Y+
Sbjct 295 SIAAQSVQHYV 305
- глобальное (1-521 AHPF_Ecoli, 1-316 TRXB_Bacsu, вес - 248)
10 20 30 40 50
AHPF_E MLDTNMKTQLKAYLEKLTKPVELIATLDDSAKSAEIKELLAEIAELSDKV
: . ::.
TRXB_B MSE-----------EKI---------------------------------
60 70 80 90 100
AHPF_E TFKEDNSLPVRKPSFLITNPGSNQGPRFAGSPLGHEFTSLVLALLWTGGH
TRXB_B --------------------------------------------------
110 120 130 140 150
AHPF_E PSKEAQSLLEQIRHIDGDFEFETYYSLSCHNCPDVVQALNLMSVLNPRIK
TRXB_B --------------------------------------------------
160 170 180 190 200
AHPF_E HTAIDGGTFQNEITDRNVMGVPAVFVNGKEFGQGRMTLTEIVAKIDTGAE
TRXB_B --------------------------------------------------
210 220 230 240
AHPF_E KRAAEELNKRDAYDVLIVGSGPAGAAAAIYSARKGIRTGLMGERF--GGQ
:::.:.:.:::: ::.:..: . : :: :: :::
TRXB_B ------------YDVIIIGAGPAGMTAAVYTSRANLST-LMIERGIPGGQ
10 20 30 40
250 260 270 280 290
AHPF_E ILDTVDIENYISVPKTEGQKLAGALKVHVDEYDVDVIDSQSASKLIPAAV
. .: :.::: : .:. . : .. . : : .
TRXB_B MANTEDVENYPGFESILGPELSNKMFEHAKKFGAEY-----AYGDIKEVI
50 60 70 80
300 310 320 330 340
AHPF_E EGGLHQIETASGAVLKARSIIVATGAKWRNMNVPGEDQYRTKGVTYCPHC
.: ... : :::..:.: ::... . :::: . .::.:: :
TRXB_B DGKEYKVVKAGSKEYKARAVIIAAGAEYKKIGVPGEKELGGRGVSYCAVC
90 100 110 120 130
350 360 370 380 390
AHPF_E DGPLFKGKRVAVIGGGNSGVEAAIDLAGIVEHVTLLEFAPEMKADQVLQD
:: :::: . :.:::.: :: . : ::.. ...: .::
TRXB_B DGAFFKGKELVVVGGGDSAVEEGVYLTRFASKVTIVHRRDKLRAQSILQA
140 150 160 170 180
400 410 420 430 440
AHPF_E KLRSLKNVDIILNAQTTEVKGDGSKVVGLEYRDRVSGDIHNIELAGIFVQ
. . :: . : :. . :: . : :.:. . :.:.
TRXB_B RAFDNEKVDFLWNKTVKEIHEENGKVGNVTLVDTVTGEESEFKTDGVFIY
190 200 210 220 230
450 460 470 480 490
AHPF_E IGLLPNTNWLEGAVERNRMGEIIIDAKCETNVKGVFAAGDCTTVPYKQII
::.:: . : : : : . . :: :.:.::::: .::.
TRXB_B IGMLPLSKPFENLGITNEEGYIETNDRMETKVEGIFAAGDIREKSLRQIV
240 250 260 270 280
500 510 520
AHPF_E IATGEGAKASLSAFDYLIRTKTA-----
:::.:. :. : :. .
TRXB_B TATGDGSIAAQSVQHYVEELQETLKTLK
290 300 310
- локальное (207-515 AHPF_Ecoli, 1-305 TRXB_Bacёsu, вес - 478)
210 220 230 240 250
AHPF_E LNKRDAYDVLIVGSGPAGAAAAIYSARKGIRTGLMGERF--GGQILDTVD
... :::.:.:.:::: ::.:..: . : :: :: :::. .: :
TRXB_B MSEEKIYDVIIIGAGPAGMTAAVYTSRANLST-LMIERGIPGGQMANTED
10 20 30 40
260 270 280 290 300
AHPF_E IENYISVPKTEGQKLAGALKVHVDEYDVDVIDSQSASKLIPAAVEGGLHQ
.::: : .:. . : .. . : : ..: ..
TRXB_B VENYPGFESILGPELSNKMFEHAKKFGAEY-----AYGDIKEVIDGKEYK
50 60 70 80 90
310 320 330 340 350
AHPF_E IETASGAVLKARSIIVATGAKWRNMNVPGEDQYRTKGVTYCPHCDGPLFK
. : :::..:.: ::... . :::: . .::.:: ::: ::
TRXB_B VVKAGSKEYKARAVIIAAGAEYKKIGVPGEKELGGRGVSYCAVCDGAFFK
100 110 120 130 140
360 370 380 390 400
AHPF_E GKRVAVIGGGNSGVEAAIDLAGIVEHVTLLEFAPEMKADQVLQDKLRSLK
:: . :.:::.: :: . : ::.. ...: .:: . .
TRXB_B GKELVVVGGGDSAVEEGVYLTRFASKVTIVHRRDKLRAQSILQARAFDNE
150 160 170 180 190
410 420 430 440 450
AHPF_E NVDIILNAQTTEVKGDGSKVVGLEYRDRVSGDIHNIELAGIFVQIGLLPN
:: . : :. . :: . : :.:. . :.:. ::.::
TRXB_B KVDFLWNKTVKEIHEENGKVGNVTLVDTVTGEESEFKTDGVFIYIGMLPL
200 210 220 230 240
460 470 480 490 500
AHPF_E TNWLEGAVERNRMGEIIIDAKCETNVKGVFAAGDCTTVPYKQIIIATGEG
. : : : : . . :: :.:.::::: .::. :::.:
TRXB_B SKPFENLGITNEEGYIETNDRMETKVEGIFAAGDIREKSLRQIVTATGDG
250 260 270 280 290
510
AHPF_E AKASLSAFDYL
. :. : :.
TRXB_B SIAAQSVQHYV
300
Никаких разительных отличий не наблюдается: программой BLASTP рассмотрен тот же участок, что и WATER,
отличается лишь положение самого большого гэпа - 282-286 AHPF_Ecoli (в двух последних выравниваниях его позиции - 285-289)
В выравнивании BLASTP аланин сопоставлен не серину, а валину, глутаминовая к-та не аспарагиновой, а глутамину, лизин не серину, а валину.
Если рассматривать BLOSUM62, то замены эти по весу одинаковы. Думаю, их вес приблизительно одинаков и для программы BLASTP.
В данном случае, скорее всего, это различие - случайность.