В этом задании нужно было построить филогенетическое дерево тех же бактерий, что и в предыдущем практикуме, но на этот раз с использованием последовательности 16S rRNA.
Для этого:
я получила последовательности 16S rRNA хромосом бактерий.
эти последовательности выравнивнила с помощью программы Jalview (метод выравнивания muscle with defaults).
построила дерево с помощью программы MEGA6 (методом Neigbour-Joining).
фаста-файл с последовательностями
дерево

Филогенетическое дерево гена 16s rRNA
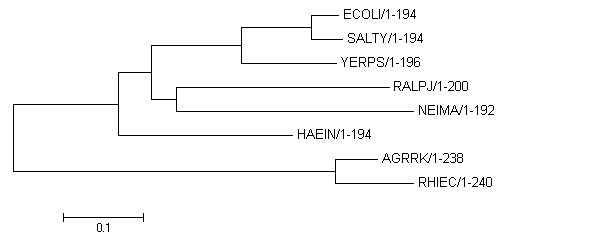
Филогенетическое дерево, построенное по белковой последовательности
При сравнении двух рисунков видно, что деревья не отличаются расположением ветвей, а только их длинами.
Точность реконструкции улучшилась. Возможно, это связано с тем, что нуклеотидные последовательности длиннее белковых.
Задание 2. Построение и анализ дерева, содержащего паралоги.
Выбранные виды бактерий и их мнемонические названия.
| Название | Мнемоника |
|---|---|
| Rhizobium etli | RHIEC |
| Rhodobacter sphaeroides | RHOS4 |
| Neisseria meningitidis | NEIMA |
| Pseudomonas aeruginosa | PSEAE |
| Erwinia tasmaniensis | ERWT9 |
| Yersinia pestis | YERPE |
| Vibrio fischeri | VIBFM |
| Proteus mirabilis | PROMH |
Для поиска гомологов белка CLPX_ECOLI была создана база данных из протеомов 8 исследуемых бактерий. (на основе файла needed_bacter.fasta). (Файлы из директории P:\y14\term4\Proteomes, содержащие полные протеомы нужных мне бактерий, скачанные из базы UniProt, были сложены в один файл)
Затем я произвела поиск гомологов программой blastp с входной последовательностью белка CLPX_ECOLI по базе данных - файлу с нужными протеомами. Порог по E-value был взят 0.001.
Использованные команды:
Выходной файл: results.txt
Нашлось 27 находок для такого E-value.
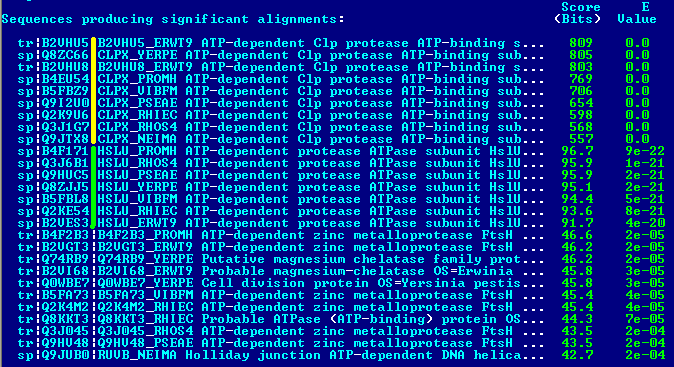
Среди найденных последовательностей легко можно выделить 3 группы: собственно белки CLPX (с более высоким E-value), HSLU и третья группа из разных белком с худшим E-value.
Поэтому дальше исследовались только гомологи из первых двух групп (жёлтой и зелёной).fasta файл 16 гомологов
Последовательности этих гомологов были открыты в JalView и выровнены программой Muscle.
Далее было построено дерево с помощью алгоритма Neighbor-Joining.
Далее нужно было найти на этом дереве ортологи и паралоги.
Паралоги - это гомологи из одного организма (произошли благодаря дупликации генов), а ортологи - гомологи из разных организмов, и разделение их общего предка произошло из-за разделения путей эволюции белков в результате видообразования.
Примеры паралогов выделены жёлтым и оранжевым; ортологов - зелёным. Малиновая линия справа отвечает дупликации гена в организме ERWT9.
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева